引言
膝关节是人体主要的运动关节,膝关节疾病严重影响人们的生活与健康.磁共振成像(MRI)对软组织的分辨力高,且可以使人体免受电离辐射的伤害,已成为临床诊断膝关节疾病的常用方法.近年来,高场MRI已经得到越来越多的应用,局部射频功率沉积(SAR)是高场磁共振扫描时需要考虑的关键因素,因为高场强有着使人体组织局部SAR超标[1]的风险,会给人体带来伤害.不同于全局SAR,对于不同的个体,局部SAR的准确估计需要由对特异的人体模型进行电磁仿真[2-4]来得到.当前,低场磁共振扫描依然得到广泛应用,某些情况下在低场扫描后需进行高场扫描以获得更清晰的细节.因此,通过分割低场磁共振图像构建膝关节模型,进行电磁仿真以在高场扫描之前得到局部SAR的估计值很有必要.
对于图像分割,专业人员手动标注的时间成本高,且分割效果会受人为因素影响;半自动分割方法,如区域生长[5]和snake算法[6],虽然可以提高分割效率,但是分割结果受初始信息的影响较大;传统的全自动分割方法,如统计形状模型[7]、基于图谱的分割[8]和支持向量机[9]等,达到了一定的效果,但由于使用预定义的模板或者模型,其对复杂图像分割的准确性和鲁棒性仍需提升.近年来,深度卷积神经网络已经广泛应用于图像分割[10, 11],相对于传统的半自动与全自动分割方法,它可以自动提取图像的浅层和深层特征,适合复杂的图像分割任务,已有使用深层卷积网络对膝关节图像进行分割的方法[12-14].U-Net[15]是一种当前使用的先进的全卷积网络,采用对称的U型结构,通过在上采样过程中不断融合浅层的图像特征,可以得到更精确的分割结果,并可以进行多尺度预测.因此,自从2015年被提出后,U-Net被广泛应用于医学图像的分割[16-18],包括膝关节图像的分割[19-21].
之前,我们研究组采用单个U-Net基于“肌-脂-骨”简化模型进行了膝关节模型重建[22].显然,如果进行更加精细的组织分类将有助于提高局部SAR估计的准确度.本文旨在更为准确的分割低场膝关节磁共振图像,得到膝关节模型以进行局部SAR的估计.本文将膝关节组织分为肌肉、脂肪、松质骨、皮质骨、半月板以及软骨;我们将其他不清晰的组织(例如肌腱、韧带、血管等)都归为肌肉,因为它们与肌肉具有接近的介电参数,因而这种合并对仿真结果影响较小.与高场磁共振图像相比,低场磁共振图像的信噪比通常较低、组织对比度不足,这为图像分割带来了较大困难.而且网络分割效果往往在肌肉、脂肪等占比较大的组织中表现较好,因此膝关节中形态细小的软骨组织与半月板的分割精度仍有很大提升空间.为了提高小组织的分割效果,本文使用焦点损失函数(Focal Loss Function)代替交叉熵损失函数,以提高网络学习中占比相对较小的组织的权重.并进一步提出一种包括两个U-Net的级联网络结构:前一个网络用于在整幅图上分割肌肉、脂肪等占比较大的组织,并根据其分割结果预测软骨与半月板等小组织的大致位置;后一个网络基于该信息在一个更小的子图上分割小组织,以提高小组织的分割精度.两个网络的分割结果合并在一起以构成膝关节模型.我们采用五种定量指标评估分割效果,并且对所重建模型进行了电磁仿真与局部SAR的计算.
1 理论方法
1.1 本文提出的网络结构
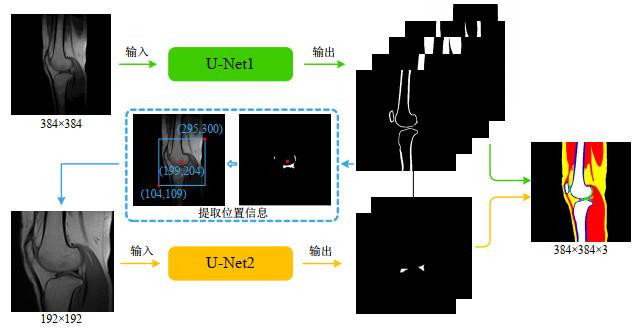
本文提出的级联网络结构如图 1所示,主要由两个U-Net构成:第一个U-Net用来分割肌肉、脂肪、皮质骨与松质骨;第二个U-Net用来分割软骨与半月板.在第一个U-Net的分割任务中,以原始图像作为U-Net网络的输入数据,在训练中以手工标注结果(包含肌肉、脂肪、皮质骨与松质骨四种组织)作为真实值带入损失函数,根据梯度下降原理不断调整参数以降低损失函数值,直到趋于稳定.这样,当输入测试图像时,网络输出为各种组织的概率矩阵,从而实现像素级分类.位于膝关节内部的软骨与半月板都被归为背景类,可以通过提取分割结果中连通组织内部的具有一定大小的空洞来实现定位.提取出的空洞可以看作对软骨与半月板联合形态的粗分割,为了得到更连通的形态,本文对空洞部分做了腐蚀与膨胀滤波.以空洞部分的质心为中心,提取出只包含软骨与半月板的原始图像切片(大小减少一半,为192×192),作为第二个网络的输入数据.这种级联的网络结构利用第一个网络提取到的软骨与半月板的大致位置信息去辅助第二个网络分割,使网络在更小的区域中分割小组织;此外,网络分割目标的减少也可以使网络专注学习这些更小的类别,进一步提升小组织的分割精度,以达到精确分割软骨与半月板的目的.级联网络结构中的两个U-Net都使用焦点损失函数,因为焦点损失函数可增大损失函数中小组织分割目标的损失比重,降低背景等易分类像素点的损失比重,可以使网络更偏向对小组织的学习.最后,我们采用小组织优先策略整合两次的分割结果,针对整合图像中组织间的空洞,提取空洞边缘像素,将之归类为拥有最大像素数的类别.此外,将分割结果对应的原图中所有像素灰度值都小于10的连通域自动归为背景,以消除背景中的噪声.
图1
图1
本文提出的级联网络的整体框图,设原始图像的左下角坐标为(0, 0),(199, 204)为提取的软骨与半月板的中心坐标,(104, 109),(295, 300)分别为裁剪区域在原图像中左下角与右上角的坐标.最后的分割结果由对两个网络的分割结果根据提取的位置信息进行融合及后处理得到
Fig.1
The overall block diagram of the proposed cascaded network. Let the coordinate of the lower left corner of the original image be (0, 0), (199, 204) is the center coordinate of the extracted cartilage and meniscus, (104, 109) and (295, 300) are the coordinates of the lower left and upper right corners of the crop area in the original image, respectively. The final result is obtained by merging the segmentation results of the two networks according to the extracted position information, and post-processing
1.2 U-Net
U-Net由编码器与解码器构成,编码器与解码器呈对称的U型结构(以提出的级联网络中第一个U-Net为例,如图 2所示).在编码阶段,U-Net通过4个卷积模块(Conv,Dropout,Relu)与4次最大池化(Max Pool)下采样提取图像特征信息,每个卷积模块由2个卷积层构成.在解码阶段,U-Net通过4次反卷积(Up-conv)与4个卷积模块恢复图像大小,并采用跳跃连接,拼接对应的编码器产生的浅层特征信息,通过卷积模块降低特征通道,融合特征,实现逐像素点的分类.U-Net在解码阶段拥有大量的特征通道,并融合了低层的空间定位信息,可以实现更精确的分割.为了保证输入输出图像的大小一致,我们在每个卷积层使用边缘0填充的卷积方式.
图2
图2
本文所提级联网络中第一个U-Net的结构
Fig.2
The structure of the first U-Net in the proposed cascade network
1.3 焦点损失函数
焦点损失函数的提出是为了解决目标检测中前景与背景不均衡的问题[23],是交叉熵函数的变体.在本文的分割任务中,半月板与软骨组织的形态细小,通常只占整张图像中很小的比例,小组织类与肌肉、脂肪等组织以及背景类的比例极不均衡,这也导致了小组织分割效果的不理想.在第一个网络的分割任务中,不易分类类别为肌肉、脂肪、皮质骨与松质骨类,易分类类别为背景类;在第二个网络的分割任务中,不易分类类别为软骨与半月板类, 易分类类别为除软骨与半月板之外的背景类. 对于一个属于易分类类别的体素来说,在网络训练时,它在所属类别上的预测概率通常较高,这部分像素点对应的损失函数值通常较小,并且它们在整张图像中所占面积很大,这会导致整体的损失函数值偏小,从而使网络无法向提升小组织分割精度的方向优化.针对这个问题,本文使用焦点损失函数代替交叉熵损失函数,公式如下:
其中
2 实验部分
2.1 数据集
本文使用的数据集共包含67位志愿者的膝关节磁共振图像,将其中48位划分为训练集,其余19位划分为测试集,无验证集.志愿者的平均年龄约为46.3岁.在实验之前,每一位志愿者均签订了知情同意书.膝关节磁共振图像于2019年在张家口仁爱医院影像科的0.35 T MRI系统JC35P(嘉恒医疗装备有限公司)上采集,均为矢位T1加权自旋回波图像,扫描参数如下:层厚为4 mm;视野为230 mm×230 mm;图像尺寸为384×384;图像切片数为30;体素尺寸约为0.6 mm×0.6 mm×4 mm,该体素分辨率满足场强为3 T的SAR仿真要求[24-26].数据的人工标注由两名相关的专业人员在Photoshop软件上完成.
2.2 网络训练
所有网络训练的预测都在TensorFlow软件平台上进行,使用Python编程语言,训练的硬件环境为4核Intel i7-4770处理器和16 GB内存的GeForce GTX TITAN GPU显卡.经过不断优化调整,第一个分割网络为5层的U-Net网络,卷积核大小为5×5;第二个分割网络为5层的U-Net网络,卷积核大小为7×7.两个网络的初始特征通道数都设置为16,并均使用Adam优化器,初始学习率设置为0.001.网络参数的初始值通过均值为0、标准差为
2.3 分割结果的评价指标
以人工标注结果作为基准,使用Dice系数(DCC)、Jaccard系数(JAC)、真阳性率(TPR)、假阳性率(FPR)来评价网络分割的性能,其中DCC与JAC用于衡量网络分割结果与人工标注结果之间的重叠度与相似度,DCC与JAC的数值越大,说明网络的分割结果越接近手动标注结果.TPR为被网络正确预测为分割目标的像素点数量与真实分割目标像素点的比例,TPR越大,说明网络的分割效果越好;而FPR为被网络错误预测为分割目标的像素点数量与真实背景像素数的比例,FPR越小,说明网络的分割效果越好,这四项指标计算公式如下:
T和U分别为分割目标的人工标注结果和待评估网络的分割结果,
此外,我们还使用平均表面距离(ASD)评估网络在各个组织边缘的分割效果,计算公式如下:
其中,
2.4 SAR估计
使用所提方法对测试集进行分割,对得到的分割结果进行三维重建,可得到每一位志愿者的膝关节模型.使用3 T发射线圈模型进行用于局部SAR估计的电磁仿真:仿真软件为REMCOM xFDTD(Remcom Inc.,State College,PA);3 T发射线圈模型为一个正交的膝关节鸟笼线圈[27],采用集总电容模式,有8个杆,长度为180 mm,环的半径为87.5 mm.构建的膝关节模型放置于该线圈中心,线圈的两个端口分别注入两个幅度为1 V、相位差为90°的正弦信号,线圈内部的电场
SAR被定义为单位质量组织吸收的射频功率,计算公式如下:
其中,
对于一个体素p,其局部SAR(SAR10g)定义如下:
其中,V是一个以p为中心的立方区域,质量约为10g,q为V中的一个体素;mq是体素q的质量.
3 结果与讨论
3.1 图像分割效果的对比
图 3以一位志愿者(女性,22岁)的膝关节磁共振图像为例,展示了利用本文所提方法进行图像分割的过程.图中分别展示了两个U-Net网络的分割结果,以及利用第一个U-Net网络分割背景中的软骨与半月板的位置信息对输入图像进行裁剪的示意图.
图3
图3
第一行分别为初始图像与对应的皮质骨、松质骨、肌肉、脂肪与背景的分割结果,本文所提方法通过提取背景中的空洞并计算其质心得到软骨与半月板的位置信息,以质心为中心进行裁剪,得到包含软骨与半月板的子图,即第二个网络的输入图像.第二行分别展示了裁剪示意图与利用第二个网络得到的软骨与半月板的分割结果
Fig.3
The first row is the initial image and the corresponding segmentation results of cortical bone, cancellous bone, muscle, fat, and background. Extract the cavity in the background and calculate its centroid to obtain the position information of cartilage and meniscus. Cut out with the centroid as the center to obtain a sub-image containing cartilage and meniscus, which is the input image of the second network. The second row respectively shows the cut-out schematic diagram and the segmentation results of cartilage and meniscus with the second network
由于所提网络是基于U-Net的级联结构,为了评估其分割性能,因此本文将所提网络的分割结果与单U-Net网络及其几种改进形式进行了对比,包括使用普通交叉熵损失函数的单U-Net网络、使用焦点损失函数的单U-Net网络[记作U-Net (focal loss)]、使用Attention机制的单U-Net网络[28] [记作U-Net (attention)]、U-Net++[29].其中,单U-Net均采用5层、7×7大小卷积核的网络结构.U-Net (attention)通过在U-Net编码器路径和解码器路径之间添加Attention门来产生归一化的权重掩膜,以增强感兴趣区域,提高分割效果.U-Net++使用一系列嵌套的密集跳跃连接路径,层数为5,卷积核大小为5×5,实验中选取U-Net++的对比数据为它最外层的分割结果.以人工标注结果为标准,计算了使用上述各方法得到的分割结果的评价指标(表 1).
表1 对本文所提方法与其它4种对比方法分割结果的评价指标的分析
Table 1
| 评价指标 | 分割方法 | 统计参数 | 半月板 | 软骨 | 皮质骨 | 松质骨 | 脂肪 | 肌肉 |
| FPR | U-Net | Mean | 0.0003 | 0.0007 | 0.0054 | 0.0024 | 0.0091 | 0.0135 |
| Std | 0.0001 | 0.0002 | 0.0013 | 0.0007 | 0.0024 | 0.0033 | ||
| U-Net (focal loss) | Mean | 0.0002 | 0.0011 | 0.0093 | 0.0024 | 0.0074 | 0.0133 | |
| Std | 0.0001 | 0.0004 | 0.0019 | 0.0007 | 0.0019 | 0.0033 | ||
| U-Net (attention) | Mean | 0.0003 | 0.0010 | 0.0054 | 0.0027 | 0.0105 | 0.0122 | |
| Std | 0.0001 | 0.0003 | 0.0010 | 0.0007 | 0.0026 | 0.0035 | ||
| U-Net++ | Mean | 0.0004 | 0.0005 | 0.0054 | 0.0022 | 0.0089 | 0.0169 | |
| Std | 0.0001 | 0.0002 | 0.0010 | 0.0008 | 0.0022 | 0.0038 | ||
| 本文所提方法 | Mean | 0.0002 | 0.0013 | 0.0050 | 0.0023 | 0.0064 | 0.0132 | |
| Std | 0.0001 | 0.0004 | 0.0010 | 0.0008 | 0.0015 | 0.0035 | ||
| TPR | U-Net | Mean | 0.6347 | 0.5435 | 0.7550 | 0.9333 | 0.8799 | 0.9372 |
| Std | 0.1030 | 0.0912 | 0.0583 | 0.0297 | 0.0498 | 0.0264 | ||
| U-Net (focal loss) | Mean | 0.7291 | 0.6025 | 0.8053 | 0.9251 | 0.8547 | 0.9333 | |
| Std | 0.0824 | 0.1005 | 0.0466 | 0.0284 | 0.0638 | 0.0242 | ||
| U-Net (attention) | Mean | 0.7076 | 0.5773 | 0.7298 | 0.9375 | 0.8792 | 0.9231 | |
| Std | 0.0617 | 0.0794 | 0.0437 | 0.0336 | 0.0604 | 0.0244 | ||
| U-Net++ | Mean | 0.6883 | 0.5144 | 0.6800 | 0.9396 | 0.8459 | 0.9349 | |
| Std | 0.0926 | 0.0959 | 0.0494 | 0.0291 | 0.0600 | 0.0185 | ||
| 本文所提方法 | Mean | 0.7562 | 0.7291 | 0.7635 | 0.9410 | 0.8803 | 0.9422 | |
| Std | 0.0671 | 0.0793 | 0.0479 | 0.0250 | 0.0630 | 0.0231 | ||
| DCC | U-Net | Mean | 0.6755 | 0.5502 | 0.6980 | 0.9292 | 0.8868 | 0.9252 |
| Std | 0.0718 | 0.0499 | 0.0378 | 0.0272 | 0.0387 | 0.0224 | ||
| U-Net (focal loss) | Mean | 0.6884 | 0.5505 | 0.6608 | 0.9310 | 0.8860 | 0.9234 | |
| Std | 0.0520 | 0.0562 | 0.0396 | 0.0244 | 0.0491 | 0.0207 | ||
| U-Net (attention) | Mean | 0.6837 | 0.5551 | 0.6866 | 0.9289 | 0.8868 | 0.9232 | |
| Std | 0.0416 | 0.0416 | 0.0347 | 0.0273 | 0.0511 | 0.0235 | ||
| U-Net++ | Mean | 0.6389 | 0.5618 | 0.6502 | 0.9296 | 0.8730 | 0.9118 | |
| Std | 0.0568 | 0.0610 | 0.0333 | 0.0310 | 0.0529 | 0.0230 | ||
| 本文所提方法 | Mean | 0.7071 | 0.6023 | 0.7083 | 0.9351 | 0.8907 | 0.9312 | |
| Std | 0.0521 | 0.0396 | 0.0293 | 0.0234 | 0.0419 | 0.0176 | ||
| JAC | U-Net | Mean | 0.5394 | 0.4092 | 0.5391 | 0.8824 | 0.8102 | 0.8677 |
| Std | 0.0699 | 0.0491 | 0.0405 | 0.0319 | 0.0596 | 0.0289 | ||
| U-Net (focal loss) | Mean | 0.5484 | 0.4107 | 0.5069 | 0.8844 | 0.8055 | 0.8632 | |
| Std | 0.0528 | 0.0462 | 0.0421 | 0.0362 | 0.0698 | 0.0280 | ||
| U-Net (attention) | Mean | 0.5418 | 0.4077 | 0.5341 | 0.8772 | 0.8073 | 0.8653 | |
| Std | 0.0414 | 0.0379 | 0.0367 | 0.0337 | 0.0691 | 0.3033 | ||
| U-Net++ | Mean | 0.4931 | 0.3296 | 0.4954 | 0.8716 | 0.7851 | 0.8468 | |
| Std | 0.0511 | 0.0471 | 0.0415 | 0.0364 | 0.0699 | 0.0314 | ||
| 本文所提方法 | Mean | 0.5610 | 0.4572 | 0.5600 | 0.8896 | 0.8139 | 0.8761 | |
| Std | 0.0525 | 0.0373 | 0.0331 | 0.0300 | 0.0600 | 0.0251 | ||
| ASD/mm | U-Net | Mean | 1.3462 | 2.8589 | 1.2567 | 1.1638 | 1.3399 | 2.2038 |
| Std | 0.7835 | 0.8687 | 0.5117 | 0.4399 | 0.1666 | 0.6693 | ||
| U-Net (focal loss) | Mean | 1.3125 | 3.1638 | 1.2982 | 1.1325 | 1.3147 | 2.2202 | |
| Std | 0.6548 | 1.1781 | 0.2242 | 0.3914 | 0.3295 | 0.7109 | ||
| U-Net (attention) | Mean | 1.2539 | 2.7799 | 1.2440 | 1.5075 | 1.3017 | 2.4292 | |
| Std | 0.6181 | 0.8609 | 0.4551 | 0.3993 | 0.2916 | 1.3714 | ||
| U-Net++ | Mean | 1.6536 | 3.6479 | 1.6531 | 1.7551 | 1.4824 | 2.9900 | |
| Std | 0.6723 | 1.5050 | 1.5734 | 1.0557 | 0.2625 | 1.3447 | ||
| 本文所提方法 | Mean | 1.2470 | 2.6555 | 1.0716 | 1.0651 | 1.2412 | 1.9097 | |
| Std | 0.5749 | 0.8258 | 0.3542 | 0.3869 | 0.2607 | 0.5289 |
Mean表示测试集的评价指标的平均值;Std表示测试集的评价指标的标准差
从5个评价指标来看,相比其他4种方法,使用本文所提方法在软骨与半月板上的分割效果有了一定的提升,可见提取原始图像切片并使用焦点损失函数可以改进小组织的分割效果.而且本文所提方法在松质骨、脂肪与肌肉上的分割效果在5种方法中也达到最优.相比于之前采用“肌-脂-骨”简化方法(肌肉、脂肪与骨头的Dice系数分别为0.950±0.038、0.929±0.046、0.928±0.049)[22],肌肉、脂肪与骨头的Dice系数均有所降低,这主要有两点原因:一是要分类的组织更多,这意味网络学习更加复杂;二是使用了不同的数据集,数据集的大小、训练集与测试集均不同.
图4
图4
使用5种自动分割方法得到的最后分割结果与人工标注结果的对比.从上到下,为一位志愿者图像集中4张连续的等间隔切片,其中,脂肪、肌肉、松质骨、皮质骨、软骨、半月板分别标记为黄色、红色、白色、蓝色、绿色与粉色
Fig.4
The comparison between the final segmentation results obtained by using the 5 automatic segmentation methods and the manual segmentation. From top to bottom, 4 consecutive equally spaced slices are collected for a volunteer image. Among them, fat, muscle, cancellous bone, cortical bone, cartilage, and meniscus are marked as yellow, red, white, blue, green, and pink, respectively
3.2 SAR估计的对比
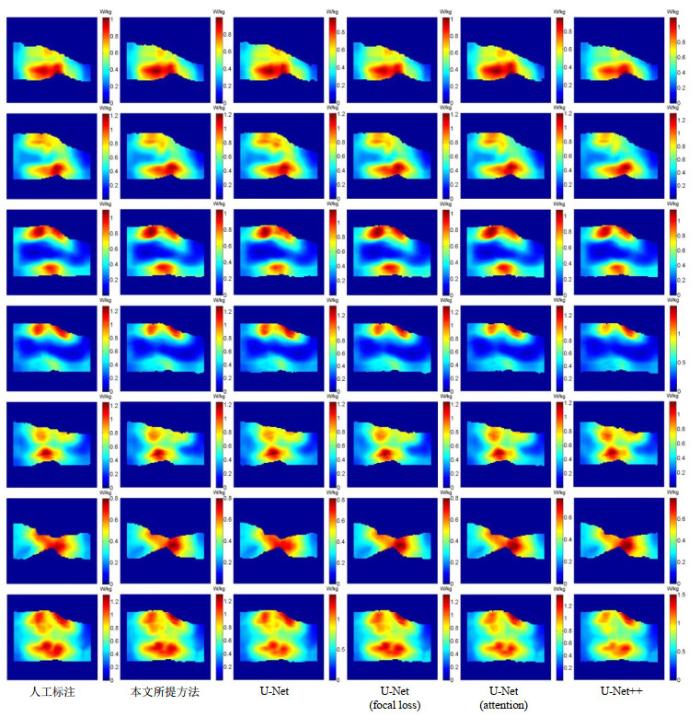
我们将本文所提方法与其他4种对比方法应用于测试集,重建出膝关节模型,进行电磁仿真,并根据仿真结果计算SAR10g的最大值.分别求出5种自动分割模型与人工标注模型得到的SAR10g最大值的相对误差,并计算相对误差的平均值与标准差.如表 2所示,相比其他方法,使用本文方法构建的模型与人工标注模型之间SAR10g的相对误差更小,两者更加接近;而且相比于与先前的“肌-脂-骨”简化模型(简化模型与手动分割模型间SAR10g的相对误差为0.038 7±0.025 7),本文所提方法与人工标注模型之间的相对误差的均值更小,但标准差更大,这主要还是由数据集的不同造成的,本研究使用了不同的、更大的训练集与测试集.图 5展示了一位志愿者的SAR10g分布图,从图中可以看出,相比另外4种方法,使用本文所提的级联网络构建的膝关节模型拥有与人工标注模型更加相似的SAR10g分布,热点区域的位置基本相同,这验证了本文所提的级联网络的有效性.
表2 5种自动分割模型和人工标注结果间最大SAR10g的相对误差的比较
Table 2
| 统计参数 | U-Net | U-Net (focal loss) | U-Net (attention) | U-Net++ | 本文所提方法 |
| Mean | 0.0538 | 0.0433 | 0.4042 | 0.0547 | 0.0366 |
| Std | 0.0599 | 0.0424 | 0.3621 | 0.0520 | 0.0357 |
图5
图5
一位志愿者的膝关节SAR10g分布图.从左到右,分别基于人工标注结果,使用本文所提方法、采用交叉熵损失的U-Net,采用焦点损失的U-Net、采用attention机制的U-Net以及U-Net++得到的模型,6种方法的SAR10g值分别为1.309 8 W/kg、1.304 5 W/kg、1.402 4 W/kg、1.316 2 W/kg、1.383 5 W/kg、1.506 0 W/kg.第1行到第6行为6个均匀分布的切片的SAR10g分布图,最后1行为SAR10g的最大密度投影
Fig.5
SAR10g distribution maps of a volunteer's knee joint. From left to right, they are based on the manual segmentation, and the models obtained by the proposed method, the U-Net using cross-entropy loss, the U-Net using focus loss, the U-Net using attention mechanism and U-Net++, respectively. The SAR10g values of the six models are 1.309 8 W/kg, 1.304 5 W/kg, 1.402 4 W/kg, 1.316 2 W/kg, 1.383 5 W/kg, 1.506 0 W/kg. The first row to the sixth row are the SAR10g distribution maps of 6 uniformly distributed slices, and the last row is the maximum density projection of the SAR10g
3.3 讨论
深度卷积神经网络在自动分割膝关节图像上的应用多数集中在提取骨骼、半月板与软骨,用于损伤鉴定与疾病诊断.局部SAR是高场MRI扫描时需要考虑的重要因素,引起了越来越多的重视.为构建用于进行SAR估计的电磁仿真的模型,我们需要分割膝关节的所有主要组织.而膝关节结构的复杂性给网络的分割性能带来了挑战.在之前的工作中,我们采用“肌肉-骨骼-脂肪”的简化策略分割低场膝关节磁共振图像,而本文分割的组织包括肌肉、皮质骨、松质骨、半月板、软骨与脂肪,组织分类的增加会使网络学习任务更加复杂,低场磁共振图像的信噪比与组织间对比度相对较低,将灰度差异并不大的皮质骨、软骨与半月板从肌肉中自动区分出来,也给网络分割增加了难度.
先前已有使用级联卷积神经网络进行医学图像诊断的相关研究[30],而本文所提方法中包含的两个U-Net网络都被应用于图像分割,分别负责大组织(4种)与小组织(2种)的分割,第一个U-Net的分割结果中包含小组织的位置信息,并且在两个网络上都使用焦点损失函数.在对几种分割方法得到的分割结果做定量分析后,我们发现级联网络方法拥有更好的分割性能.得到膝关节模型后,对其做电磁仿真,得到的SAR10g对比进一步说明级联网络可以得到更接近人工标注的分割结果.我们认为,级联网络能够取得更好的分割性能主要有三个原因:一是焦点损失函数使网络对小组织的关注度提升;二是在得到半月板与软骨组织的定位信息后,图像中的分割目标更明确,避免了多余背景的学习;三是网络分割目标类别的减少,使得每个网络的分割任务减轻,降低了网络拟合分布的复杂性.
虽然以上研究展示了令人满意的初步成果,但是仍存在一些不足.首先是数据集方面的不足,更充足的数据集可以得到更可靠的研究结果,尽管相对上一次的工作,我们对数据集与训练集分别做了扩充,但是我们的数据集还应包括各个年龄段和膝关节疾病严重程度不同的人群的数据.其次,一些组织(例如皮质骨)的分割性能仍需提升,我们采取的级联网络与焦点损失函数对这类组织效果不够理想,如何改善这类组织的分割性能,是我们接下来研究的方向.
4 结论
基于精确构建用于SAR估计的膝关节模型的目的,本文提出了一种包含两个U-Net的级联网络架构,并使用焦点损失函数,实现膝关节低场磁共振图像的自动分割.实验结果表明,基于本文所提方法构建的模型拥有与人工标注结果相近的SAR10g分布,有助于膝关节高场磁共振扫描的SAR评估.
无
参考文献
Parallel transmission for ultrahigh-field imaging
[J].DOI:10.1002/nbm.3313 [本文引用: 1]
A specific absorption rate prediction concept for parallel transmission MR
[J].DOI:10.1002/mrm.24138 [本文引用: 1]
Comprehensive RF safety concept for parallel transmission MR
[J].
7 Tesla body MRI: B1 shimming with simultaneous SAR reduction
[J].DOI:10.1088/0031-9155/52/17/022 [本文引用: 1]
Automatic segmentation of cartilage in high-field magnetic resonance images of the knee joint with an improved voxel-classification-driven region-growing algorithm using vicinity-correlated subsampling
[J].
Surface extraction and thickness measurement of the articular cartilage from MR images using directional gradient vector flow snakes
[J].DOI:10.1109/TBME.2006.872816 [本文引用: 1]
Anatomically corresponded regional analysis of cartilage in asymptomatic and osteoarthritic knees by statistical shape modelling of the bone
[J].DOI:10.1109/TMI.2010.2047653 [本文引用: 1]
Automatic atlas-based three-label cartilage segmentation from MR knee images
[J].DOI:10.1016/j.media.2014.05.008 [本文引用: 1]
Automatic knee cartilage segmentation from multi-contrast MR images using support vector machine classification with spatial dependencies
[J].DOI:10.1016/j.mri.2013.06.005 [本文引用: 1]
DeepLab: semantic image segmentation with deep convolutional nets, atrous convolution, and fully connected CRFs
[J].DOI:10.1109/TPAMI.2017.2699184 [本文引用: 1]
Automated segmentation of knee bone and cartilage combining statistical shape knowledge and convolutional neural networks: Data from the osteoarthritis initiative
[J].DOI:10.1016/j.media.2018.11.009 [本文引用: 1]
Deep convolutional neural network and 3D deformable approach for tissue segmentation in musculoskeletal magnetic resonance imaging
[J].
Deep convolutional neural network for segmentation of knee joint anatomy
[J].DOI:10.1002/mrm.27229 [本文引用: 1]
NAS-Unet neural architecture search for medical image segmentation
[J].DOI:10.1109/ACCESS.2019.2908991 [本文引用: 1]
Use of 2D U-Net convolutional neural networks for automated cartilage and meniscus segmentation of knee MR imaging data to determine relaxometry and morphometry
[J].
Automated segmentation of knee bone and cartilage combining statistical shape knowledge and convolutional neural networks
[J].DOI:10.1016/j.media.2018.11.009
Knee menisci segmentation and relaxometry of 3D ultrashort echo time cones MR imaging using attention U-Net with transfer learning
[J].DOI:10.1002/mrm.27969 [本文引用: 1]
A U-Net network-based rapid construction of knee models for specific absorption rate estimation
[J].
用于SAR估计的基于U-Net网络的快速膝关节模型重建
[J].
Focal loss for dense object detection
[J].DOI:10.1109/TPAMI.2018.2858826 [本文引用: 1]
Toward individualized SAR models and in vivo validation
[J].DOI:10.1002/mrm.22948 [本文引用: 1]
SAR simulations for high-field MRI: how much detail, effort, and accuracy is needed?
[J].
Spatial resolution of numerical models of manand calculated specific absorption rate using the FDTD method: a study at 64 MHz in a magnetic resonance imaging coil
[J].DOI:10.1002/jmri.10359 [本文引用: 1]
Application of a birdcage coil at 3 Tesla to imaging of the human knee using MRI
[J].DOI:10.1002/(SICI)1522-2594(199908)42:2<215::AID-MRM1>3.0.CO;2-8 [本文引用: 1]
Knee menisci segmentation and relaxometry of 3D ultrashort echo time cones MR imaging using attention U‐Net with transfer learning
[J].