引言
脑胶质瘤是一种恶性概率较高的脑肿瘤.它是由大脑和脊髓胶质细胞癌变所产生的原发性颅脑肿瘤.根据肿瘤细胞在病理学上的恶性程度,可将脑胶质瘤分为低级别脑胶质瘤(Low-Grade Glioma,LGG)和高级别脑胶质瘤(High-Grade Glioma,HGG).HGG的致死率非常高,患者的平均生存期仅为12个月左右,随着脑肿瘤细胞的剧烈繁殖,LGG可以转化为HGG,因此,尽早地发现和治疗脑胶质瘤是提高患者存活率的关键.
磁共振成像(Magnetic Resonance Imaging,MRI)能够有效地提供有关脑肿瘤的形状、大小、位置、性状等信息,是最常见的检测和监测脑肿瘤的方法.通过多模态磁共振影像对脑胶质瘤的子结构区域(全肿瘤(Whole Tumor,WT)、肿瘤核心(Tumor Core,TC)和增强肿瘤(Enhance Tumor,ET))进行先分割再定量分析可辅助确定脑胶质瘤的恶化程度或侵袭程度,制定手术计划和治疗方案.在传统的临床诊断中,医生逐切片地手工分割肿瘤区域,非常耗时且易产生标注疏漏和不同医生间的标注差异.因此,需要一种面向MRI的自动脑肿瘤分割方法辅助临床,这不仅节省医生的时间,同时也减少了人为标注的失误和差异,可有效提升诊断的一致性、准确性和高效性.
近年来,随着卷积神经网络(Convolutional Neural Network,CNN)的快速发展,深度学习的方法被广泛应用于脑肿瘤分割.现有的研究工作主要是以经典Unet[1]为框架,提出各种改进的Unet模型实现脑肿瘤分割任务.例如,2021年,Rehman等[2]基于Unet设计了特征增强模块,从底层特征中提取中层特征,并与密集层共享,解决了脑胶质瘤分割中病灶区域和健康区域占比不平衡的问题.2022年,Zhao等[3]开发了一种用于脑胶质瘤分割的多模态特征融合Unet网络,该网络从对应的成像模态中独立提取低级特征,并通过混合注意力块对特征进行强化.Sheng等[4]提出一种SoResU-Net模型,该模型在Unet的跨层连接处采用一种二阶模块,将特征图相乘使特征通道扩展至二阶维度.Han等[5]通过在Unet框架中引用三重注意力模块,建立了融合图像全局与局部特征的脑肿瘤分割模型.此外,还有一些学者基于具有更复杂连接的Unet衍生框架进行脑肿瘤分割.例如,2018年,Zhou等[6]采用Unet++进行脑肿瘤分割;2020年,Huang等[7]提出了具有多尺度深度监督的Unet3+网络,将低级语义与高级语义结合起来用于脑肿瘤分割;2022年,Qin等[8]用残差模块替换Unet3+网络编码阶段的普通卷积,以此避免Unet3+网络在训练过程中的梯度消失,并将改进模型用于脑肿瘤分割.
但上述方法所提出的模型缺乏对远程特征相关性的建模能力.近两年,Transformer被用来解决这一问题,它最初被用于机器翻译领域[9],其在自然语言处理方面的巨大成功引起了计算机视觉研究人员的关注[10]. 2020年,Dosovitskiy等[11]提出了视觉转换器(Vision Transformer,VIT),它在ILSVRC-2012数据集上通过预训练实现了图像分类且性能出色,但是,与传统CNN相比,它的数据处理量过大、实验时间过长.因此,后续的研究中,研究人员不断完善VIT,并以此提出新的网络结构,如2021年提出的DeiT模型[12]、Swin模型[13]和Le-VIT模型[14].一些研究尝试将Transformer结构应用于医学脑胶质瘤图像分割.如2021年,Chen等[15]提出了TransU-Net,利用CNN提取底层特征,然后通过Transformer对全局特征进行建模,解决了Unet在建模长距离依赖关系表达方面的局限性问题.2021年,Wang等[16]提出了TransBTS,将基于CNN的编码器和解码器与VIT结合起来,以提高低分辨率阶段的分割性能.Huang等[17]提出了一种结合Transformer与对抗网络的模型,用于多模态MRI脑肿瘤分割.
上述工作为脑肿瘤分割技术的研究提供了许多借鉴,但由于脑胶质瘤存在形状多异,位置大小分布不均匀以及边界模糊等特点,导致现有网络分割仍存在精度不高,脑胶质瘤病灶边缘区域分割不精确的问题.
为提升脑胶质瘤的分割精度,本文提出了一种融合多重自注意力机制、残差网络和可变形卷积(Deformable Convolutional Module,DCM)的Unet改进网络.首先在网络的瓶颈层引入基于Transformer的多重自注意力模块(Multi-Attention Transformer Module,MATM),通过自注意力机制计算局部以及全局的特征信息,并通过共享的记忆单元计算样本之间相关性,使网络更好地学习图像特征.其次,在跳跃连接处采用DCM,通过对卷积核增加额外的偏移量以得到最优的卷积效果,进而提升网络对病灶边缘区域的敏感度.最后,将经典Unet框架中普通卷积替换为残差模块以优化训练过程中梯度的反向传播.
1 本文算法
1.1 总体模型
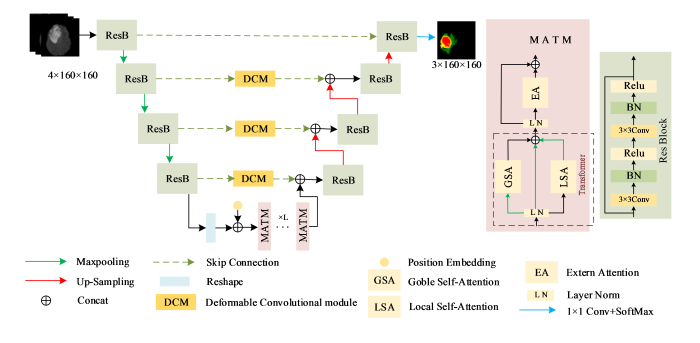
本文提出的模型结构如图1所示.该模型基于典型的编码器-解码器结构,具有两条对称的路径.编码器包括4个残差模块(Res Block,ResB)和3个下采样操作,下采样操作为最大池化(Maxpooling).每个残差模块包含两个3×3的卷积层(Conv),每个卷积层后面连接着一个标准化层(Batch Normalize,BN)和一个激活函数层(Rectified Linear Unit,Relu).残差模块能够有效减少网络训练过程中梯度消失的影响,在一定程度上促进了信息的传播.编码过后,图像特征被重构(Reshape),并加入位置编码(Position Embedding),以捕捉病变区域的绝对位置和相对位置,之后传入瓶颈层的MATM生成含有局部以及全局依赖关系的特征图,再将特征图传入外部注意力模块生成含有样本间相关性的特征图.为了提升病灶边缘的分割精度,在跳跃连接(Skip Connection)上增加了DCM,它能够从不规则形状的病变区域中捕捉形状敏感特征.语义信息在经过解码路径的4个残差模块和3次上采样(Up-Sampling)后,通过1个1×1的卷积(1×1 Conv)和SoftMax激活函数,输出分割结果预测图.
图1
1.2 多重自注意力模块
传统的Unet网络在进行特征提取时只关心局部感受野内卷积核内信息的过滤,没有充分考虑图像整体像素间的自相关性以及整个样本集中样本间的潜在相关性.本文设计了一种融合Transformer和外部注意力的MATM,并将其用于网络的瓶颈层.MATM的整体结构如图1中的MATM子图所示.整个MATM由局部自注意力(Local Self-Attention,LSA)、全局自注意力(Global Self-Attention,GSA)以及外部注意力(External Attention,EA)三部分构成.其中,LSA和GSA是基于对Transformer的一种改进,目的是通过Transformer的自注意力机制从不同尺度对特征图的依赖关系进行建模,进而捕捉图像内部像素之间的自相关性;而EA的引入为了学习样本间的外部相关性;三个模块的综合使用及交互融合形成了一种多重自注意力机制,可增强网络提取特征的能力.
如图1的MATM子图所示,在MATM模块内,带有位置编码的语义信息经过归一化(Layer Norm,LN)之后分别输入到三条支路进行处理:一条经过最大池化(MaxPooling)后输入到GSA模块;另一条下采样残差支路,即输入经过MaxPooling后直接输出;还有一条输入给LSA模块,并对输出结果进行MaxPooling,以便与GSA的输出保持尺寸一致.下采样残差支路的目的是防止自注意力计算过程中信息丢失,而在输入给GSA之前进行MaxPooling是为了减少全局自注意力的计算量.三条支路的语义信息随后通过拼接(Concat)整合到一起,最后形成带有自注意力关注的新特征信息.然后,再进一步输入给EA模块进行处理,以获取样本之间的相互依赖性.
1.2.1 基于Transformer的自注意力模块
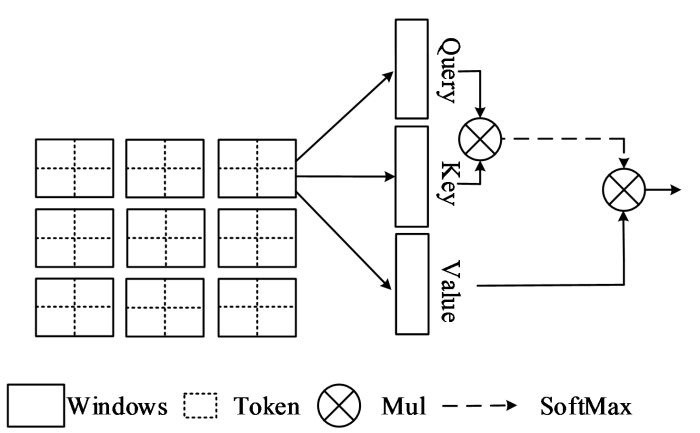
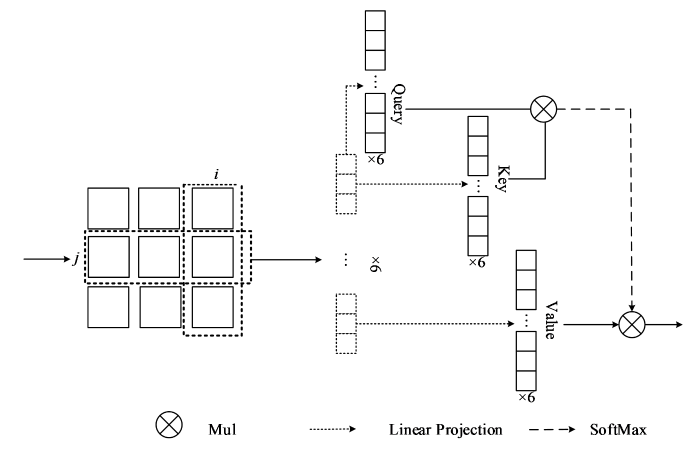
特征图的元素之间存在一定的关联,传统Unet网络缺乏对特征图内像素间相关性的建模,导致模型的特征学习能力不够全面,分割精度不高.基于Transformer的自注意力机制能够捕捉特征图内像素之间的长距离依赖关系,从而获得特征内部的自相关性.但以整张图为单位构建自相关特征不仅计算量大,而且所构建的自注意力特征图缺少层次性,为此,提出一种改进的Transformer自注意力机制,其结构如图1中的MATM模块图中的虚线框内所示.该自注意力机制可处理特征之间的局部自相关性和全局自相关性(分别由LSA模块和GSA模块完成),并将生成的两个自相关性矩阵融合形成新的自注意力特征图.
图2
图3
基于Transformer的自注意力模块的具体计算公式如下:
其中,X表示输入特征,Clocal代表局部自注意力的输出,Cglobal代表全局自注意力的输出,X′代表进行最大池化后的输出;XQ、XK、XV、X′Q、X′K、X′V表示特征图分别乘以Q、K、V矩阵后的结果,dk代表缩放系数,此处为K矩阵的列维度数.
1.2.2 外部注意力模块
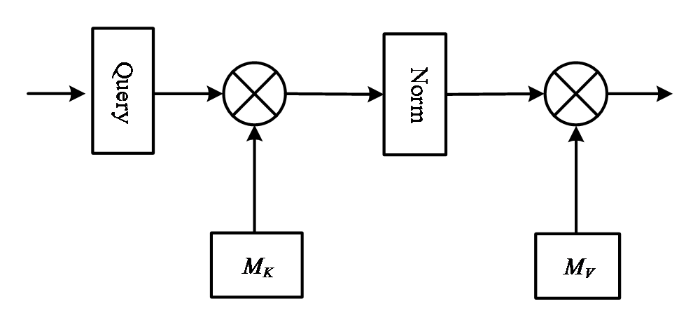
前述的Transformer自注意力模块是计算同一个样本内的自相关性.然而,该模块不具备构建不同样本间相关性的能力.为此,本文引入了EA模块[19].
EA模块用于处理各样本之间的相关性.该模块使用了两块可学习的共享记忆单元MK和MV.通过不同样本的不断更迭,可学习的MK和MV能够更新参数记录下目前为止所有样本中高频出现的重要特征.随后,当前样本特征向量与MK和MV进行矩阵运算,输出当前样本与目前为止样本中有重要相关性的特征向量.具体结构如图4所示.
图4
1.3 可变形卷积模块
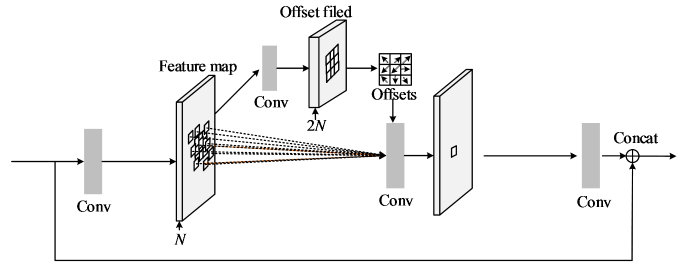
传统CNNs卷积对输入按固定的卷积核尺寸进行过滤操作.DCM[20]是在固定采样点附近进行偏移采样,即为卷积核的每一个采样点增加一对x、y方向的偏移,通过采样偏移的变化改变卷积的采样位置,从而产生卷积核的形变效果,以提取到不规则形状下的特征信息.本文分割的脑胶质瘤区域具有不同形状和规则的特点,使用DCM的卷积核能够更好地感知脑肿瘤区域的特征,有利于提升脑肿瘤区域的分割精度.同时,对DCM进行了改进,即在两端加入了两个卷积(Conv)以调整输入到DCM的特征尺寸和通道,并在输出端进行拼接(Concat)后输出.
DCM结构如图5所示.首先,为调整输入的尺寸和通道,对输入进行一次卷积(Conv),得到卷积后的特征图(Feature map).随后,将Feature map输入给两条并行支路,一条支路生成采样偏移场(Offset field),并将采样偏移量与另一条平行的标准卷积支路配合使用,进行可变形卷积.对于N通道的Feature map,由于每个通道上的固定采样点需要x和y两个方向上的偏移值,因此,需生成2N个通道的偏移场.偏移场是网络结构的一部分,可以通过标准卷积计算得到,故可通过梯度反向传播进行端到端的学习.此外,由于使用卷积来预测偏移,得到的偏移值往往是一个小数,因此使用双线性差值的采样方式,即当前采样的像素点的值取决于偏移之后的浮点位置的周围四个整数邻居.进行完上述操作之后,再对可变形卷积的输出进行一次卷积(Conv),得到与输入尺寸和通道相同的特征,再与输入特征进行拼接(Concat)后输出.
图5
由于编码器得到的特征图对肿瘤区域的识别至关重要,且对几何信息更敏感,故本文将DCM放置在网络的跳跃连接上,将每一层编码器的输出作为DCM的输入.
2 实验部分
2.1 数据集
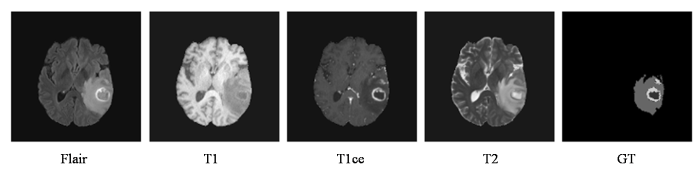
本文采用的数据集来自于2019年多模态脑肿瘤分割挑战赛(BraTS)提供的公开数据集.BraTS2019含有335例样本,其中HGG 259例,LGG 76例.每例样本的每个模态的磁共振图像大小为240×240×155. 每个病例均包含4种模态的MRI数据(Flair、T1ce、T1、T2),各模态均已配准至相同的图像空间并去除头骨部分,且每例样本均提供了专家的分割标注,图6为4个模态下单张切片和标签的示意图,图中GT(Ground Truth)表示金标准.
图6
2.2 预处理
由于本文使用的数据集有4个模态且需要分割的内容为3个标签,标签分别为WT、ET和CT.因此该数据集为一个多模态多标签的数据集,针对这样一个数据集,数据预处理显得尤为重要,本文对数据样本做了以下2种处理.
标准化:为了解决不同模态的磁共振图像存在的对比度差异问题,本文采用z-score方式来对每个图像进行标准化,将每个模态的数据标准化为零均值和单位方差,可表示为:
其中Xmean、Xstd、Xi、X′i分别表示为均值、标准差、原始数据和z-score归一化数据.
切片筛选:考虑到训练过程中的存储和计算问题,我们设计的模型是2D模型,而数据集中的影像是3D数据,因此,需要将3D数据转为2D切片.数据集中每个病例包含4个模态的3D影像,由于脑肿瘤只出现在大脑的局部,所以我们只选取含有脑肿瘤的切片,共得到88 568张2D切片.此外,为减少计算量,我们将每张切片的尺寸统一调整为160×160大小.
2.3 损失函数
本文采用的损失函数为BCE Dice loss[21],它是由BCE(Binary Cross Entropy)Loss和Dice loss组合而成.BCE Dice loss结合了BCE loss和Dice loss的优点,同时考虑了像素级别和区域级别的相似度,可以在保证像素级别和区域级别准确性的同时,缓解梯度消失和过拟合等问题,有助于提高模型的泛化能力和鲁棒性.
2.4 实验环境与评价指标
实验使用的软件环境为Pytorch 0.4.0、python 3.6后端、CUDA 11.0架构平台、CuDNN10.1深度神经网络计算库;硬件环境是Ubuntu18.0、NVIDIA GeForce RTX 2080 GPU,内存为22 G.在训练过程中,批大小为20;设置初始学习率为3e-4,优化模式采用Adam随机梯度下降优化器;设置迭代次数100次.对于分割性能的评估,使用Dice相似系数(Dice similarity coefficient,Dice)、豪斯多夫距离(Hausdorff Distance)、灵敏度(Sensitivity)和阳性预测值(Positive Predictive,PPV)作为评价标准.计算公式分别为:
其中,TP(True Positive)表示为真阳性;TN(True Negative)表示为真阴性;FP(False Positive)表示为假阳性;FN(False Negative)表示假阴性.h(A,B)和h(B,A)指的是分别从A集合到B集合以及从B集合到A集合的单向Hausdorff距离,a和b表示A集合与B集合中的任意一点,同时为了排除一些离合点所造成的不合理的距离,保持整体数值的稳定性,一般取豪斯多夫距离(Hausdorff Distance)的95%作为评价值,即Hausdorff_95,单位为毫米(mm).Hausdorff_95指标是指分割结果的边界与Ground Truth边界之间的距离,距离越小代表分割边界越贴近Ground Truth边界.
3 结果与分析
3.1 对比实验分析
为验证本文所提方法的有效性和收敛性,将本文所提模型与现有经典模型TransUnet[22]、DeepResUnet[23]、Unet++[24]进行对比,所有实验采用与本文相同的训练设置,如损失函数、优化器等.实验采用五折交叉验证方法,将筛选后的88 568张切片数据均分为5个子集,每次选取3个子集作为训练集,剩余2个子集作为验证集与测试集.测试集对比结果如表1所示,DeepResUnet模型和Unet++模型分割精度明显优于Unet模型,WT、TC和ET结构区域的Dice系数、Hausdoff_95指标、PPV和Sensitivity平均值(mean value,MV)相较于Unet分别改善了1.30%、0.027 mm、2.51%、0.88%和1.81%、0.004 mm、1.11%、1.58%.原因在于DeepResUnet模型和Unet++模型是通过增加网络连接的复杂性对Unet模型进行改进,因此分割精度较Unet模型有所改善.但DeepResUnet模型和Unet++在特征提取时没有考虑特征间的相关性.而TransUnet是在Unet基础上增加了全局自注意力Transform机制,使得模型能够专注于脑胶质瘤的病变区域,分割精度不仅明显高于传统Unet模型,而且大部分指标均高于Unet++和DeepResUnet.这说明相比于增加模型连接复杂度,自注意力机制对提升分割精度更具优势,而本文方法不仅增加了自注意力机制,还增加了可变形卷积,可进一步优化脑肿瘤病变边缘分割,本文提出的模型在WT、TC和ET结构区域Dice系数、Hausdoff_95指标、PPV和Sensitivity平均值(MV)为85.53%、2.252 8 mm、85.54%和88.01%,可以看出本文模型的分割精度相比所有对比模型最高.由以上结果可知,本文提出的模型分割性能最佳.
表1 BraTs2019数据集上不同网络的分割
Table 1
| 网络 | Dice/% | Hausdorff_95 | PPV/% | Sensitivity/% | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| WT | TC | ET | MV | WT | TC | ET | MV | WT | TC | ET | MV | WT | TC | ET | MV | |
| Unet | 83.24 | 84.57 | 76.84 | 81.55 | 2.6167 | 1.6551 | 2.7735 | 2.3488 | 85.57 | 85.34 | 78.52 | 83.14 | 85.94 | 90.58 | 80.55 | 85.69 |
| DeepResUnet | 85.00 | 84.80 | 78.74 | 82.85 | 2.5781 | 1.6267 | 2.7588 | 2.3212 | 87.67 | 88.50 | 80.79 | 85.65 | 86.55 | 91.24 | 81.92 | 86.57 |
| Unet++ | 85.76 | 86.44 | 77.88 | 83.36 | 2.5874 | 1.6902 | 2.7558 | 2.3445 | 86.70 | 86.30 | 79.75 | 84.25 | 87.13 | 92.51 | 82.16 | 87.27 |
| TransUnet | 86.73 | 86.83 | 79.09 | 84.22 | 2.6562 | 1.5829 | 2.7395 | 2.3262 | 86.57 | 88.39 | 80.06 | 85.00 | 87.87 | 92.34 | 83.34 | 87.85 |
| 本文方法 | 88.15 | 87.98 | 80.46 | 85.33 | 2.5637 | 1.5323 | 2.6623 | 2.2528 | 87.75 | 88.98 | 79.89 | 85.54 | 88.22 | 92.16 | 83.66 | 88.01 |
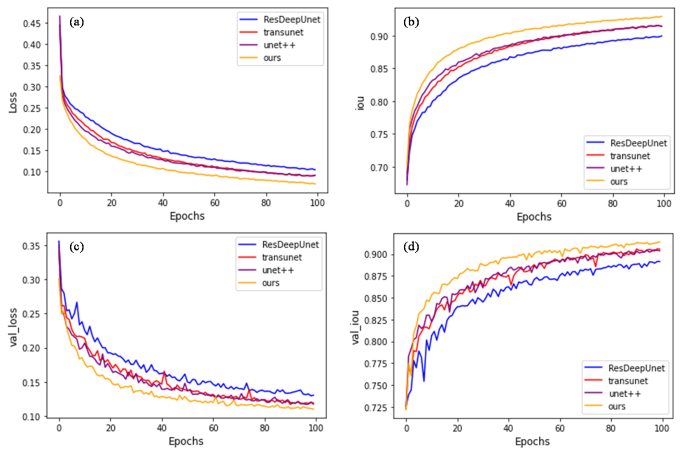
图7依次给出了我们提出的模型与DeepResUnet、Unet++、TransUnet 3个网络模型的训练集和测试集的loss曲线与交并比(Intersection Over Union,IOU)曲线.可以看出,随着迭代次数的增加,四个模型的曲线虽然都趋向平稳,但本文的网络模型梯度下降得更快,同时,模型停止训练时(迭代次数为100),本文模型的loss值最低、IOU值最高.说明本文模型的收敛效果相较于其他模型要好.
图7
图7
不同网络的loss和IOU曲线对比.(a)训练集loss曲线;(b)训练集IOU曲线;(c)测试集loss曲线;(d)测试集IOU曲线
Fig. 7
Comparison of loss curves and IOU curves among different networks. (a)Loss curves of training set; (b) IOU curves of training set; (c) Loss curves of test set; (d) IOU curves of test set
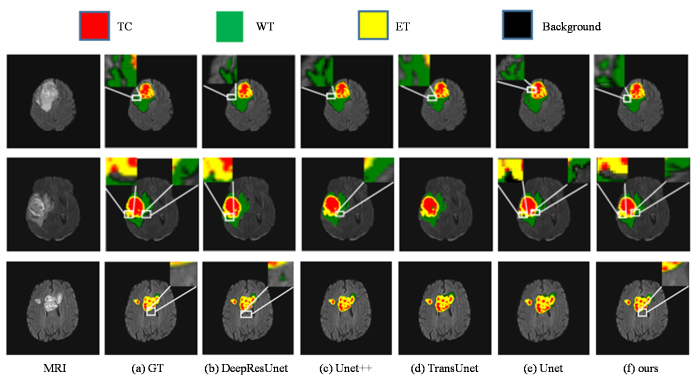
将Unet++、TransUnet、DeepResUnet、Unet和本文提出的方法的分割结果进行可视化,发现Unet++和DeepResUnet都存在着假阳性预测的问题,在图8(b)和图8(c)分割出的肿瘤白框区域在标签中并没有出现,且有较多明显的斑点.这是因为模型缺乏对图片目标区域进行自关注的原因.而本文模型通过添加多重自注意力使得模型能够建立远程依赖关系,增强图片特征的表达能力,能够产生更完整和准确的结果.模型Unet++、TransUnet、DeepResUnet、Unet都存在缺乏边缘敏感性问题,如第一行白框中WT存在着分割出来的脑肿瘤边缘区域不够连续,且存在一些空洞问题,分割效果也较为粗糙.且在8(e)中第二行,Unet和DeepResUnet网络会出现TC分割不完整的情况.本文模型中的可变形卷积很好的解决了这些问题,由于可变形卷积对边缘区域的敏感性,使得最终分割结果有较为完整的边缘和区域.与其他模型相比,本文模型在图片的目标核心以及边缘都表现出了良好的性能.
图8
图8
BraTs2019数据集上不同网络的分割结果的可视化
Fig. 8
Visualization of segmentation results using different models on BraTs2019 dataset
为了进一步验证本文方法在脑胶质瘤分割任务中的有效性,我们将本文所提方法的结果与近两年具有代表性的文献中(包括文献[4]、文献[5]、文献[8]和文献[17])的分割结果进行了对比(表2),其中,本文结果取值均为每个指标中的TC、WT和ET的平均值.除文献[17]采用为BraTs2015数据集外,其他文献所采用的数据均来自于BraTs2019数据集.从表2可以看到,对比文献的Dice指标均低于本文方法,这说明本文的分割结果与Ground Truth更加接近.而本文方法的Hausdorff_95指标低于所有对比文献.尽管文献[8]采用了具有复杂连接的Unet3+网络,文献[4]在跨层连接采用将特征图相乘来扩展通道至二阶维度,文献[17]采用了基于Transformer的生成对抗网络,但它们的分割指标仍然与本文结果差距较大,这说明,单纯增加网络连接复杂度和计算复杂度并不能有效提升模型分割的有效性.文献[5]在跨层连接上引入了三重注意力模块,各项指标与本文方法最接近,而本文也正是侧重于注意力的改进,这说明引入注意力机制是改进脑肿瘤分割的有效途径.
表2 本文所提方法的分割结果与其他文献中的评估指标对比
Table 2
3.2 消融实验
本文模型是以Unet为框架,为了验证添加模块的有效性,本文通过向U-Net添加不同模块来进行消融实验.本消融实验共分为5组:Unet为第一组,Unet+Res为第二组,Unet+Res+DCM为第三组,Unet+Res+MATM为第四组,Unet+Res+DCM+MATM为第五组,其中Res代表残差模块,DCM代表可变形卷积网络,MATM代表多重自注意力模块.从表3第一行与第二行的数据对比可知,Unet+Res组的各项指标均优于Unet分割,说明残差块的添加是有效的.而后,我们在Unet+Res的结构基础上分别单独添加DCM模块和MATM模块.结果显示,分别采用DCM和MATM模块后,模型的大部分指标相比Unet+Res明显提升,由于MATM能够增强提取图片本身的局部性和全局性,建立远程依赖关系,进而提升了分割精度,同时,DCM通过提升模型对肿瘤边缘区域的敏感性进而提升了网络分割性能.但Unet+Res+DCM和Unet+Res+MATM两组的分割指标比较相近.最后,在Unet+Res基础上同时添加DCM和MATM的结果显示,肿瘤分割的各区域的Dice指标、PPV指标和Sensitivity指标均有提升,其中,WT的Dice指标提升的最明显,Sensitivity指标提升也较明显,PPV指标中的TC区域提升也较为明显,Hausdorff_95出现微小的下降.考虑到同时采用两种模块对WT的分割有改善,最终将Unet+Res+DCM+MATM作为本文的最终模型.
表3 添加不同模块的Unet网络在BraTs2019数据集上的分割结果
Table 3
| 网络 | Dice/% | Hausdorff_95/mm | PPV/% | Sensitivity/% | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| WT | TC | ET | WT | TC | ET | WT | TC | ET | WT | TC | ET | ||||
| Unet | 83.24 | 84.57 | 76.84 | 2.6167 | 1.6551 | 2.7735 | 85.57 | 85.34 | 78.52 | 85.94 | 90.58 | 80.55 | |||
| Unet+Res | 84.81 | 85.35 | 77.32 | 2.5977 | 1.6414 | 2.7408 | 86.06 | 85.95 | 78.64 | 86.47 | 90.89 | 81.23 | |||
| Unet+Res+DCM | 86.67 | 85.57 | 78.23 | 2.5677 | 1.6143 | 2.4234 | 87.07 | 86.50 | 78.79 | 86.55 | 91.24 | 81.92 | |||
| Unet+Res+MATM | 86.88 | 87.24 | 79.45 | 2.5681 | 1.5667 | 2.7588 | 87.70 | 86.30 | 79.75 | 87.13 | 92.01 | 82.16 | |||
| Unet+Res+DCM+MATM (本文方法) | 88.15 | 87.98 | 80.46 | 2.5637 | 1.5323 | 2.6623 | 87.75 | 88.98 | 79.89 | 88.22 | 92.16 | 83.66 | |||
此外,为验证多模态分割优于单一模态的分割效果,本文将网络模型的输入端调整为单通道,目的是只接收单一模态数据的输入,然后分别对四种单一模态的数据进行训练和测试,所有训练设置与多模态相同,得到的评价指标结果如表4所示.从表中可以看到,多模态的分割指标高于所有单一模态的分割,这是由于每种单一模态只对特定的病灶区域具有显著特征,因此分割效果也各有侧重.比如,在WT,分割效果较好的模态有Flair和T2,它们能够更好的分割出脑胶质瘤的水肿病灶区域;在TC区域,T1和T1ce的分割效果较好,而T2的分割效果大幅度下降;在ET区域,Flair和T1ce的分割效果突出.因此,我们在进行脑胶质瘤分割时,采用多模态数据,目的是综合利用这四种模态的信息,以获得更加准确的分割结果.
表4 不同模态的消融实验
Table 4
| 模态 | Dice/% | Hausdorff_95/mm | PPV/% | Sensitivity/% | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| WT | TC | ET | WT | TC | ET | WT | TC | ET | WT | TC | ET | ||||
| Flair | 86.63 | 84.71 | 78.46 | 2.5831 | 1.5842 | 2.6957 | 86.07 | 86.84 | 79.76 | 86.54 | 89.75 | 81.75 | |||
| T1 | 84.83 | 86.94 | 77.23 | 2.6011 | 1.5548 | 2.7634 | 84.51 | 88.07 | 77.61 | 85.24 | 90.84 | 78.21 | |||
| T1ce | 84.61 | 87.26 | 79.26 | 2.6109 | 1.5424 | 2.6847 | 84.47 | 88.51 | 80.47 | 85.19 | 91.57 | 82.47 | |||
| T2 | 87.05 | 84.09 | 77.48 | 2.5785 | 1.6042 | 2.7498 | 86.64 | 85.89 | 77.89 | 86.67 | 88.43 | 78.64 | |||
| Multi modal (本文方法) | 88.15 | 87.98 | 80.46 | 2.5637 | 1.5323 | 2.6623 | 87.75 | 88.98 | 81.09 | 88.22 | 92.16 | 83.66 | |||
4 结论
针对脑肿瘤图像分割精度不足的问题,本文提出一种基于MATM和DCM的U-Net改进模型用于脑胶质瘤磁共振影像的分割.模型将原始U-Net框架的标准卷积替换为残差模块,防止模型训练过程中出现梯度消失,并通过在瓶颈层加入基于Transformer的MATM来提取局部特征和全局上下文信息,更好地挖掘出像素间的相关性.此外,在跨层连接处采用DCM来增强模型对形状感知的敏感性,提升了肿瘤边缘特征的提取能力.在BraTS2019数据集上的实验结果表明,本文方法在各类指标上相较于对比文献和主流模型整体表现更为优异,且对肿瘤病灶边缘区域分割更精确,能够更好的完成脑部肿瘤的分割任务.
从理论上讲,注意力机制可以提升分割精度,因为它可以增加网络对可能的目标区域的关注度.但分割精度的提升不能仅仅只依靠注意力机制,精度提升应该是一个全方位的综合策略的实施效果.此外,网络不是越复杂越好,过于复杂会使网络训练产生过拟合.本文在使用注意力机制的同时也配合了其他改进策略,同时,通过本文的消融实验结果可以看出,添加多种改进模块后,网络模型并没有产生过拟合,其分割结果呈现越来越好的分割指标.
尽管磁共振数据是3D影像数据,但本文没有采用3D网络模型,原因是3D模型的计算量太大,训练数据量和存储空间在现有的实验条件下很难保障.下一步工作中,拟探索支持小样本的3D分割模型设计方法,并将其运用在更多的脑肿瘤医学图像的分割任务中.
利益冲突
无
参考文献
U-net: Convolutional networks for biomedical image segmentation
[C]//
Brainseg-net: Brain tumor mr image segmentation via enhanced encoder-decoder network
[J].
DOI:10.3390/diagnostics11020169
URL
[本文引用: 1]

Efficient segmentation of Magnetic Resonance (MR) brain tumor images is of the utmost value for the diagnosis of tumor region. In recent years, advancement in the field of neural networks has been used to refine the segmentation performance of brain tumor sub-regions. The brain tumor segmentation has proven to be a complicated task even for neural networks, due to the small-scale tumor regions. These small-scale tumor regions are unable to be identified, the reason being their tiny size and the huge difference between area occupancy by different tumor classes. In previous state-of-the-art neural network models, the biggest problem was that the location information along with spatial details gets lost in deeper layers. To address these problems, we have proposed an encoder–decoder based model named BrainSeg-Net. The Feature Enhancer (FE) block is incorporated into the BrainSeg-Net architecture which extracts the middle-level features from low-level features from the shallow layers and shares them with the dense layers. This feature aggregation helps to achieve better performance of tumor identification. To address the problem associated with imbalance class, we have used a custom-designed loss function. For evaluation of BrainSeg-Net architecture, three benchmark datasets are utilized: BraTS2017, BraTS 2018, and BraTS 2019. Segmentation of Enhancing Core (EC), Whole Tumor (WT), and Tumor Core (TC) is carried out. The proposed architecture have exhibited good improvement when compared with existing baseline and state-of-the-art techniques. The MR brain tumor segmentation by BrainSeg-Net uses enhanced location and spatial features, which performs better than the existing plethora of brain MR image segmentation approaches.
MM-UNet: A multimodality brain tumor segmentation network in MRI images
[J].
DOI:10.3389/fonc.2022.950706
URL
[本文引用: 1]

The global annual incidence of brain tumors is approximately seven out of 100,000, accounting for 2% of all tumors. The mortality rate ranks first among children under 12 and 10th among adults. Therefore, the localization and segmentation of brain tumor images constitute an active field of medical research. The traditional manual segmentation method is time-consuming, laborious, and subjective. In addition, the information provided by a single-image modality is often limited and cannot meet the needs of clinical application. Therefore, in this study, we developed a multimodality feature fusion network, MM-UNet, for brain tumor segmentation by adopting a multi-encoder and single-decoder structure. In the proposed network, each encoder independently extracts low-level features from the corresponding imaging modality, and the hybrid attention block strengthens the features. After fusion with the high-level semantic of the decoder path through skip connection, the decoder restores the pixel-level segmentation results. We evaluated the performance of the proposed model on the BraTS 2020 dataset. MM-UNet achieved the mean Dice score of 79.2% and mean Hausdorff distance of 8.466, which is a consistent performance improvement over the U-Net, Attention U-Net, and ResUNet baseline models and demonstrates the effectiveness of the proposed model.
Second-order ResU-Net for automatic MRI brain tumor segmentation
[J].
DOI:10.3934/mbe.2021251
PMID:34517471
[本文引用: 4]

Tumor segmentation using magnetic resonance imaging (MRI) plays a significant role in assisting brain tumor diagnosis and treatment. Recently, U-Net architecture with its variants have become prevalent in the field of brain tumor segmentation. However, the existing U-Net models mainly exploit coarse first-order features for tumor segmentation, and they seldom consider the more powerful second-order statistics of deep features. Therefore, in this work, we aim to explore the effectiveness of second-order statistical features for brain tumor segmentation application, and further propose a novel second-order residual brain tumor segmentation network, i.e., SoResU-Net. SoResU-Net utilizes a number of second-order modules to replace the original skip connection operations, thus augmenting the series of transformation operations and increasing the non-linearity of the segmentation network. Extensive experimental results on the BraTS 2018 and BraTS 2019 datasets demonstrate that SoResU-Net outperforms its baseline, especially on core tumor and enhancing tumor segmentation, illuminating the effectiveness of second-order statistical features for the brain tumor segmentation application.
Triple attention segmentation network for brain tumor images
[J].
基于三重注意力的脑肿瘤图像分割网络
[J].
Unet++: A nested u-net architecture for medical image segmentation
[C]// Deep Learning in Medical Image Analysis and Multimodal Learning for Clinical Decision Support:4th International Workshop,
Unet 3+: A full-scale connected unet for medical image segmentation
[C]//
Improved U-Net3+ with stage residual for brain tumor segmentation
[J].
DOI:10.1186/s12880-021-00730-0
[本文引用: 4]

Regulation of temperature is clinically important in the care of neonates because it has a significant impact on prognosis. Although probes that make contact with the skin are widely used to monitor temperature and provide spot central and peripheral temperature information, they do not provide details of the temperature distribution around the body. Although it is possible to obtain detailed temperature distributions using multiple probes, this is not clinically practical. Thermographic techniques have been reported for measurement of temperature distribution in infants. However, as these methods require manual selection of the regions of interest (ROIs), they are not suitable for introduction into clinical settings in hospitals. Here, we describe a method for segmentation of thermal images that enables continuous quantitative contactless monitoring of the temperature distribution over the whole body of neonates.
Generating long sequences with sparse transformers
[J].
Learning texture transformer network for image super-resolution
[C]//
An image is worth 16×16 words: Transformers for image recognition at scale
[J].
Training data-efficient image transformers & distillation through attention
[C]//
Swin transformer: Hierarchical vision transformer using shifted windows
[C]//
Levit: a vision transformer in convnet's clothing for faster inference
[C]//
Transunet: Transformers make strong encoders for medical image segmentation
[J].
Transbts: Multimodal brain tumor segmentation using transformer
[C]// Medical Image Computing and Computer Assisted Intervention-MICCAI 2021: 24th International Conference, Strasbourg, France, Proceedings, Part I 24. Springer International Publishing,
A transformer-based generative adversarial network for brain tumor segmentation
[J].
Axial attention in multidimensional transformers
[J].
Beyond self-attention: External attention using two linear layers for visual tasks
[J].
Deformable convolutional networks
[C]//
Brain Segmentation Based on UNet++ with Weighted Parameters and Convolutional Neural Network
[C]//
Transunet: Transformers make strong encoders for medical image segmentation
[J].
Automatic land-water classification using multispectral airborne LiDAR data for near-shore and river environments
[J].DOI:10.1016/j.isprsjprs.2019.04.005 URL [本文引用: 1]
Unet++: Redesigning skip connections to exploit multiscale features in image segmentation
[J].
Attention is all you need
[C]//