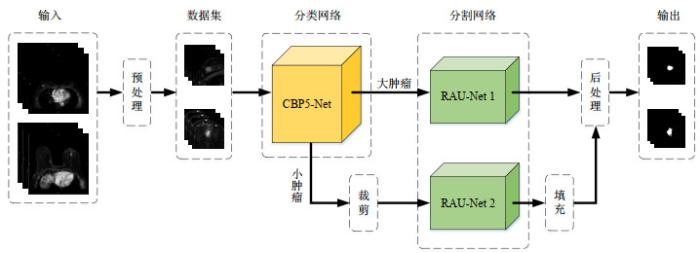
图1
本文实验流程
Fig.1
Experimental process in this research
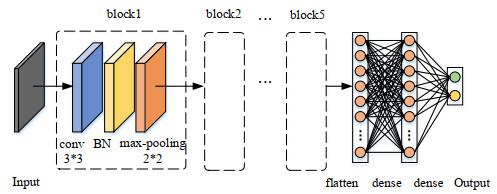
图2
CBP5-Net的网络结构图
Fig.2
The network structure diagram of CBP5-Net
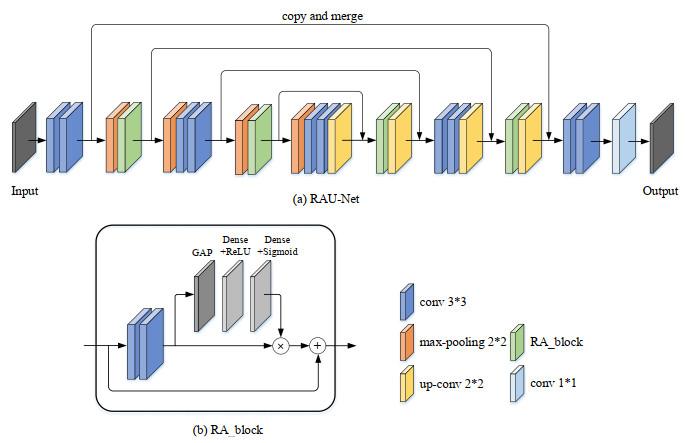
图3
RAU-Net结构图. (a) RAU-Net网络;(b) RA_block模块
Fig.3
The network structure diagram of RAU-Net. (a) RAU-Net; (b) RA_block
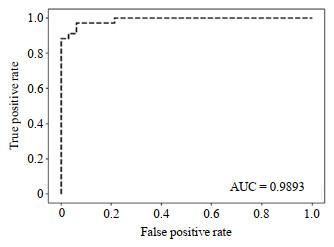
图4
CBP5-Net分类网络的ROC曲线
Fig.4
The ROC curve of classification model CBP5-Net
图5
使用本文所提方法(CBP5-Net+RAU-Net)得到的分割结果
Fig.5
Segmentation results of image samples using CBP5-Net+RAU-Net method proposed in this research
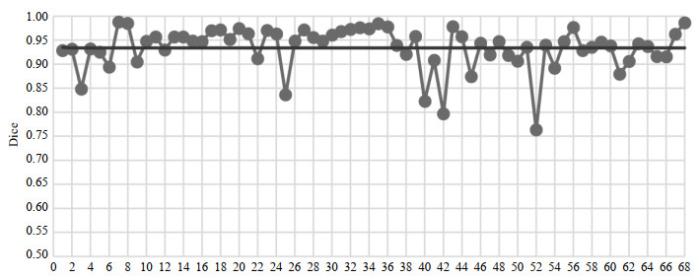
图6
使用本文所提方法(CBP5-Net+RAU-Net)得到的分割结果的Dice系数分布
Fig.6
The Dice coefficient distribution using CBP5-Net+RAU-Net method proposed in this research
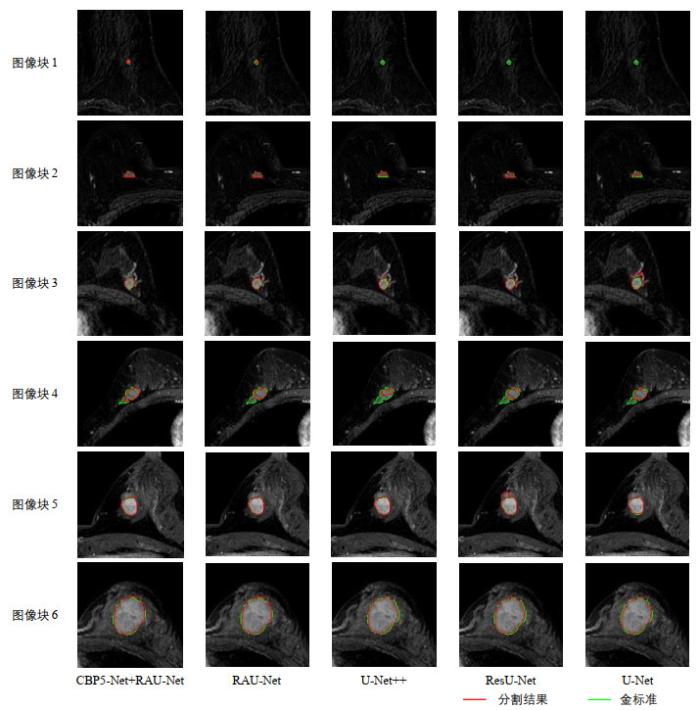
图7
多种方法分割结果的比较
Fig.7
Comparison of segmentation results of various methods
[1]
SIEGEL R L , MILLE R K D , JEMAL A . Cancer statistics, 2018
[J]. CA: A Cancer Journal for Clinicians, 2018 , 68 (1 ): 7 - 30 .
DOI:10.3322/caac.21442
[本文引用: 1]
[2]
FOROUZANFAR M H , FOREMAN K J , DELOSSANTOS A M , et al . Breast and cervical cancer in 187 countries betweem 1980 and 2010: a systematic analysis
[J]. Lancet, 2011 , 378 (9801 ): 1461 - 1484 .
DOI:10.1016/S0140-6736(11)61351-2
[本文引用: 1]
[3]
WILD C P, WEIDERPASS E, STEWART B W. World cancer report: cancer research for cancer prevention[M]//Lyon: International Agency for Research on Cancer, 2020: 23-33.
[本文引用: 1]
[5]
SMITH R A , COKKINDES V , BROOKS D , et al . Cancer screening in the United States, 2011
[J]. CA-Cancer J Clin, 2011 , 61 (1 ): 8 - 30 .
DOI:10.3322/caac.20096
[本文引用: 1]
[6]
DIDKOWSKA J , WOJCIECHOWSKA U . WHO position paper on mammography screening
[J]. Oncology in Clinical Practice, 2014 ,
[本文引用: 1]
[9]
BHOOSHAN N , GIGER M L , JANSEN S A , et al . Cancerous breast lesions on dynamic contrast-enhanced MR images: computerized characterization for image-based prognostic markers
[J]. Radiology, 2010 , 254 (3 ): 680 - 690 .
DOI:10.1148/radiol.09090838
[本文引用: 1]
[10]
杨骞. 基于DCE-MRI影像的乳腺癌早期诊断研究[D]. 杭州: 杭州电子科技大学, 2014.
[本文引用: 1]
[11]
EADIE L H , TAYLOR P , GIBSON A P . A systematic review of computer-assisted diagnosis in diagnostic cancer imaging
[J]. Eur J Radiol, 2011 , 81 (1 ): e70 - e76 .
[本文引用: 1]
[12]
REIG B , HEACOCK L , GERAS K J , et al . Machine learning in breast MRI
[J]. J Magn Reson Imaging, 2020 , 52 (4 ): 998 - 1018 .
DOI:10.1002/jmri.26852
[本文引用: 1]
[13]
TAN Z , HONG R R , YE S Z . A method to extract the region of interest in dynamic contrast-enhanced MR imaging of breast cancer
[J]. Journal of Fuzhou University (Natural Science Edition) , 2016 , 44 (1 ): 33 - 39 .
URL
[本文引用: 1]
檀哲 , 洪容容 , 叶少珍 . 一种提取乳腺癌DCE-MRI感兴趣区域的分割方法
[J]. 福州大学学报(自然科学版), 2016 , 44 (1 ): 33 - 39 .
URL
[本文引用: 1]
[14]
QIAO M Y , SUO S T , CHENG F , et al . Three-dimensional breast tumor segmentation on DCE-MRI with a multilabel attention-guided joint-phase-learning network
[J]. Comput Med Imag Grap, 2021 , 90 , 101909 .
DOI:10.1016/j.compmedimag.2021.101909
[本文引用: 2]
[15]
LIU T T , XU H Y , JIN W , et al . Medical image segmentation based on a hybrid region-based active contour model
[J]. Comput Math Methods Med, 2014 , 890725 .
[本文引用: 1]
[16]
AL-FARIS A Q , NGAH U K , ISA N A M , et al . Computer-aided segmentation system for breast MRI tumour using modified automatic seeded region growing (BMRI-MASRG)
[J]. J Digit Imaging, 2014 , 27 (1 ): 133 - 144 .
DOI:10.1007/s10278-013-9640-5
[17]
CHEN X, WILLIAMS B M, VALLABHANENI S R, et al. Learning active contour models for medical image segmentation[C]//CVF Conference on Computer Vision and Pattern Recognition (CVPR). IEEE, 2019.
[本文引用: 1]
[18]
KANNAN S R , RAMATHILAGAM S , DEVI P , et al . Improved fuzzy clustering algorithms in segmentation of DC-enhanced breast MRI
[J]. J Med Syst, 2012 , 36 (1 ): 321 - 333 .
DOI:10.1007/s10916-010-9478-z
[本文引用: 1]
[19]
BAI P R , LIU Q Y , LEI L , et al . A novel region-based level set method initialized with mean shift clustering for automated medical image segmentation
[J]. Comput Biol Med, 2013 , 43 (11 ): 1827 - 1832 .
DOI:10.1016/j.compbiomed.2013.08.024
[本文引用: 1]
[20]
PEREIRA S , PINTO A , ALVES V , et al . Brain tumor segmentation using convolutional neural networks in MRI images
[J]. IEEE T Med Imaging, 2016 , 35 (5 ): 1240 - 1251 .
DOI:10.1109/TMI.2016.2538465
[本文引用: 1]
[21]
DOLZ J, AYED I B, DESROSIERS C. Dense multi-path U-Net for ischemic stroke lesion segmentation in multiple image modalities[C]//International MICCAI Brainlesion Workshop. Springer, Cham, 2018.
[22]
LEE P Q , GUIDA A , PATTERSON S , et al . Model-free prostate cancer segmentation from dynamic contrast-enhanced MRI with recurrent convolutional networks: A feasibility study
[J]. Comput Med Imag Grap, 2019 , 75 , 14 - 23 .
DOI:10.1016/j.compmedimag.2019.04.006
[本文引用: 1]
[23]
AMIT G, HADAD O, ALPERT S, et al. Hybrid mass detection in breast MRI combining unsupervised saliency analysis and deep learning[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention, MICCAI 2017.
[本文引用: 1]
[24]
BENJELLOUN M, ADOUI M E, LARHMAM M A, et al. Automated breast tumor segmentation in DCE-MRI using deep learning[C]//International Conference on Cloud Computing Technologies and Applications, 2018.
[本文引用: 1]
[25]
LONG J, SHELHAMER E, DARRELL T. Fully convolutional networks for semantic segmentation[C]//IEEE Conference on Computer Vision and Pattern Recognition. IEEE Computer Society, 2015: 3431-3440.
[本文引用: 1]
[26]
ZHANG J, SAHA A, ZHU Z, et al. Breast tumor segmentation in DCE-MRI using fully convolutional networks with an application in radiogenomics[C]//Computer-aided Diagnosis, 2018.
[本文引用: 1]
[27]
ZHANG J , SAHA A , ZHU Z , et al . Hierarchical convolutional neural networks for segmentation of breast tumors in MRI with application to radiogenomics
[J]. IEEE T Med Imaging, 2019 , 38 (2 ): 435 - 447 .
DOI:10.1109/TMI.2018.2865671
[本文引用: 1]
[28]
ADOUI M E , MAHMOUDI S A , LARHMAM M A , et al . MRI breast tumor segmentation using different encoder and decoder CNN architectures
[J]. Computers, 2019 , 8 (3 ): 52 .
DOI:10.3390/computers8030052
[本文引用: 1]
[29]
LOFFE S , SZEGEDY C . Batch normalization: accelerating deep network training by reducing internal covariate shift
[J]. arXiv preprint, 2015 , 1502.03167 .
[本文引用: 1]
[30]
RONNEBERGER O , FISCHER P , BROX T . U-Net: convolutional networks for biomedical image segmentation[M]. Spring Internation Publishing , 2015 , 234 - 241 .
[本文引用: 1]
[31]
JIN Q G , MENG Z P , SUN C M , et al . RA-UNet: a hybrid deep attention-aware network to extract liver and tumor in CT scans
[J]. Front Bioeng Biotechnol, 2020 , 8 , 605132 .
DOI:10.3389/fbioe.2020.605132
[本文引用: 1]
[32]
HE K M, ZHANG X Y, REN S Q, et al. Deep residual learning for image recognition[C]//Proceedings of 2016 IEEE Conference on Computer Vision and Pattern Recognition. Las Vegas: IEEE, 2016: 770-778.
[本文引用: 1]
[33]
HU J , SHEN L , SUN G , et al . Squeeze-and-excitation networks
[J]. arXiv preprint, 2017 , 1709.01507 .
[本文引用: 1]
[34]
SRIVASTAVA N , HINTON G , KRIZHEVSKY A , et al . Dropout: a simple way to prevent neural networks from overfitting
[J]. J Mach Learn Res, 2014 , 15 (1 ): 1929 - 1958 .
[本文引用: 1]
[35]
BADRINARAYANAN V , KENDALL A , CIPOLLA R . SegNet: a deep convolutional encoder-decoder architecture for image segmentation
[J]. IEEE T Pattern Anal, 2017 , 39 (12 ): 2481 - 2495 .
DOI:10.1109/TPAMI.2016.2644615
[本文引用: 1]
[36]
ZHOU Z W, SIDDIQUEE M M R, TAJBAKHSH N, et al. UNet++: a nested U-Net architecture for medical image segmentation[C]//Deep Learning in Medical Image Anylysis and Multimodal Learning for Clinical Decision Support, 2018: 3-11.
[本文引用: 1]
Cancer statistics, 2018
1
2018
... 乳腺癌是最常见的全球性女性健康问题之一[1 ] ,且发病年龄呈年轻化趋势[2 ] .世界卫生组织下属的国际癌症研究所在2020年发布的全球癌症报告中表示,2018年全球女性乳腺癌的发病率为24.2%,远超其他癌症所占比例[3 ] .另有文献[4 ] 指出乳腺癌的发病率平均每年以0.2%~8%的速度增加.坚持早发现、早诊断、早治疗是降低乳腺癌患者死亡率、提高预后生存率的重要手段[5 , 6 ] .快速发展的医学影像技术为乳腺癌的早期检测和诊断提供了有力支撑,用于乳腺癌检测和诊断的最常用的医学成像方式是乳腺钼靶检查和磁共振成像(magnetic resonance imaging,MRI).乳腺钼靶检查更多的是致力于早期发现乳腺肿瘤.MRI中最常用于乳腺检查的是动态对比增强磁共振成像(dynamic contrast enhanced-MRI,DCE-MRI)[7 ] ,它能够直观显示解剖结构和生理组织特征,是乳腺癌诊断评估的有效工具[8 ] .基于DCE-MRI的肿瘤定量分析通常采用肿瘤磁共振图像的形态、纹理特征[9 ] 等进行表征,这强烈依赖于肿瘤的准确分割,但即使对于专业的放射科医师来说,这也有难度,而且手动分割十分耗时. ...
Breast and cervical cancer in 187 countries betweem 1980 and 2010: a systematic analysis
1
2011
... 乳腺癌是最常见的全球性女性健康问题之一[1 ] ,且发病年龄呈年轻化趋势[2 ] .世界卫生组织下属的国际癌症研究所在2020年发布的全球癌症报告中表示,2018年全球女性乳腺癌的发病率为24.2%,远超其他癌症所占比例[3 ] .另有文献[4 ] 指出乳腺癌的发病率平均每年以0.2%~8%的速度增加.坚持早发现、早诊断、早治疗是降低乳腺癌患者死亡率、提高预后生存率的重要手段[5 , 6 ] .快速发展的医学影像技术为乳腺癌的早期检测和诊断提供了有力支撑,用于乳腺癌检测和诊断的最常用的医学成像方式是乳腺钼靶检查和磁共振成像(magnetic resonance imaging,MRI).乳腺钼靶检查更多的是致力于早期发现乳腺肿瘤.MRI中最常用于乳腺检查的是动态对比增强磁共振成像(dynamic contrast enhanced-MRI,DCE-MRI)[7 ] ,它能够直观显示解剖结构和生理组织特征,是乳腺癌诊断评估的有效工具[8 ] .基于DCE-MRI的肿瘤定量分析通常采用肿瘤磁共振图像的形态、纹理特征[9 ] 等进行表征,这强烈依赖于肿瘤的准确分割,但即使对于专业的放射科医师来说,这也有难度,而且手动分割十分耗时. ...
1
... 乳腺癌是最常见的全球性女性健康问题之一[1 ] ,且发病年龄呈年轻化趋势[2 ] .世界卫生组织下属的国际癌症研究所在2020年发布的全球癌症报告中表示,2018年全球女性乳腺癌的发病率为24.2%,远超其他癌症所占比例[3 ] .另有文献[4 ] 指出乳腺癌的发病率平均每年以0.2%~8%的速度增加.坚持早发现、早诊断、早治疗是降低乳腺癌患者死亡率、提高预后生存率的重要手段[5 , 6 ] .快速发展的医学影像技术为乳腺癌的早期检测和诊断提供了有力支撑,用于乳腺癌检测和诊断的最常用的医学成像方式是乳腺钼靶检查和磁共振成像(magnetic resonance imaging,MRI).乳腺钼靶检查更多的是致力于早期发现乳腺肿瘤.MRI中最常用于乳腺检查的是动态对比增强磁共振成像(dynamic contrast enhanced-MRI,DCE-MRI)[7 ] ,它能够直观显示解剖结构和生理组织特征,是乳腺癌诊断评估的有效工具[8 ] .基于DCE-MRI的肿瘤定量分析通常采用肿瘤磁共振图像的形态、纹理特征[9 ] 等进行表征,这强烈依赖于肿瘤的准确分割,但即使对于专业的放射科医师来说,这也有难度,而且手动分割十分耗时. ...
乳腺癌的分布规律及其危险因素
1
2002
... 乳腺癌是最常见的全球性女性健康问题之一[1 ] ,且发病年龄呈年轻化趋势[2 ] .世界卫生组织下属的国际癌症研究所在2020年发布的全球癌症报告中表示,2018年全球女性乳腺癌的发病率为24.2%,远超其他癌症所占比例[3 ] .另有文献[4 ] 指出乳腺癌的发病率平均每年以0.2%~8%的速度增加.坚持早发现、早诊断、早治疗是降低乳腺癌患者死亡率、提高预后生存率的重要手段[5 , 6 ] .快速发展的医学影像技术为乳腺癌的早期检测和诊断提供了有力支撑,用于乳腺癌检测和诊断的最常用的医学成像方式是乳腺钼靶检查和磁共振成像(magnetic resonance imaging,MRI).乳腺钼靶检查更多的是致力于早期发现乳腺肿瘤.MRI中最常用于乳腺检查的是动态对比增强磁共振成像(dynamic contrast enhanced-MRI,DCE-MRI)[7 ] ,它能够直观显示解剖结构和生理组织特征,是乳腺癌诊断评估的有效工具[8 ] .基于DCE-MRI的肿瘤定量分析通常采用肿瘤磁共振图像的形态、纹理特征[9 ] 等进行表征,这强烈依赖于肿瘤的准确分割,但即使对于专业的放射科医师来说,这也有难度,而且手动分割十分耗时. ...
乳腺癌的分布规律及其危险因素
1
2002
... 乳腺癌是最常见的全球性女性健康问题之一[1 ] ,且发病年龄呈年轻化趋势[2 ] .世界卫生组织下属的国际癌症研究所在2020年发布的全球癌症报告中表示,2018年全球女性乳腺癌的发病率为24.2%,远超其他癌症所占比例[3 ] .另有文献[4 ] 指出乳腺癌的发病率平均每年以0.2%~8%的速度增加.坚持早发现、早诊断、早治疗是降低乳腺癌患者死亡率、提高预后生存率的重要手段[5 , 6 ] .快速发展的医学影像技术为乳腺癌的早期检测和诊断提供了有力支撑,用于乳腺癌检测和诊断的最常用的医学成像方式是乳腺钼靶检查和磁共振成像(magnetic resonance imaging,MRI).乳腺钼靶检查更多的是致力于早期发现乳腺肿瘤.MRI中最常用于乳腺检查的是动态对比增强磁共振成像(dynamic contrast enhanced-MRI,DCE-MRI)[7 ] ,它能够直观显示解剖结构和生理组织特征,是乳腺癌诊断评估的有效工具[8 ] .基于DCE-MRI的肿瘤定量分析通常采用肿瘤磁共振图像的形态、纹理特征[9 ] 等进行表征,这强烈依赖于肿瘤的准确分割,但即使对于专业的放射科医师来说,这也有难度,而且手动分割十分耗时. ...
Cancer screening in the United States, 2011
1
2011
... 乳腺癌是最常见的全球性女性健康问题之一[1 ] ,且发病年龄呈年轻化趋势[2 ] .世界卫生组织下属的国际癌症研究所在2020年发布的全球癌症报告中表示,2018年全球女性乳腺癌的发病率为24.2%,远超其他癌症所占比例[3 ] .另有文献[4 ] 指出乳腺癌的发病率平均每年以0.2%~8%的速度增加.坚持早发现、早诊断、早治疗是降低乳腺癌患者死亡率、提高预后生存率的重要手段[5 , 6 ] .快速发展的医学影像技术为乳腺癌的早期检测和诊断提供了有力支撑,用于乳腺癌检测和诊断的最常用的医学成像方式是乳腺钼靶检查和磁共振成像(magnetic resonance imaging,MRI).乳腺钼靶检查更多的是致力于早期发现乳腺肿瘤.MRI中最常用于乳腺检查的是动态对比增强磁共振成像(dynamic contrast enhanced-MRI,DCE-MRI)[7 ] ,它能够直观显示解剖结构和生理组织特征,是乳腺癌诊断评估的有效工具[8 ] .基于DCE-MRI的肿瘤定量分析通常采用肿瘤磁共振图像的形态、纹理特征[9 ] 等进行表征,这强烈依赖于肿瘤的准确分割,但即使对于专业的放射科医师来说,这也有难度,而且手动分割十分耗时. ...
WHO position paper on mammography screening
1
2014
... 乳腺癌是最常见的全球性女性健康问题之一[1 ] ,且发病年龄呈年轻化趋势[2 ] .世界卫生组织下属的国际癌症研究所在2020年发布的全球癌症报告中表示,2018年全球女性乳腺癌的发病率为24.2%,远超其他癌症所占比例[3 ] .另有文献[4 ] 指出乳腺癌的发病率平均每年以0.2%~8%的速度增加.坚持早发现、早诊断、早治疗是降低乳腺癌患者死亡率、提高预后生存率的重要手段[5 , 6 ] .快速发展的医学影像技术为乳腺癌的早期检测和诊断提供了有力支撑,用于乳腺癌检测和诊断的最常用的医学成像方式是乳腺钼靶检查和磁共振成像(magnetic resonance imaging,MRI).乳腺钼靶检查更多的是致力于早期发现乳腺肿瘤.MRI中最常用于乳腺检查的是动态对比增强磁共振成像(dynamic contrast enhanced-MRI,DCE-MRI)[7 ] ,它能够直观显示解剖结构和生理组织特征,是乳腺癌诊断评估的有效工具[8 ] .基于DCE-MRI的肿瘤定量分析通常采用肿瘤磁共振图像的形态、纹理特征[9 ] 等进行表征,这强烈依赖于肿瘤的准确分割,但即使对于专业的放射科医师来说,这也有难度,而且手动分割十分耗时. ...
MR imaging of the breast using gadolinium-DTPA
1
1986
... 乳腺癌是最常见的全球性女性健康问题之一[1 ] ,且发病年龄呈年轻化趋势[2 ] .世界卫生组织下属的国际癌症研究所在2020年发布的全球癌症报告中表示,2018年全球女性乳腺癌的发病率为24.2%,远超其他癌症所占比例[3 ] .另有文献[4 ] 指出乳腺癌的发病率平均每年以0.2%~8%的速度增加.坚持早发现、早诊断、早治疗是降低乳腺癌患者死亡率、提高预后生存率的重要手段[5 , 6 ] .快速发展的医学影像技术为乳腺癌的早期检测和诊断提供了有力支撑,用于乳腺癌检测和诊断的最常用的医学成像方式是乳腺钼靶检查和磁共振成像(magnetic resonance imaging,MRI).乳腺钼靶检查更多的是致力于早期发现乳腺肿瘤.MRI中最常用于乳腺检查的是动态对比增强磁共振成像(dynamic contrast enhanced-MRI,DCE-MRI)[7 ] ,它能够直观显示解剖结构和生理组织特征,是乳腺癌诊断评估的有效工具[8 ] .基于DCE-MRI的肿瘤定量分析通常采用肿瘤磁共振图像的形态、纹理特征[9 ] 等进行表征,这强烈依赖于肿瘤的准确分割,但即使对于专业的放射科医师来说,这也有难度,而且手动分割十分耗时. ...
Breast cancer imaging with MRI
1
2002
... 乳腺癌是最常见的全球性女性健康问题之一[1 ] ,且发病年龄呈年轻化趋势[2 ] .世界卫生组织下属的国际癌症研究所在2020年发布的全球癌症报告中表示,2018年全球女性乳腺癌的发病率为24.2%,远超其他癌症所占比例[3 ] .另有文献[4 ] 指出乳腺癌的发病率平均每年以0.2%~8%的速度增加.坚持早发现、早诊断、早治疗是降低乳腺癌患者死亡率、提高预后生存率的重要手段[5 , 6 ] .快速发展的医学影像技术为乳腺癌的早期检测和诊断提供了有力支撑,用于乳腺癌检测和诊断的最常用的医学成像方式是乳腺钼靶检查和磁共振成像(magnetic resonance imaging,MRI).乳腺钼靶检查更多的是致力于早期发现乳腺肿瘤.MRI中最常用于乳腺检查的是动态对比增强磁共振成像(dynamic contrast enhanced-MRI,DCE-MRI)[7 ] ,它能够直观显示解剖结构和生理组织特征,是乳腺癌诊断评估的有效工具[8 ] .基于DCE-MRI的肿瘤定量分析通常采用肿瘤磁共振图像的形态、纹理特征[9 ] 等进行表征,这强烈依赖于肿瘤的准确分割,但即使对于专业的放射科医师来说,这也有难度,而且手动分割十分耗时. ...
Cancerous breast lesions on dynamic contrast-enhanced MR images: computerized characterization for image-based prognostic markers
1
2010
... 乳腺癌是最常见的全球性女性健康问题之一[1 ] ,且发病年龄呈年轻化趋势[2 ] .世界卫生组织下属的国际癌症研究所在2020年发布的全球癌症报告中表示,2018年全球女性乳腺癌的发病率为24.2%,远超其他癌症所占比例[3 ] .另有文献[4 ] 指出乳腺癌的发病率平均每年以0.2%~8%的速度增加.坚持早发现、早诊断、早治疗是降低乳腺癌患者死亡率、提高预后生存率的重要手段[5 , 6 ] .快速发展的医学影像技术为乳腺癌的早期检测和诊断提供了有力支撑,用于乳腺癌检测和诊断的最常用的医学成像方式是乳腺钼靶检查和磁共振成像(magnetic resonance imaging,MRI).乳腺钼靶检查更多的是致力于早期发现乳腺肿瘤.MRI中最常用于乳腺检查的是动态对比增强磁共振成像(dynamic contrast enhanced-MRI,DCE-MRI)[7 ] ,它能够直观显示解剖结构和生理组织特征,是乳腺癌诊断评估的有效工具[8 ] .基于DCE-MRI的肿瘤定量分析通常采用肿瘤磁共振图像的形态、纹理特征[9 ] 等进行表征,这强烈依赖于肿瘤的准确分割,但即使对于专业的放射科医师来说,这也有难度,而且手动分割十分耗时. ...
1
... 随着计算机技术、图像处理和模式识别技术的迅速发展,同时为了减轻临床医生的工作量,乳腺计算机辅助诊断(computer-aided detection,CAD)系统应运而生,并且在乳腺癌早期检测和诊断中起到越来越重要的作用.传统的CAD系统主要由三个模块构成[10 ] :(1)图像预处理与感兴趣区域(region of interest,ROI)提取;(2)特征提取与优化;(3)分类与预测.肿瘤分割是ROI提取模块的主要内容,是肿瘤诊断、治疗方案制定和预后预测等工作的重要基础[11 ] .然而,乳腺肿瘤可以出现在乳房的任何位置,大小参差不齐、形态复杂多变,肿瘤区域与背景组织的差异较小,且占整幅图像的面积过小,使得乳腺肿瘤图像的自动分割成为一个具有挑战性的任务.此外,MRI本身的密度不均匀性进一步增加了乳腺肿瘤磁共振图像自动分割的难度[12 , 13 ] . ...
A systematic review of computer-assisted diagnosis in diagnostic cancer imaging
1
2011
... 随着计算机技术、图像处理和模式识别技术的迅速发展,同时为了减轻临床医生的工作量,乳腺计算机辅助诊断(computer-aided detection,CAD)系统应运而生,并且在乳腺癌早期检测和诊断中起到越来越重要的作用.传统的CAD系统主要由三个模块构成[10 ] :(1)图像预处理与感兴趣区域(region of interest,ROI)提取;(2)特征提取与优化;(3)分类与预测.肿瘤分割是ROI提取模块的主要内容,是肿瘤诊断、治疗方案制定和预后预测等工作的重要基础[11 ] .然而,乳腺肿瘤可以出现在乳房的任何位置,大小参差不齐、形态复杂多变,肿瘤区域与背景组织的差异较小,且占整幅图像的面积过小,使得乳腺肿瘤图像的自动分割成为一个具有挑战性的任务.此外,MRI本身的密度不均匀性进一步增加了乳腺肿瘤磁共振图像自动分割的难度[12 , 13 ] . ...
Machine learning in breast MRI
1
2020
... 随着计算机技术、图像处理和模式识别技术的迅速发展,同时为了减轻临床医生的工作量,乳腺计算机辅助诊断(computer-aided detection,CAD)系统应运而生,并且在乳腺癌早期检测和诊断中起到越来越重要的作用.传统的CAD系统主要由三个模块构成[10 ] :(1)图像预处理与感兴趣区域(region of interest,ROI)提取;(2)特征提取与优化;(3)分类与预测.肿瘤分割是ROI提取模块的主要内容,是肿瘤诊断、治疗方案制定和预后预测等工作的重要基础[11 ] .然而,乳腺肿瘤可以出现在乳房的任何位置,大小参差不齐、形态复杂多变,肿瘤区域与背景组织的差异较小,且占整幅图像的面积过小,使得乳腺肿瘤图像的自动分割成为一个具有挑战性的任务.此外,MRI本身的密度不均匀性进一步增加了乳腺肿瘤磁共振图像自动分割的难度[12 , 13 ] . ...
一种提取乳腺癌DCE-MRI感兴趣区域的分割方法
1
2016
... 随着计算机技术、图像处理和模式识别技术的迅速发展,同时为了减轻临床医生的工作量,乳腺计算机辅助诊断(computer-aided detection,CAD)系统应运而生,并且在乳腺癌早期检测和诊断中起到越来越重要的作用.传统的CAD系统主要由三个模块构成[10 ] :(1)图像预处理与感兴趣区域(region of interest,ROI)提取;(2)特征提取与优化;(3)分类与预测.肿瘤分割是ROI提取模块的主要内容,是肿瘤诊断、治疗方案制定和预后预测等工作的重要基础[11 ] .然而,乳腺肿瘤可以出现在乳房的任何位置,大小参差不齐、形态复杂多变,肿瘤区域与背景组织的差异较小,且占整幅图像的面积过小,使得乳腺肿瘤图像的自动分割成为一个具有挑战性的任务.此外,MRI本身的密度不均匀性进一步增加了乳腺肿瘤磁共振图像自动分割的难度[12 , 13 ] . ...
一种提取乳腺癌DCE-MRI感兴趣区域的分割方法
1
2016
... 随着计算机技术、图像处理和模式识别技术的迅速发展,同时为了减轻临床医生的工作量,乳腺计算机辅助诊断(computer-aided detection,CAD)系统应运而生,并且在乳腺癌早期检测和诊断中起到越来越重要的作用.传统的CAD系统主要由三个模块构成[10 ] :(1)图像预处理与感兴趣区域(region of interest,ROI)提取;(2)特征提取与优化;(3)分类与预测.肿瘤分割是ROI提取模块的主要内容,是肿瘤诊断、治疗方案制定和预后预测等工作的重要基础[11 ] .然而,乳腺肿瘤可以出现在乳房的任何位置,大小参差不齐、形态复杂多变,肿瘤区域与背景组织的差异较小,且占整幅图像的面积过小,使得乳腺肿瘤图像的自动分割成为一个具有挑战性的任务.此外,MRI本身的密度不均匀性进一步增加了乳腺肿瘤磁共振图像自动分割的难度[12 , 13 ] . ...
Three-dimensional breast tumor segmentation on DCE-MRI with a multilabel attention-guided joint-phase-learning network
2
2021
... 对于肿瘤图像的分割,目前已有很多自动的、半自动的方法,主要可以分为以下三类[14 ] :基于轮廓的方法[15 -17 ] 、基于模型的方法[18 , 19 ] 和基于数据的方法[20 -22 ] .基于轮廓的方法主要包括基于活动轮廓模型、主动轮廓模型、区域生长算法等方法;基于模型的方法主要包括基于模糊均值、聚类等传统机器学习的方法.这两类方法对图像质量敏感,精度相对较低且耗时较长.基于数据的方法主要是指基于一定规模数据集用于训练的深度学习的方法.随着深度学习算法的发展,越来越多基于数据的卷积神经网络的图像分割方法得以开发,这类方法虽然需要一定数量的标注数据用于网络训练,但可以得到相对较高的分割精度.已有很多方法被应用于基于DCE-MRI的乳腺肿瘤的自动分割,并且取得了不错的成效,如基于卷积神经网络(convolutional neural network,CNN)的分割方法[14 , 23 , 24 ] 、基于全卷积神经网络(fully convolutional networks,FCN)[25 ] 的方法,其中基于FCN的方法的分割性能尤为突出.Zhang等[26 , 27 ] 在2018年、2019年分别提出了基于FCN的乳腺肿瘤DCE-MR图像分割的两个模型,平均Dice相似性系数(Dice)均达到0.72;Adoui等[28 ] 提出分别基于SegNet和U-Net的两个FCN用于乳腺DCE-MR图像的肿瘤分割,但由于数据量有限,平均交并比(intersection over union,IoU)只分别达到0.688 8和0.741 6.但目前这些基于FCN的乳腺肿瘤磁共振图像的分割效果还不够理想,主要存在以下问题:(1)乳腺肿瘤大小参差不齐,将所有图像一并投入同一分割网络进行训练,部分小目标肿瘤容易被遗漏;(2)传统的基于全卷积网络的模型中默认特征图的每个通道的权重是相等的,从而丢失了不同通道之间的权重差异所隐藏的信息. ...
... [14 , 23 , 24 ]、基于全卷积神经网络(fully convolutional networks,FCN)[25 ] 的方法,其中基于FCN的方法的分割性能尤为突出.Zhang等[26 , 27 ] 在2018年、2019年分别提出了基于FCN的乳腺肿瘤DCE-MR图像分割的两个模型,平均Dice相似性系数(Dice)均达到0.72;Adoui等[28 ] 提出分别基于SegNet和U-Net的两个FCN用于乳腺DCE-MR图像的肿瘤分割,但由于数据量有限,平均交并比(intersection over union,IoU)只分别达到0.688 8和0.741 6.但目前这些基于FCN的乳腺肿瘤磁共振图像的分割效果还不够理想,主要存在以下问题:(1)乳腺肿瘤大小参差不齐,将所有图像一并投入同一分割网络进行训练,部分小目标肿瘤容易被遗漏;(2)传统的基于全卷积网络的模型中默认特征图的每个通道的权重是相等的,从而丢失了不同通道之间的权重差异所隐藏的信息. ...
Medical image segmentation based on a hybrid region-based active contour model
1
2014
... 对于肿瘤图像的分割,目前已有很多自动的、半自动的方法,主要可以分为以下三类[14 ] :基于轮廓的方法[15 -17 ] 、基于模型的方法[18 , 19 ] 和基于数据的方法[20 -22 ] .基于轮廓的方法主要包括基于活动轮廓模型、主动轮廓模型、区域生长算法等方法;基于模型的方法主要包括基于模糊均值、聚类等传统机器学习的方法.这两类方法对图像质量敏感,精度相对较低且耗时较长.基于数据的方法主要是指基于一定规模数据集用于训练的深度学习的方法.随着深度学习算法的发展,越来越多基于数据的卷积神经网络的图像分割方法得以开发,这类方法虽然需要一定数量的标注数据用于网络训练,但可以得到相对较高的分割精度.已有很多方法被应用于基于DCE-MRI的乳腺肿瘤的自动分割,并且取得了不错的成效,如基于卷积神经网络(convolutional neural network,CNN)的分割方法[14 , 23 , 24 ] 、基于全卷积神经网络(fully convolutional networks,FCN)[25 ] 的方法,其中基于FCN的方法的分割性能尤为突出.Zhang等[26 , 27 ] 在2018年、2019年分别提出了基于FCN的乳腺肿瘤DCE-MR图像分割的两个模型,平均Dice相似性系数(Dice)均达到0.72;Adoui等[28 ] 提出分别基于SegNet和U-Net的两个FCN用于乳腺DCE-MR图像的肿瘤分割,但由于数据量有限,平均交并比(intersection over union,IoU)只分别达到0.688 8和0.741 6.但目前这些基于FCN的乳腺肿瘤磁共振图像的分割效果还不够理想,主要存在以下问题:(1)乳腺肿瘤大小参差不齐,将所有图像一并投入同一分割网络进行训练,部分小目标肿瘤容易被遗漏;(2)传统的基于全卷积网络的模型中默认特征图的每个通道的权重是相等的,从而丢失了不同通道之间的权重差异所隐藏的信息. ...
Computer-aided segmentation system for breast MRI tumour using modified automatic seeded region growing (BMRI-MASRG)
0
2014
1
... 对于肿瘤图像的分割,目前已有很多自动的、半自动的方法,主要可以分为以下三类[14 ] :基于轮廓的方法[15 -17 ] 、基于模型的方法[18 , 19 ] 和基于数据的方法[20 -22 ] .基于轮廓的方法主要包括基于活动轮廓模型、主动轮廓模型、区域生长算法等方法;基于模型的方法主要包括基于模糊均值、聚类等传统机器学习的方法.这两类方法对图像质量敏感,精度相对较低且耗时较长.基于数据的方法主要是指基于一定规模数据集用于训练的深度学习的方法.随着深度学习算法的发展,越来越多基于数据的卷积神经网络的图像分割方法得以开发,这类方法虽然需要一定数量的标注数据用于网络训练,但可以得到相对较高的分割精度.已有很多方法被应用于基于DCE-MRI的乳腺肿瘤的自动分割,并且取得了不错的成效,如基于卷积神经网络(convolutional neural network,CNN)的分割方法[14 , 23 , 24 ] 、基于全卷积神经网络(fully convolutional networks,FCN)[25 ] 的方法,其中基于FCN的方法的分割性能尤为突出.Zhang等[26 , 27 ] 在2018年、2019年分别提出了基于FCN的乳腺肿瘤DCE-MR图像分割的两个模型,平均Dice相似性系数(Dice)均达到0.72;Adoui等[28 ] 提出分别基于SegNet和U-Net的两个FCN用于乳腺DCE-MR图像的肿瘤分割,但由于数据量有限,平均交并比(intersection over union,IoU)只分别达到0.688 8和0.741 6.但目前这些基于FCN的乳腺肿瘤磁共振图像的分割效果还不够理想,主要存在以下问题:(1)乳腺肿瘤大小参差不齐,将所有图像一并投入同一分割网络进行训练,部分小目标肿瘤容易被遗漏;(2)传统的基于全卷积网络的模型中默认特征图的每个通道的权重是相等的,从而丢失了不同通道之间的权重差异所隐藏的信息. ...
Improved fuzzy clustering algorithms in segmentation of DC-enhanced breast MRI
1
2012
... 对于肿瘤图像的分割,目前已有很多自动的、半自动的方法,主要可以分为以下三类[14 ] :基于轮廓的方法[15 -17 ] 、基于模型的方法[18 , 19 ] 和基于数据的方法[20 -22 ] .基于轮廓的方法主要包括基于活动轮廓模型、主动轮廓模型、区域生长算法等方法;基于模型的方法主要包括基于模糊均值、聚类等传统机器学习的方法.这两类方法对图像质量敏感,精度相对较低且耗时较长.基于数据的方法主要是指基于一定规模数据集用于训练的深度学习的方法.随着深度学习算法的发展,越来越多基于数据的卷积神经网络的图像分割方法得以开发,这类方法虽然需要一定数量的标注数据用于网络训练,但可以得到相对较高的分割精度.已有很多方法被应用于基于DCE-MRI的乳腺肿瘤的自动分割,并且取得了不错的成效,如基于卷积神经网络(convolutional neural network,CNN)的分割方法[14 , 23 , 24 ] 、基于全卷积神经网络(fully convolutional networks,FCN)[25 ] 的方法,其中基于FCN的方法的分割性能尤为突出.Zhang等[26 , 27 ] 在2018年、2019年分别提出了基于FCN的乳腺肿瘤DCE-MR图像分割的两个模型,平均Dice相似性系数(Dice)均达到0.72;Adoui等[28 ] 提出分别基于SegNet和U-Net的两个FCN用于乳腺DCE-MR图像的肿瘤分割,但由于数据量有限,平均交并比(intersection over union,IoU)只分别达到0.688 8和0.741 6.但目前这些基于FCN的乳腺肿瘤磁共振图像的分割效果还不够理想,主要存在以下问题:(1)乳腺肿瘤大小参差不齐,将所有图像一并投入同一分割网络进行训练,部分小目标肿瘤容易被遗漏;(2)传统的基于全卷积网络的模型中默认特征图的每个通道的权重是相等的,从而丢失了不同通道之间的权重差异所隐藏的信息. ...
A novel region-based level set method initialized with mean shift clustering for automated medical image segmentation
1
2013
... 对于肿瘤图像的分割,目前已有很多自动的、半自动的方法,主要可以分为以下三类[14 ] :基于轮廓的方法[15 -17 ] 、基于模型的方法[18 , 19 ] 和基于数据的方法[20 -22 ] .基于轮廓的方法主要包括基于活动轮廓模型、主动轮廓模型、区域生长算法等方法;基于模型的方法主要包括基于模糊均值、聚类等传统机器学习的方法.这两类方法对图像质量敏感,精度相对较低且耗时较长.基于数据的方法主要是指基于一定规模数据集用于训练的深度学习的方法.随着深度学习算法的发展,越来越多基于数据的卷积神经网络的图像分割方法得以开发,这类方法虽然需要一定数量的标注数据用于网络训练,但可以得到相对较高的分割精度.已有很多方法被应用于基于DCE-MRI的乳腺肿瘤的自动分割,并且取得了不错的成效,如基于卷积神经网络(convolutional neural network,CNN)的分割方法[14 , 23 , 24 ] 、基于全卷积神经网络(fully convolutional networks,FCN)[25 ] 的方法,其中基于FCN的方法的分割性能尤为突出.Zhang等[26 , 27 ] 在2018年、2019年分别提出了基于FCN的乳腺肿瘤DCE-MR图像分割的两个模型,平均Dice相似性系数(Dice)均达到0.72;Adoui等[28 ] 提出分别基于SegNet和U-Net的两个FCN用于乳腺DCE-MR图像的肿瘤分割,但由于数据量有限,平均交并比(intersection over union,IoU)只分别达到0.688 8和0.741 6.但目前这些基于FCN的乳腺肿瘤磁共振图像的分割效果还不够理想,主要存在以下问题:(1)乳腺肿瘤大小参差不齐,将所有图像一并投入同一分割网络进行训练,部分小目标肿瘤容易被遗漏;(2)传统的基于全卷积网络的模型中默认特征图的每个通道的权重是相等的,从而丢失了不同通道之间的权重差异所隐藏的信息. ...
Brain tumor segmentation using convolutional neural networks in MRI images
1
2016
... 对于肿瘤图像的分割,目前已有很多自动的、半自动的方法,主要可以分为以下三类[14 ] :基于轮廓的方法[15 -17 ] 、基于模型的方法[18 , 19 ] 和基于数据的方法[20 -22 ] .基于轮廓的方法主要包括基于活动轮廓模型、主动轮廓模型、区域生长算法等方法;基于模型的方法主要包括基于模糊均值、聚类等传统机器学习的方法.这两类方法对图像质量敏感,精度相对较低且耗时较长.基于数据的方法主要是指基于一定规模数据集用于训练的深度学习的方法.随着深度学习算法的发展,越来越多基于数据的卷积神经网络的图像分割方法得以开发,这类方法虽然需要一定数量的标注数据用于网络训练,但可以得到相对较高的分割精度.已有很多方法被应用于基于DCE-MRI的乳腺肿瘤的自动分割,并且取得了不错的成效,如基于卷积神经网络(convolutional neural network,CNN)的分割方法[14 , 23 , 24 ] 、基于全卷积神经网络(fully convolutional networks,FCN)[25 ] 的方法,其中基于FCN的方法的分割性能尤为突出.Zhang等[26 , 27 ] 在2018年、2019年分别提出了基于FCN的乳腺肿瘤DCE-MR图像分割的两个模型,平均Dice相似性系数(Dice)均达到0.72;Adoui等[28 ] 提出分别基于SegNet和U-Net的两个FCN用于乳腺DCE-MR图像的肿瘤分割,但由于数据量有限,平均交并比(intersection over union,IoU)只分别达到0.688 8和0.741 6.但目前这些基于FCN的乳腺肿瘤磁共振图像的分割效果还不够理想,主要存在以下问题:(1)乳腺肿瘤大小参差不齐,将所有图像一并投入同一分割网络进行训练,部分小目标肿瘤容易被遗漏;(2)传统的基于全卷积网络的模型中默认特征图的每个通道的权重是相等的,从而丢失了不同通道之间的权重差异所隐藏的信息. ...
Model-free prostate cancer segmentation from dynamic contrast-enhanced MRI with recurrent convolutional networks: A feasibility study
1
2019
... 对于肿瘤图像的分割,目前已有很多自动的、半自动的方法,主要可以分为以下三类[14 ] :基于轮廓的方法[15 -17 ] 、基于模型的方法[18 , 19 ] 和基于数据的方法[20 -22 ] .基于轮廓的方法主要包括基于活动轮廓模型、主动轮廓模型、区域生长算法等方法;基于模型的方法主要包括基于模糊均值、聚类等传统机器学习的方法.这两类方法对图像质量敏感,精度相对较低且耗时较长.基于数据的方法主要是指基于一定规模数据集用于训练的深度学习的方法.随着深度学习算法的发展,越来越多基于数据的卷积神经网络的图像分割方法得以开发,这类方法虽然需要一定数量的标注数据用于网络训练,但可以得到相对较高的分割精度.已有很多方法被应用于基于DCE-MRI的乳腺肿瘤的自动分割,并且取得了不错的成效,如基于卷积神经网络(convolutional neural network,CNN)的分割方法[14 , 23 , 24 ] 、基于全卷积神经网络(fully convolutional networks,FCN)[25 ] 的方法,其中基于FCN的方法的分割性能尤为突出.Zhang等[26 , 27 ] 在2018年、2019年分别提出了基于FCN的乳腺肿瘤DCE-MR图像分割的两个模型,平均Dice相似性系数(Dice)均达到0.72;Adoui等[28 ] 提出分别基于SegNet和U-Net的两个FCN用于乳腺DCE-MR图像的肿瘤分割,但由于数据量有限,平均交并比(intersection over union,IoU)只分别达到0.688 8和0.741 6.但目前这些基于FCN的乳腺肿瘤磁共振图像的分割效果还不够理想,主要存在以下问题:(1)乳腺肿瘤大小参差不齐,将所有图像一并投入同一分割网络进行训练,部分小目标肿瘤容易被遗漏;(2)传统的基于全卷积网络的模型中默认特征图的每个通道的权重是相等的,从而丢失了不同通道之间的权重差异所隐藏的信息. ...
1
... 对于肿瘤图像的分割,目前已有很多自动的、半自动的方法,主要可以分为以下三类[14 ] :基于轮廓的方法[15 -17 ] 、基于模型的方法[18 , 19 ] 和基于数据的方法[20 -22 ] .基于轮廓的方法主要包括基于活动轮廓模型、主动轮廓模型、区域生长算法等方法;基于模型的方法主要包括基于模糊均值、聚类等传统机器学习的方法.这两类方法对图像质量敏感,精度相对较低且耗时较长.基于数据的方法主要是指基于一定规模数据集用于训练的深度学习的方法.随着深度学习算法的发展,越来越多基于数据的卷积神经网络的图像分割方法得以开发,这类方法虽然需要一定数量的标注数据用于网络训练,但可以得到相对较高的分割精度.已有很多方法被应用于基于DCE-MRI的乳腺肿瘤的自动分割,并且取得了不错的成效,如基于卷积神经网络(convolutional neural network,CNN)的分割方法[14 , 23 , 24 ] 、基于全卷积神经网络(fully convolutional networks,FCN)[25 ] 的方法,其中基于FCN的方法的分割性能尤为突出.Zhang等[26 , 27 ] 在2018年、2019年分别提出了基于FCN的乳腺肿瘤DCE-MR图像分割的两个模型,平均Dice相似性系数(Dice)均达到0.72;Adoui等[28 ] 提出分别基于SegNet和U-Net的两个FCN用于乳腺DCE-MR图像的肿瘤分割,但由于数据量有限,平均交并比(intersection over union,IoU)只分别达到0.688 8和0.741 6.但目前这些基于FCN的乳腺肿瘤磁共振图像的分割效果还不够理想,主要存在以下问题:(1)乳腺肿瘤大小参差不齐,将所有图像一并投入同一分割网络进行训练,部分小目标肿瘤容易被遗漏;(2)传统的基于全卷积网络的模型中默认特征图的每个通道的权重是相等的,从而丢失了不同通道之间的权重差异所隐藏的信息. ...
1
... 对于肿瘤图像的分割,目前已有很多自动的、半自动的方法,主要可以分为以下三类[14 ] :基于轮廓的方法[15 -17 ] 、基于模型的方法[18 , 19 ] 和基于数据的方法[20 -22 ] .基于轮廓的方法主要包括基于活动轮廓模型、主动轮廓模型、区域生长算法等方法;基于模型的方法主要包括基于模糊均值、聚类等传统机器学习的方法.这两类方法对图像质量敏感,精度相对较低且耗时较长.基于数据的方法主要是指基于一定规模数据集用于训练的深度学习的方法.随着深度学习算法的发展,越来越多基于数据的卷积神经网络的图像分割方法得以开发,这类方法虽然需要一定数量的标注数据用于网络训练,但可以得到相对较高的分割精度.已有很多方法被应用于基于DCE-MRI的乳腺肿瘤的自动分割,并且取得了不错的成效,如基于卷积神经网络(convolutional neural network,CNN)的分割方法[14 , 23 , 24 ] 、基于全卷积神经网络(fully convolutional networks,FCN)[25 ] 的方法,其中基于FCN的方法的分割性能尤为突出.Zhang等[26 , 27 ] 在2018年、2019年分别提出了基于FCN的乳腺肿瘤DCE-MR图像分割的两个模型,平均Dice相似性系数(Dice)均达到0.72;Adoui等[28 ] 提出分别基于SegNet和U-Net的两个FCN用于乳腺DCE-MR图像的肿瘤分割,但由于数据量有限,平均交并比(intersection over union,IoU)只分别达到0.688 8和0.741 6.但目前这些基于FCN的乳腺肿瘤磁共振图像的分割效果还不够理想,主要存在以下问题:(1)乳腺肿瘤大小参差不齐,将所有图像一并投入同一分割网络进行训练,部分小目标肿瘤容易被遗漏;(2)传统的基于全卷积网络的模型中默认特征图的每个通道的权重是相等的,从而丢失了不同通道之间的权重差异所隐藏的信息. ...
1
... 对于肿瘤图像的分割,目前已有很多自动的、半自动的方法,主要可以分为以下三类[14 ] :基于轮廓的方法[15 -17 ] 、基于模型的方法[18 , 19 ] 和基于数据的方法[20 -22 ] .基于轮廓的方法主要包括基于活动轮廓模型、主动轮廓模型、区域生长算法等方法;基于模型的方法主要包括基于模糊均值、聚类等传统机器学习的方法.这两类方法对图像质量敏感,精度相对较低且耗时较长.基于数据的方法主要是指基于一定规模数据集用于训练的深度学习的方法.随着深度学习算法的发展,越来越多基于数据的卷积神经网络的图像分割方法得以开发,这类方法虽然需要一定数量的标注数据用于网络训练,但可以得到相对较高的分割精度.已有很多方法被应用于基于DCE-MRI的乳腺肿瘤的自动分割,并且取得了不错的成效,如基于卷积神经网络(convolutional neural network,CNN)的分割方法[14 , 23 , 24 ] 、基于全卷积神经网络(fully convolutional networks,FCN)[25 ] 的方法,其中基于FCN的方法的分割性能尤为突出.Zhang等[26 , 27 ] 在2018年、2019年分别提出了基于FCN的乳腺肿瘤DCE-MR图像分割的两个模型,平均Dice相似性系数(Dice)均达到0.72;Adoui等[28 ] 提出分别基于SegNet和U-Net的两个FCN用于乳腺DCE-MR图像的肿瘤分割,但由于数据量有限,平均交并比(intersection over union,IoU)只分别达到0.688 8和0.741 6.但目前这些基于FCN的乳腺肿瘤磁共振图像的分割效果还不够理想,主要存在以下问题:(1)乳腺肿瘤大小参差不齐,将所有图像一并投入同一分割网络进行训练,部分小目标肿瘤容易被遗漏;(2)传统的基于全卷积网络的模型中默认特征图的每个通道的权重是相等的,从而丢失了不同通道之间的权重差异所隐藏的信息. ...
1
... 对于肿瘤图像的分割,目前已有很多自动的、半自动的方法,主要可以分为以下三类[14 ] :基于轮廓的方法[15 -17 ] 、基于模型的方法[18 , 19 ] 和基于数据的方法[20 -22 ] .基于轮廓的方法主要包括基于活动轮廓模型、主动轮廓模型、区域生长算法等方法;基于模型的方法主要包括基于模糊均值、聚类等传统机器学习的方法.这两类方法对图像质量敏感,精度相对较低且耗时较长.基于数据的方法主要是指基于一定规模数据集用于训练的深度学习的方法.随着深度学习算法的发展,越来越多基于数据的卷积神经网络的图像分割方法得以开发,这类方法虽然需要一定数量的标注数据用于网络训练,但可以得到相对较高的分割精度.已有很多方法被应用于基于DCE-MRI的乳腺肿瘤的自动分割,并且取得了不错的成效,如基于卷积神经网络(convolutional neural network,CNN)的分割方法[14 , 23 , 24 ] 、基于全卷积神经网络(fully convolutional networks,FCN)[25 ] 的方法,其中基于FCN的方法的分割性能尤为突出.Zhang等[26 , 27 ] 在2018年、2019年分别提出了基于FCN的乳腺肿瘤DCE-MR图像分割的两个模型,平均Dice相似性系数(Dice)均达到0.72;Adoui等[28 ] 提出分别基于SegNet和U-Net的两个FCN用于乳腺DCE-MR图像的肿瘤分割,但由于数据量有限,平均交并比(intersection over union,IoU)只分别达到0.688 8和0.741 6.但目前这些基于FCN的乳腺肿瘤磁共振图像的分割效果还不够理想,主要存在以下问题:(1)乳腺肿瘤大小参差不齐,将所有图像一并投入同一分割网络进行训练,部分小目标肿瘤容易被遗漏;(2)传统的基于全卷积网络的模型中默认特征图的每个通道的权重是相等的,从而丢失了不同通道之间的权重差异所隐藏的信息. ...
Hierarchical convolutional neural networks for segmentation of breast tumors in MRI with application to radiogenomics
1
2019
... 对于肿瘤图像的分割,目前已有很多自动的、半自动的方法,主要可以分为以下三类[14 ] :基于轮廓的方法[15 -17 ] 、基于模型的方法[18 , 19 ] 和基于数据的方法[20 -22 ] .基于轮廓的方法主要包括基于活动轮廓模型、主动轮廓模型、区域生长算法等方法;基于模型的方法主要包括基于模糊均值、聚类等传统机器学习的方法.这两类方法对图像质量敏感,精度相对较低且耗时较长.基于数据的方法主要是指基于一定规模数据集用于训练的深度学习的方法.随着深度学习算法的发展,越来越多基于数据的卷积神经网络的图像分割方法得以开发,这类方法虽然需要一定数量的标注数据用于网络训练,但可以得到相对较高的分割精度.已有很多方法被应用于基于DCE-MRI的乳腺肿瘤的自动分割,并且取得了不错的成效,如基于卷积神经网络(convolutional neural network,CNN)的分割方法[14 , 23 , 24 ] 、基于全卷积神经网络(fully convolutional networks,FCN)[25 ] 的方法,其中基于FCN的方法的分割性能尤为突出.Zhang等[26 , 27 ] 在2018年、2019年分别提出了基于FCN的乳腺肿瘤DCE-MR图像分割的两个模型,平均Dice相似性系数(Dice)均达到0.72;Adoui等[28 ] 提出分别基于SegNet和U-Net的两个FCN用于乳腺DCE-MR图像的肿瘤分割,但由于数据量有限,平均交并比(intersection over union,IoU)只分别达到0.688 8和0.741 6.但目前这些基于FCN的乳腺肿瘤磁共振图像的分割效果还不够理想,主要存在以下问题:(1)乳腺肿瘤大小参差不齐,将所有图像一并投入同一分割网络进行训练,部分小目标肿瘤容易被遗漏;(2)传统的基于全卷积网络的模型中默认特征图的每个通道的权重是相等的,从而丢失了不同通道之间的权重差异所隐藏的信息. ...
MRI breast tumor segmentation using different encoder and decoder CNN architectures
1
2019
... 对于肿瘤图像的分割,目前已有很多自动的、半自动的方法,主要可以分为以下三类[14 ] :基于轮廓的方法[15 -17 ] 、基于模型的方法[18 , 19 ] 和基于数据的方法[20 -22 ] .基于轮廓的方法主要包括基于活动轮廓模型、主动轮廓模型、区域生长算法等方法;基于模型的方法主要包括基于模糊均值、聚类等传统机器学习的方法.这两类方法对图像质量敏感,精度相对较低且耗时较长.基于数据的方法主要是指基于一定规模数据集用于训练的深度学习的方法.随着深度学习算法的发展,越来越多基于数据的卷积神经网络的图像分割方法得以开发,这类方法虽然需要一定数量的标注数据用于网络训练,但可以得到相对较高的分割精度.已有很多方法被应用于基于DCE-MRI的乳腺肿瘤的自动分割,并且取得了不错的成效,如基于卷积神经网络(convolutional neural network,CNN)的分割方法[14 , 23 , 24 ] 、基于全卷积神经网络(fully convolutional networks,FCN)[25 ] 的方法,其中基于FCN的方法的分割性能尤为突出.Zhang等[26 , 27 ] 在2018年、2019年分别提出了基于FCN的乳腺肿瘤DCE-MR图像分割的两个模型,平均Dice相似性系数(Dice)均达到0.72;Adoui等[28 ] 提出分别基于SegNet和U-Net的两个FCN用于乳腺DCE-MR图像的肿瘤分割,但由于数据量有限,平均交并比(intersection over union,IoU)只分别达到0.688 8和0.741 6.但目前这些基于FCN的乳腺肿瘤磁共振图像的分割效果还不够理想,主要存在以下问题:(1)乳腺肿瘤大小参差不齐,将所有图像一并投入同一分割网络进行训练,部分小目标肿瘤容易被遗漏;(2)传统的基于全卷积网络的模型中默认特征图的每个通道的权重是相等的,从而丢失了不同通道之间的权重差异所隐藏的信息. ...
Batch normalization: accelerating deep network training by reducing internal covariate shift
1
2015
... CBP5-Net网络的构架如图 2 所示.该网络由5个相同的block堆叠而成,每个block包含一个卷积(convolution,conv)层和一个最大池化(max-pooling)层,并引入批量正则化(batch normalization,BN)层[29 ] 以提高网络的收敛速度、避免过拟合.网络的最后是扁平(flatten)层和两个全连接(dense)层,将卷积层产生的特征图映射到类别数目.决策层使用Sigmoid函数,其余卷积层均使用线性整流(rectified linear unit,ReLU)函数进行激活.网络各层具体细节如表 1 所示. ...
1
2015
... U-Net[30 ] 是一种通过上采样和下采样的结构对图像进行多尺度特征提取的CNN,并且在上采样部分依然有很多特征通道,充分利用了上下文信息.同时U-Net还借鉴并优化了FCN中跳跃连接的思想,实现特征图的跨层连接. ...
RA-UNet: a hybrid deep attention-aware network to extract liver and tumor in CT scans
1
2020
... 为了充分利用图像特征、获取更多所关注目标的细节信息,本文借鉴U-Net的上采样、下采样结构及其连接方式,设计了RAU-Net模型对DCE-MRI乳腺肿瘤进行分割.RAU-Net网络构架如图 3(a) 所示.与之前研究者提出的将注意力机制用于上、下采样通道的跳跃连接处的RA-UNet网络模型[31 ] 不同的是,本研究设计的RAU-Net引入新的RA-block(RA为Residual和Attention的首字母组合,即该模块是残差结构和注意力机制的融合)模块用于上、下采样通道中,在获取通道之间权重差异信息的同时加深了U-Net的网络深度,有效提高了模型的学习能力.RA_block模块结构如图 3(b) 所示:由两个卷积层、全局平均池化(global average pooling,GAP)层、两个分别由ReLU函数和Sigmoid函数激活的全连接层构成主要路径,同时添加一个恒等映射连接[32 ] 将前层的特征直接传递到下一模块.一方面该模块可以获取不同通道之间深层语义特征的依赖关系,对不同重要性的通道赋予不同权重[33 ] ,多维度的信息提高了模型的学习性能;另一方面该模块通过添加恒等映射连接解决了由于网络深度增加引起的梯度消失现象.同时,还加入Dropout[34 ] 技术以避免过拟合.RAU-Net网络及RA_block模块的具体细节如表 2 、表 3 所示,网络通过max-pooling层实现下采样,上卷积(up-convolution,up-conv)层则完成上采样操作. ...
1
... 为了充分利用图像特征、获取更多所关注目标的细节信息,本文借鉴U-Net的上采样、下采样结构及其连接方式,设计了RAU-Net模型对DCE-MRI乳腺肿瘤进行分割.RAU-Net网络构架如图 3(a) 所示.与之前研究者提出的将注意力机制用于上、下采样通道的跳跃连接处的RA-UNet网络模型[31 ] 不同的是,本研究设计的RAU-Net引入新的RA-block(RA为Residual和Attention的首字母组合,即该模块是残差结构和注意力机制的融合)模块用于上、下采样通道中,在获取通道之间权重差异信息的同时加深了U-Net的网络深度,有效提高了模型的学习能力.RA_block模块结构如图 3(b) 所示:由两个卷积层、全局平均池化(global average pooling,GAP)层、两个分别由ReLU函数和Sigmoid函数激活的全连接层构成主要路径,同时添加一个恒等映射连接[32 ] 将前层的特征直接传递到下一模块.一方面该模块可以获取不同通道之间深层语义特征的依赖关系,对不同重要性的通道赋予不同权重[33 ] ,多维度的信息提高了模型的学习性能;另一方面该模块通过添加恒等映射连接解决了由于网络深度增加引起的梯度消失现象.同时,还加入Dropout[34 ] 技术以避免过拟合.RAU-Net网络及RA_block模块的具体细节如表 2 、表 3 所示,网络通过max-pooling层实现下采样,上卷积(up-convolution,up-conv)层则完成上采样操作. ...
Squeeze-and-excitation networks
1
2017
... 为了充分利用图像特征、获取更多所关注目标的细节信息,本文借鉴U-Net的上采样、下采样结构及其连接方式,设计了RAU-Net模型对DCE-MRI乳腺肿瘤进行分割.RAU-Net网络构架如图 3(a) 所示.与之前研究者提出的将注意力机制用于上、下采样通道的跳跃连接处的RA-UNet网络模型[31 ] 不同的是,本研究设计的RAU-Net引入新的RA-block(RA为Residual和Attention的首字母组合,即该模块是残差结构和注意力机制的融合)模块用于上、下采样通道中,在获取通道之间权重差异信息的同时加深了U-Net的网络深度,有效提高了模型的学习能力.RA_block模块结构如图 3(b) 所示:由两个卷积层、全局平均池化(global average pooling,GAP)层、两个分别由ReLU函数和Sigmoid函数激活的全连接层构成主要路径,同时添加一个恒等映射连接[32 ] 将前层的特征直接传递到下一模块.一方面该模块可以获取不同通道之间深层语义特征的依赖关系,对不同重要性的通道赋予不同权重[33 ] ,多维度的信息提高了模型的学习性能;另一方面该模块通过添加恒等映射连接解决了由于网络深度增加引起的梯度消失现象.同时,还加入Dropout[34 ] 技术以避免过拟合.RAU-Net网络及RA_block模块的具体细节如表 2 、表 3 所示,网络通过max-pooling层实现下采样,上卷积(up-convolution,up-conv)层则完成上采样操作. ...
Dropout: a simple way to prevent neural networks from overfitting
1
2014
... 为了充分利用图像特征、获取更多所关注目标的细节信息,本文借鉴U-Net的上采样、下采样结构及其连接方式,设计了RAU-Net模型对DCE-MRI乳腺肿瘤进行分割.RAU-Net网络构架如图 3(a) 所示.与之前研究者提出的将注意力机制用于上、下采样通道的跳跃连接处的RA-UNet网络模型[31 ] 不同的是,本研究设计的RAU-Net引入新的RA-block(RA为Residual和Attention的首字母组合,即该模块是残差结构和注意力机制的融合)模块用于上、下采样通道中,在获取通道之间权重差异信息的同时加深了U-Net的网络深度,有效提高了模型的学习能力.RA_block模块结构如图 3(b) 所示:由两个卷积层、全局平均池化(global average pooling,GAP)层、两个分别由ReLU函数和Sigmoid函数激活的全连接层构成主要路径,同时添加一个恒等映射连接[32 ] 将前层的特征直接传递到下一模块.一方面该模块可以获取不同通道之间深层语义特征的依赖关系,对不同重要性的通道赋予不同权重[33 ] ,多维度的信息提高了模型的学习性能;另一方面该模块通过添加恒等映射连接解决了由于网络深度增加引起的梯度消失现象.同时,还加入Dropout[34 ] 技术以避免过拟合.RAU-Net网络及RA_block模块的具体细节如表 2 、表 3 所示,网络通过max-pooling层实现下采样,上卷积(up-convolution,up-conv)层则完成上采样操作. ...
SegNet: a deep convolutional encoder-decoder architecture for image segmentation
1
2017
... 训练大肿瘤RAU-Net分割模型时,将大肿瘤训练集和对应标签送入网络,同样采用Adam进行梯度下降,学习率为1e-6 ,卷积层使用He初始化方法初始化,训练集和验证集的批尺寸分别为32和16,迭代次数为300并设置早停.我们以加权二元交叉熵损失函数作为损失层函数,并通过中值频率平衡(median frequency balancing)[35 ] 计算出两个类别的权重值:前景权值取0.7、背景权值取0.3. ...
1
... 为进一步验证本文方法的有效性,我们对测试集做了多个对比实验.表 4 记录了测试集中68张图像采用不同方法得到的各项评价指标.RAU-Net在多个方面都优于U-Net:分割结果的Dice系数、敏感度、IoU分别增加了约2%、1%、2%,且两个模型指标之间的p 值分别为0.042 2,0.041 8和0.035 0,均小于0.05,具有统计学意义,特异性指标则不相上下;RAU-Net也要优于在U-Net基础上仅添加残差结构的ResU-Net;对比U-Net++[36 ] 也具有一定优势:Dice系数和敏感度分别提高了约0.6%、3.6%.以图 1 所示方式将RAU-Net与CBP5-Net连接后,模型的性能又有了进一步提升,Dice系数、IoU分别提高了1.3%、1.4%,敏感度和特异性则与单一网络时持平.从该表中也可以看出,在使用基于连通域分析的后处理操作后,各方法的分割结果Dice系数均有所提升.此外,表格最后一列为各模型预测测试集中68张图像的耗时,可见RAU-Net使用RA模块替代U-Net的部分卷积层,利用更少的特征通道获得了更好的分割性能,同时还具有较高的预测效率;而CBP5-Net是个极轻量化的网络,RAU-Net与其连接后,整个模型依然保持较高的效率. ...