引言
乳腺密度是表征乳腺癌风险的因素之一[1],可通过乳房内纤维腺体组织(fibroglandular tissue,FGT)占乳房组织的比率来评估.乳腺动态增强磁共振成像(dynamic contrast enhanced magnetic resonance imaging,DCE-MRI)是乳房组织的三维成像方法,其提供了脂肪和腺体的高对比度信息,是乳腺密度计算的理想选择[2].评估乳腺密度的关键步骤是乳房和腺体的精确分割.乳房分割包括空气-乳房边界检测和胸壁线检测,后者由于胸壁、胸肌和乳房组织周围无明显的边界而成为乳房分割的主要难点.腺体分割的主要难点是乳腺DCE-MR图像中乳房内部组织的强度分布不均匀.
上述提到的传统方法和深度学习方法通常设有预处理和后处理操作,且针对不同患者和不同扫描设备需对模型参数进行手动调整,因此模型泛化性能较低,例如在本文前期研究[21]中提出的方法,针对不同的数据集,需要手动进行繁琐的参数调整和预处理,不利于临床应用.另外,目前多数方法是基于非脂肪抑制乳腺MR图像实现组织分割,而临床乳腺DCE-MRI扫描过程中,通常采用脂肪抑制成像方式获取造影前、后图像序列.脂肪抑制图像噪声水平较高、对比度较低、脂肪抑制不均匀,因此在脂肪抑制MR图像中分割乳房和腺体更具挑战性和实际意义.
本文在前期研究[21]的基础上,针对模型泛化能力较低和脂肪抑制乳腺DCE-MR图像分割更具挑战性两个问题,提出了基于nnU-Net的乳腺DCE-MR图像分割框架,它可根据图像的分辨率、体素间距等特征自动进行重采样、数据扩增、网络结构及参数的动态调整,并针对脂肪抑制乳腺DCE-MR图像噪声水平较高、对比度较低对比和不均匀的脂肪抑制等特点改进了nnU-Net的自适应策略,增加了对比度增强和噪声抑制的预处理,以提高脂肪抑制图像分割的质量.本文以具有不同图像参数和乳腺密度等级的三维脂肪抑制乳腺DCE-MR图像为研究对象,训练该模型并评估了分割性能.
1 乳房和腺体的自动分割
1.1 整体框架
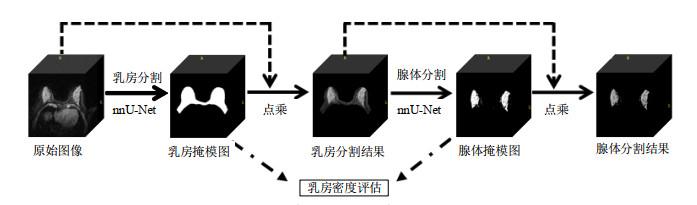
本文提出的乳腺DCE-MR图像分割框架如图 1所示,采用两个nnU-Net模型级联方式实现乳房和腺体的自动分割.首先,乳房分割网络以包含双侧乳房的脂肪抑制乳腺DCE-MR图像为输入,输出为分割后的乳房二值掩模图像;然后将乳房二值掩模图像与原始图像做矩阵点乘运算得到乳房分割结果;再将乳房分割结果作为腺体分割网络的输入,输出是分割后的腺体二值掩模图像;最后将腺体二值掩模图像与乳房分割结果做矩阵点乘运算得到腺体分割结果.
图1
图1
脂肪抑制乳腺DCE-MR图像中乳房和腺体(FGT)的分割框架
Fig.1
The framework for breast and FGT segmentation in fat-suppressed breast DCE-MR images
本文还基于乳房分割和腺体分割的结果,计算了双侧乳房体积和双侧腺体体积,进而计算乳腺密度.计算方法将在3.2节详细说明.
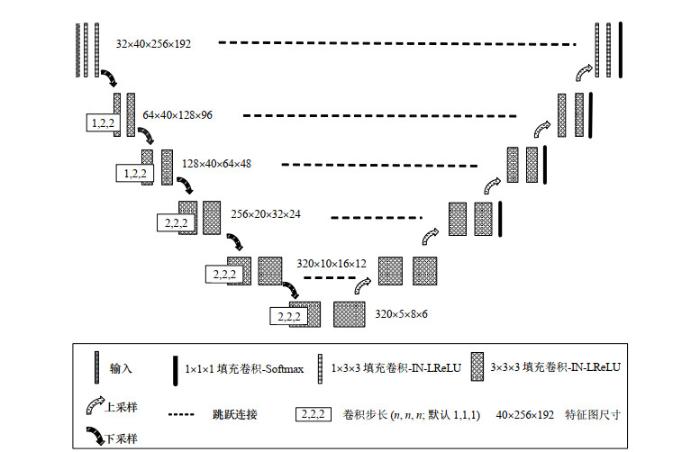
1.2 网络结构
本文分割模型使用的三维网络结构如图 2所示,由编码路径、解码路径和跳跃连接三部分组成.
图2
编码路径是下采样过程,包含若干个块(block),每个block由两个3×3×3的卷积层和一个2×2×2的最大池化层(下采样)组成,每个卷积层后连接一个归一化(normalization)层和一个修正线性单元(rectified linear unit,ReLU);解码路径是上采样过程,包含若干个块(block),每个block与编码阶段相同层次的block组成相似,上采样通过2×2×2的反卷积实现.图 2中两条路径之间的虚线表示将上采样后的特征图与对应下采样的特征图拼接结合,避免细节信息丢失,提高分割精度.解码路径的最后一层,通过1×1×1的卷积将特征图映射到整个网络的输出层,输出图像为输入图像每个体素点(voxel)的分类结果,即背景体素或前景体素.
为适于脂肪抑制乳腺DCE-MR图像处理,本文对U-Net[13]结构进行了改进,如图 2图例中的卷积层所示,包括:1)使用填充卷积保证输出图像和输入图像尺寸相同.卷积运算中由于图像边缘处的体素点不会位于卷积核中心,卷积核也无法扩展到边缘以外,因此图像边缘处的信息会被遗漏,导致输入图像和输出图像大小不一致.填充卷积是对输入图像进行边界填充,以增加图像矩阵的大小.通常用“0”来进行填充.当卷积核与图像运算时,能延伸到边缘以外的伪体素,从而使输出图像和输入图像大小相同.2)由于GPU内存限制训练过程中批处理(batch)的大小,因此使用实例归一化(instance normalization,IN)代替使用批归一化(batch normalization).3)由于乳腺DCE-MR图像中背景体素(体素值近似为0)较多,经预处理后可能变为负值,在基于梯度的学习过程中,如果使用ReLU,神经元的梯度参数一直为0,在以后的训练过程中将不会被激活,导致训练缓慢.本文使用带泄露修正线性单元(Leaky ReLU,LReLU)代替ReLU,输入为负值时,神经元梯度参数不为0(而是一个很小的数),能够使神经元一直被激活,从而加快学习速度.
1.3 自适应分割方案
本文基于nnU-Net[21]的思路设计自适应分割策略,使分割模型对具有不同成像参数的图像自动进行预处理、网络参数调整和数据扩增,无需人工干预.(1)首先根据图像尺寸(分辨率)、体素间距等特征对图像自动进行重采样,设定重采样后的图像尺寸为数据集中所有图像尺寸的中值.对于各向异性的图像,由于各方向的体素间距不相同,因此先对图像分辨率较高的方向进行下采样,使其分辨率与其它方向一致,之后再按照各向同性图像的处理方法,同时对所有方向进行重采样.本文对原始图像和分割金标准分别采用三阶样条插值和最近邻插值实现重采样.针对脂肪抑制图像相对较高的噪声水平、较低的强度对比和不均匀的脂肪抑制等特点,本文相比nnU-Net增加了限制对比度自适应直方图均衡化(contrast limited adaptive histogram equalization,CLAHE)[25]的预处理操作,将图像分块,以块为单位进行直方图均衡化,再利用线性插值确定块间体素值,同时实现对比度增强和噪声抑制,提高分割的准确性;(2)经过预处理后,输入网络的图像尺寸统一为原始数据集中图像的中值尺寸,再根据GPU内存大小确定训练过程的批尺寸(batch size),优先满足输入尺寸尽可能大以保证分割结果的准确性,其次满足批尺寸最小为2以防止因样本过少引起的梯度噪声.为获取具有足够信息量的特征,本文设定特征图的尺寸最小为4×4×4;为限制模型大小,设定特征数目上限为320.下采样次数根据输入图像的尺寸及特征图的大小确定,本文的下采样次数为5;(3)在网络训练阶段,采用镜像变换、尺度伸缩变换、旋转平移等数据扩增操作,扩大训练样本容量,防止模型过拟合.
1.4 损失函数
本文使用Dice损失和交叉熵损失之和作为分割误差的损失函数.Dice损失可直接对分割相似度进行优化,且能够解决训练样本类别不平衡问题,但其梯度形式复杂,反向传播过程易出现梯度变化剧烈,导致模型难以收敛.交叉熵损失在分割任务中能够衡量金标准和分割结果之间的差异,交叉熵值越小,分割效果越好,但不能解决类别不平衡问题.将二者结合,可以有效提高训练过程的稳定性和分割的精准性.本文Dice损失函数和交叉熵损失函数如(1)式和(2)式所示:
其中,K表示类别数目(本文K为2,代表前景和背景),I表示每个batch中的体素集,u表示Softmax输出概率值,v表示金标准的one-hot编码值.分割误差的总损失函数为:
2 实验部分
2.1 数据集
本文系回顾性研究,数据集由复旦大学附属肿瘤医院的临床医学影像存储与传输系统(picture archiving and communication system,PACS)随机收集的80例三维T1加权的脂肪抑制乳腺DCE-MR图像组成,包括低、中、高风险病例,以及具有良性和恶性肿瘤的病例,所有数据使用Aurora 1.5 Tesla MR扫描仪获得.本文根据采集参数将图像分为两组:组别1包含42例,分辨率为512×512×160,层厚为1.125 mm,扫描160层,扫描时间为3 min;组别2包含38例,分辨率为512×512×108,层厚为1.481 mm,扫描108层,扫描时间为2 min.x、y方向的像素尺寸为0.701 3 mm,视野(field of view,FOV)和翻转角(flip angle)分别为360 mm和90˚.每层图像的尺寸是512像素×512像素,所有数据的图像强度均被预处理为[0,255].
放射科专家根据美国放射学会乳房影像报告和数据系统(American college of radiology breast imaging reporting and data system,ACR BI-RADS)评估了每例乳房的密度(Ⅰ-脂肪: < 25%;Ⅱ-分散:25% ~ 50%;Ⅲ-非均匀致密:50% ~ 75%;Ⅳ-致密: > 75%),四个等级分别有11、41、22和6例.
同一病例的DCE-MR图像包括注射造影剂前和注射造影剂后的多个时间段的图像,注射造影剂前、后图像均采用脂肪抑制成像序列获得.本文对增强前和增强后的图像进行了配准,配准结果表明,由于乳腺磁共振扫描时线圈固定,增强前后图像的位移不明显.因此除了灰度发生变化外,增强前后组织的形态结构并未有明显变化.同时使用增强前后的图像与只使用增强前的图像,对乳房分割结果影响不大.同时,由于本文乳房分割和腺体分割的目的是计算乳腺密度,增强后的图像中腺体组织被增强,会影响图像中腺体的强度,进而导致乳腺密度计算的不准确,因此本文仅使用增强前的图像.
2.2 实验实施
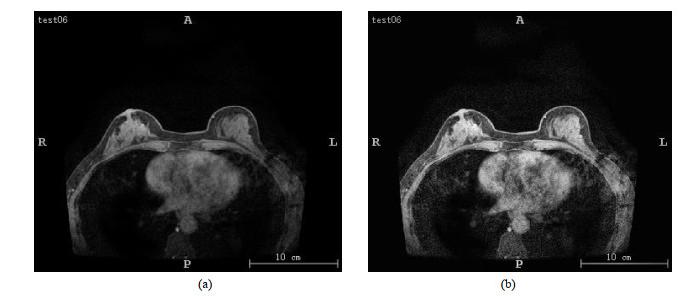
本文将数据集按照1:1划分为训练(验证)集40例(包括21例160层的样本和19例108层的样本)和测试集40例(包括21例160层的样本和19例108层的样本),并保持具有不同成像参数和乳腺密度等级的图像均衡分布.经过重采样和CLAHE预处理后,图像对比度增强,噪声点减少,如图 3所示.本文乳房分割和腺体分割网络的图像输入尺寸分别为40×256×192和64×96×256.根据该输入大小和GPU内存(12 GB),批尺寸设置为2.为防止过拟合,本文初始学习率设为0.01,训练过程中随着迭代的继续采用指数衰减(exponential decay)逐步减小学习率,使得模型更加稳定.采用正态分布随机初始化权重策略.利用带有Nesterov Momentum(参数设为0.9)的随机梯度下降方法对损失函数进行优化.本文将训练过程的时期(Epoch)定义为250个batch上的迭代优化,最大值设为1 000.网络训练过程中,采用五折交叉验证(five-fold cross validation),经过数据扩增后,训练(验证)集的样本量扩增至200例,将扩增后的200例样本随机划分五等份,每次训练取其中一份作为验证集用来确定模型的超参数.五个模型训练完成后,随机选择一个模型用于测试.
图3
图3
CLAHE处理前后图像对比
Fig.3
Comparison between the images (a) before and (b) after CLAHE processing
本文实验在配置有4个NVIDIA GeForce GTX Titan V(12GB,Volta架构)GPU、Ubuntu 16.04操作系统的虚拟环境中进行,开发工具为Python和PyTorch框架.本文在nnU-Net源代码(可从https://github.com/MIC-DKFZ/nnUNet开源获取)基础上进行了修改,以更好适应运行环境和实验数据.
2.3 评价方法
为准确客观地评价实验结果,本文使用Dice相似系数(Dice similarity coefficient,DSC)、准确度(accuracy,Acc)、灵敏度(sensitivity,Sen)、特异度(specificity,Spec)和平均对称表面距离(average symmetric surface distance,ASD)作为评价指标.上述指标计算公式如下:
其中TP、TN、FP和FN分别表示真阳性、真阴性、假阳性和假阴性,X表示金标准表面,Y表示分割结果表面,
此外,使用皮尔逊相关系数(Pearson correlation coefficient)评估分割结果与金标准之间的相关性;根据乳腺密度进一步评价腺体分割的性能.
同时,放射科专家对分割结果的有效性进行主观评价,使用三个分数表示分割质量:“3”表示良好,几乎不需要手动修订;“2”表示可接受,需要进行较小的手动修正;“1”表示无法在临床实践中使用.
3 结果与讨论
3.1 分割结果的指标评价
表 1给出了使用本文方法自动分割的结果相对于金标准的五种评价指标,以平均值±标准差的形式表示.使用本文方法得到的乳房分割和腺体分割的DSC分别为0.969±0.007和0.893±0.054,ASD为0.181±0.032 mm和0.240±0.021 mm.通常情况下,用于医学图像的深度学习方法容易产生较高的假阳性率(false positive rate,FPR),从而导致分割过度[27].但如表 1所示,对于乳房和腺体分割,使用本文提出的基于nnU-Net的模型分割结果具有高敏感性和特异性,表明该模型具有较高的真阳性率(true positive rate,TPR)和较低的FPR,可以有效避免过度分割.
表1 分割性能评价
Table 1
| DSC | Acc | Sen | Spec | ASD/mm | |
| 乳房分割 | 0.969±0.007 | 0.995±0.006 | 0.961±0.027 | 0.994±0.002 | 0.181±0.032 |
| 腺体分割 | 0.893±0.054 | 0.997±0.003 | 0.926±0.017 | 0.998±0.002 | 0.240±0.021 |
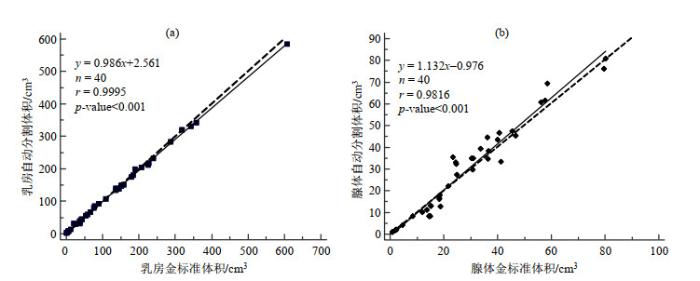
图4
图4
使用本文提出的模型得到的自动分割体积与金标准体积的相关性. (a) 乳房;(b) 腺体
Fig.4
The correlation between the automatic segmentation volume obtained using the proposed model and the ground truth volume. (a) Breast; (b) FGT
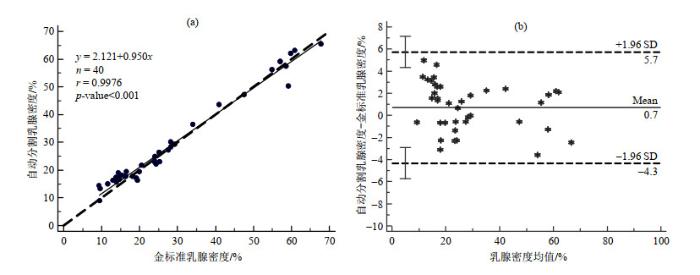
体素数是分割得到的三维掩膜体值为1的体素数量.本文对三维MR图像的体素间距(voxel spacing)进行了各向同性处理,体素间距调整为1×1×1,因此x、y、z三个方向的分辨率均为0.701 3 mm.为便于在图中展示,实际体积的单位是立方厘米(cm3).图 4中,乳房自动分割和腺体自动分割的物理实际体积与金标准的相关系数r分别为0.999 5(p-value < 0.001)和0.981 6(p-value < 0.001),表明使用本文提出的模型得到的自动分割结果与金标准有很强的一致性.
3.2 乳腺密度评估
乳房分割和腺体分割的临床应用之一是计算乳腺密度.基于3.1节中得到的乳房体积(记为|Breast|)和腺体体积(记为
图 5(a)展示了分割结果和金标准中之间乳腺密度计算结果的相关性,其中x轴表示基于金标准的乳腺密度,y轴表示基于自动分割结果的乳腺密度,两者的相关系数r为0.997 6(p-value < 0.001),进一步表明使用本文模型得到的自动分割结果的准确性.图 5(b)展示了使用本文基于nnU-net改进方法得到的分割结果与金标准的一致性,其中横轴为两种方法所得结果的均值,纵轴为两种方法所得结果的差值,Mean表示差值的平均值,SD表示差值的标准差,(Mean ± 1.96 SD)为一致性界限(limits of agreement,LOA)(图中虚线).可以看出,使用本文分割模型得到的乳腺密度与金标准的差值均值为0.7%,普遍高于手动分割结果;一致性界限为[-4.3%,5.7%],重复性系数(coefficient of repeatability,CR)值为5,绝大部分差值在一致性界限内.说明使用本文方法的分割结果与手动分割结果基本一致.
图5
图5
使用本文提出的模型得到的乳腺密度与金标准乳腺密度的(a)相关性和(b) Bland-Altman一致性
Fig.5
(a) The correlation and (b) Bland-Altman plot between the automatic segmentation breast densities obtained using the proposed model and the ground truth breast density
3.3 图像参数和乳腺密度对分割性能的影响
表2 不同图像参数组别的乳房和腺体分割的DSC比较
Table 2
| 乳房分割 | 腺体分割 | |
| 组1 | 0.973±0.011 | 0.903±0.055 |
| 组2 | 0.977±0.026 | 0.946±0.042 |
表3 不同乳腺密度类别的乳房和腺体分割的DSC比较
Table 3
| 乳房分割 | 腺体分割 | |
| Ⅰ类 | 0.989±0.004 | 0.906±0.048 |
| Ⅱ类 | 0.980±0.008 | 0.951±0.021 |
| Ⅲ类 | 0.941±0.029 | 0.917±0.031 |
| Ⅳ类 | 0.956±0.010 | 0.868±0.037 |
图6
图6
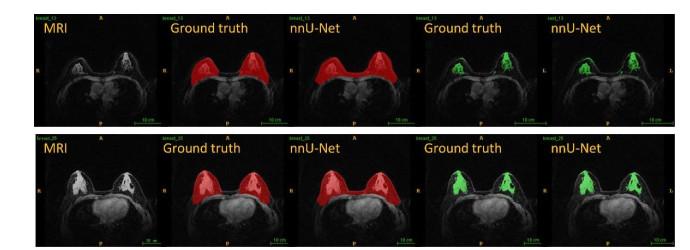
不同图像参数组别的乳腺DCE-MR图像分割结果示例.上、下两行分别为组1的代表性样本和组2的代表性样本.从左至右,分别为原始图像、乳房分割的金标准、乳房分割结果、腺体(FGT)分割的金标准和腺体分割结果
Fig.6
Segmentation examples of two groups with different breast DCE-MR imaging parameters. The top and bottom lines are Group 1 and Group 2. From left to right: the original image, the ground truth of whole breast, the segmentation mask of whole breast, the ground truth of FGT, and the segmentation mask of FGT
图7
图7
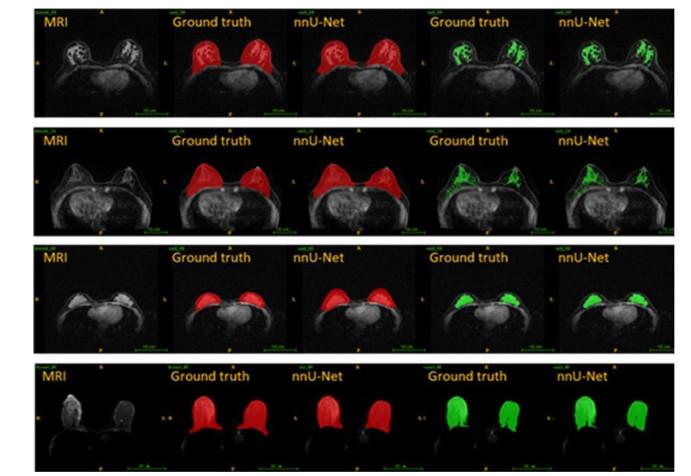
不同乳腺密度类别的乳腺DCE-MR图像分割结果示例.从上至下,依次为乳腺密度类别Ⅰ、类别Ⅱ、类别Ⅲ和类别Ⅳ的代表性样本(Ⅰ-脂肪: < 25%;Ⅱ-分散:25% ~ 50%;Ⅲ-非均匀致密:50% ~ 75%;Ⅳ-致密: > 75%).从左至右,分别为原始图像、乳房分割的金标准、乳房分割结果、腺体(FGT)分割的金标准和腺体分割结果
Fig.7
Segmentation examples of four groups with different breast density ratings. From top down: Category I, Category Ⅱ, Category Ⅲ and Category Ⅳ (Ⅰ - fatty: < 25%; Ⅱ - scattered: 25% ~ 50%; Ⅲ - heterogeneously dense: 50% ~ 75%; Ⅳ - dense: > 75%). From left to right: the original image, the ground truth of whole breast, the segmentation mask of whole breast, the ground truth of FGT, and the segmentation mask of FGT
3.4 模型泛化性能的评估
本文在模型训练过程中使用了五折交叉验证来提高模型的泛化性能.结果(表 4)显示,在五折交叉验证中,乳房分割结果的DSC值介于0.970和0.982之间,平均值±标准差为0.976±0.027,腺体分割结果的DSC介于0.941和0.946之间,平均值±标准差为0.944±0.047.五个模型在对应的验证集上都取得了准确、稳定的DSC.
表4 交叉验证中的分割性能评价(以DSC为例)
Table 4
| 乳房分割 | 腺体分割 | |||
| 验证集 | Fold 1 | 0.970±0.049 | 0.941±0.061 | |
| Fold 2 | 0.982±0.014 | 0.945±0.046 | ||
| Fold 3 | 0.971±0.044 | 0.942±0.047 | ||
| Fold 4 | 0.982±0.016 | 0.946±0.052 | ||
| Fold 5 | 0.975±0.012 | 0.942±0.031 | ||
| 平均值±标准差 | 0.976±0.027 | 0.944±0.047 | ||
| 测试集 | 0.969±0.007 | 0.893±0.054 | ||
图8
图8
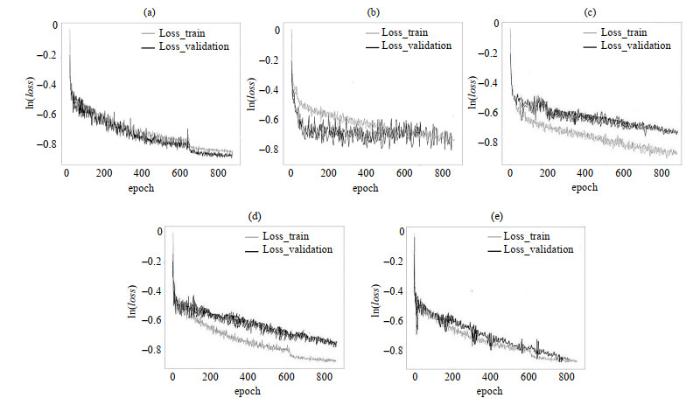
乳房分割模型在训练(验证)集上的Loss曲线.(a) ~ (e)分别为五折交叉验证中五个模型的Loss曲线
Fig.8
Loss curves of the breast segmentation model on the training (validation) set. Figures (a) ~ (e) represent the loss curves of each model of five-fold cross-validation, respectively
图9
图9
腺体分割模型在训练(验证)集上的Loss曲线.图(a) ~ (e)分别为五折交叉验证中五个模型的Loss曲线.
Fig.9
Loss curves of the FGT segmentation model on the training (validation) set. Figures (a) ~ (e) represent the loss curves of each model of five-fold cross-validation, respectively
为进一步验证模型的泛化性能,将本文分割方法应用于从复旦大学附属肿瘤医院收集的另一个独立数据集,包含40例使用Siemens 1.5 T MR扫描仪获得的病例,分辨率为512×512×104(30例)或384×384×80(10例),层厚为1.5 mm或2.2 mm,x、y方向的像素尺寸为0.585 9 mm或0.937 5 mm,放射科专家主观评价组织分割也取得了良好的效果.但是,本研究中使用的所有DCE-MR图像来源于同一家医院,未来将在多中心和多设备的数据集上验证本文提出的分割框架,以获得更可靠的模型.
3.5 与其他方法分割结果的比较
如引言所述,由于针对脂肪抑制乳腺DCE-MR图像的分割模型较少,难以提供足够的细节信息来对不同模型自动分割的准确性和效率进行客观的比较.因此在表 5中,我们仅就DSC与已有文献进行比较.结果显示本文在40例脂肪抑制乳腺DCE-MR图像上取得了相对更准确的结果,尤其是对于腺体分割,DSC为0.89 ± 0.05,显著高于其他方法(p-value = 0.011 < 0.05),表明本文基于nnU-Net的自动分割方法比传统方法和需手动调节参数的深度学习方法更具有优势.
表5 使用本文提出的模型自动分割的DSC与文献值的对比
Table 5
已公开发表的多数研究在处理不同的数据集时需要对参数进行微调.本文基于nnU-Net的分割方法通过采用具有鲁棒性的训练策略以及具有自适应性的预处理和后处理操作,避免了繁琐而复杂的参数调整,该分割框架可以直接使用,也可以集成到临床工作流程中.
3.6 运行时间
本实验训练模型时,平均每个epoch用时400 s.测试过程中,乳房分割平均需要20 s/例,FGT分割平均需要15 s/例.具体而言,在160层的MR扫描中平均分别需要38 s/例和18 s/例来分割乳房和FGT,在108层的MR扫描中分别为25 s/例和12 s/例.
4 结论
本文研究了基于nnU-Net的乳房分割和腺体分割模型,在具有不同参数的三维脂肪抑制乳腺DCE-MR图像上对模型进行了训练(验证)和测试,结果表明该模型可以稳定地实现乳房和腺体的准确分割,与传统方法相比,无需手动调节网络参数,在处理脂肪抑制乳腺DCE-MR图像方面具有广阔的潜力.下一步研究考虑进行模型压缩以缩短分割时间,并在多中心数据集上应用该分割框架,进一步提高模型的泛化性能.
霍璐和姜娈得到了上海联影医疗科技股份有限公司的部分支持.
参考文献
Mammographic density and the risk and detection of breast cancer
[J].
Magnetic resonance imaging for secondary assessment of breast density in a high-risk cohort
[J].DOI:10.1016/j.mri.2009.05.040 [本文引用: 1]
Automated chest wall line detection for whole-breast segmentation in sagittal breast MR images
[J].DOI:10.1118/1.4793255 [本文引用: 1]
Development of a quantitative method for analysis of breast density based on three-dimensional automated segmentation of breast in 3-D MR images
[J].DOI:10.1118/1.3002306 [本文引用: 1]
Breast segmentation and density estimation in breast MRI: A fully automatic framework
[J].DOI:10.1109/JBHI.2014.2311163 [本文引用: 3]
A level set based framework for quantitative evaluation of breast tissue density from MRI data
[J].DOI:10.1371/journal.pone.0112709 [本文引用: 3]
Automated fibroglandular tissue segmentation and volumetric density estimation in breast MRI using an atlas-aided fuzzy C-means method
[J].DOI:10.1118/1.4829496 [本文引用: 1]
Structured learning for 3-D perivascular space segmentation using vascular features
[J].
Classification of Alzheimer's disease patients based on magnetic resonance images and an improved UNet++ model
[J].
基于磁共振图像和改进的UNet++模型区分阿尔茨海默症患者和健康人群
[J].
Unsupervised deep learning applied to breast density segmentation and mammographic risk scoring
[J].
Automatic segmentation of right ventricle in cine cardiac magnetic resonance image based on a dense and multi-scale u-net method
[J].
基于密集多尺度U-net网络的电影心脏磁共振图像右心室自动分割
[J].
A U-Net network-based rapid construction of knee models for specific absorption rate estimation
[J].
用于SAR估计的基于U-Net网络的快速膝关节模型重建
[J].
Automatic breast and fibroglandular tissue segmentation in breast MRI using deep learning by a fully-convolutional residual neural network U-Net
[J].DOI:10.1016/j.acra.2019.01.012 [本文引用: 4]
Using deep learning to segment breast and fibroglandular tissue in MRI volumes
[J].
Fully automated segmentation of whole breast using dynamic programming in dynamic contrast enhanced MR images
[J].DOI:10.1002/mp.12254 [本文引用: 3]
The state of the art in kidney and kidney tumor segmentation in contrast-enhanced CT imaging: Results of the KiTS19 challenge
[J].DOI:10.1016/j.media.2020.101821 [本文引用: 1]
User-guided 3D active contour segmentation of anatomical structures: Significantly improved efficiency and reliability
[J].DOI:10.1016/j.neuroimage.2006.01.015 [本文引用: 1]
Medical image segmentation using deep learning
[J].