Automatic segmentation of breast and fibroglandular tissues in DCE-MR images based on nnU-Net
1
2021
... 胰腺囊性肿瘤自动分割是一种利用计算机图像处理和人工智能技术的创新方法,通过快速、自动地提取胰腺囊性肿瘤区域来提高临床工作效率,这对胰腺囊性肿瘤的诊断和评估具有至关重要的意义.胰腺具有复杂的形态结构和密集的周围组织,传统的手动分割方法费时费力,且存在主观性和人为误差的风险.尽管自动分割技术在医学领域取得了许多显著进展[1,2],但是由于胰腺与邻近的器官和血管之间可能存在相似的密度和纹理特征,其自动分割仍然面临一些独特的难题.此外,胰腺囊性肿瘤常常导致器官形态和密度的变化,进一步增加了自动分割的难度. ...
基于nnU-Net的乳腺DCE-MR图像中乳房和腺体自动分割
1
2021
... 胰腺囊性肿瘤自动分割是一种利用计算机图像处理和人工智能技术的创新方法,通过快速、自动地提取胰腺囊性肿瘤区域来提高临床工作效率,这对胰腺囊性肿瘤的诊断和评估具有至关重要的意义.胰腺具有复杂的形态结构和密集的周围组织,传统的手动分割方法费时费力,且存在主观性和人为误差的风险.尽管自动分割技术在医学领域取得了许多显著进展[1,2],但是由于胰腺与邻近的器官和血管之间可能存在相似的密度和纹理特征,其自动分割仍然面临一些独特的难题.此外,胰腺囊性肿瘤常常导致器官形态和密度的变化,进一步增加了自动分割的难度. ...
An improved level set algorithm for prostate region segmentation
1
2021
... 胰腺囊性肿瘤自动分割是一种利用计算机图像处理和人工智能技术的创新方法,通过快速、自动地提取胰腺囊性肿瘤区域来提高临床工作效率,这对胰腺囊性肿瘤的诊断和评估具有至关重要的意义.胰腺具有复杂的形态结构和密集的周围组织,传统的手动分割方法费时费力,且存在主观性和人为误差的风险.尽管自动分割技术在医学领域取得了许多显著进展[1,2],但是由于胰腺与邻近的器官和血管之间可能存在相似的密度和纹理特征,其自动分割仍然面临一些独特的难题.此外,胰腺囊性肿瘤常常导致器官形态和密度的变化,进一步增加了自动分割的难度. ...
一种用于前列腺区域分割的改进水平集算法
1
2021
... 胰腺囊性肿瘤自动分割是一种利用计算机图像处理和人工智能技术的创新方法,通过快速、自动地提取胰腺囊性肿瘤区域来提高临床工作效率,这对胰腺囊性肿瘤的诊断和评估具有至关重要的意义.胰腺具有复杂的形态结构和密集的周围组织,传统的手动分割方法费时费力,且存在主观性和人为误差的风险.尽管自动分割技术在医学领域取得了许多显著进展[1,2],但是由于胰腺与邻近的器官和血管之间可能存在相似的密度和纹理特征,其自动分割仍然面临一些独特的难题.此外,胰腺囊性肿瘤常常导致器官形态和密度的变化,进一步增加了自动分割的难度. ...
An automatic segmentation method for lung tumor based on improved region growing algorithm
1
2022
... 传统的图像分割大体分为3类:①基于阈值的分割方法;②基于边缘的分割方法;③基于区域的分割算法.阈值分割方法的优点在于简单容易实现,计算效率高,适用于一些具有明显灰度差异和较为简单的图像分割任务,然而该方法不具有鲁棒性,对于噪声较大或者纹理特征复杂的图像效果可能较差,胰腺囊性肿瘤形态复杂、边界模糊,且与临近的器官和血管之间存在相似的密度和纹理特征,所以难以选择合适的阈值进行分割任务.基于边缘的分割方法对具有明显边缘的图像分割效果较好,能较好提取物体的形状和轮廓信息,但是边缘检测可能受到噪声的干扰,导致边缘不准确或者断裂.由于胰腺周围的组织非常密集,边缘分割效果不理想.基于区域的分割方法可以有效地将图像分割为具有一定一致性的区域,适用于医学图像中具有明显结构和纹理特征的区域.然而,这些方法可能对于具有复杂结构、相似特征或重叠区域的图像具有挑战性.文献[3]中提出了一种基于改进区域生长算法的自动分割算法,该方法利用肺部肿瘤的先验信息自动选择初始种子点,并采用种子点扩展和自动阈值更新机制来提高分割性能. ...
Evolution of image segmentation using deep convolutional neural network: A survey
1
2020
... 传统的图像分割方法往往依赖于手工设计的特征和规则,对于胰腺囊性肿瘤这种形态复杂、边界模糊的情况往往表现不佳.而深度学习技术通过使用神经网络模型,如卷积神经网络(Convolutional Neural Networks,CNN)[4-6]和U-Net[7],可以从大量的标注数据集中学习到肿瘤的表征和上下文信息,从而实现更准确、鲁棒的分割结果.深度学习模型能够自动学习图像中的特征,并通过逐层的特征提取,实现对胰腺囊性肿瘤的精准定位和分割.针对图像质量不佳以及胰腺边界模糊的问题,Cai等人[8]将循环神经网络(Recurrent Neural Network,RNN)和CNN结合,使模型在CT数据和MRI数据上具有更加出色的性能.Oktay等人[9]提出了一种新的适用于CT影像数据的注意力门(Attention Gate,AG)模块,此模块可以同时提高模型的敏感性和预测准确性,能自动学习并聚焦于不同形状和大小的目标结构.Wang等人[10]提出了双通道输入的V-mesh FCN架构和注意力网络,以及空间变换与融合(Spatial Transformation and Fusion,SF)模块,便于更加准确地获取CT中胰腺的位置信息.Xue等人[11]提出由两部分组成的级联网络,第一部分专注于快速定位CT中胰腺区域,第二部分使用具有密集连接的多任务FCN来细化分割图片以进行精细的像素级分割.Asaturyan等人[12]开发了一个名为Hausdorff Sine SegNet(HSSN)的新模型,该模型结合了改进的豪斯多夫距离(Hausdorff Distance, HD)度量和正弦分量来捕获局部边界信息,从而提高MRI中胰腺预测的精度.Proietto等人[13]提出一种新的3D全卷积神经网络,通过学习不同尺度的特征,将多个解码器得到的分割图融合成唯一的分割掩膜.上述模型都是基于不断提取数据的深层特征来达到提高模型对感兴趣区域(Region of Interest,ROI)分割的精度,没有充分利用数据的浅层特征信息,在加深网络层数的同时可能丢失了一部分浅层特征信息,也可能使模型过拟合,从而减低了模型的鲁棒性和有效性. ...
Doubleu-net: A deep convolutional neural network for medical image segmentation
0
2020
HyperDense-Net: a hyper-densely connected CNN for multi-modal image segmentation
1
2018
... 传统的图像分割方法往往依赖于手工设计的特征和规则,对于胰腺囊性肿瘤这种形态复杂、边界模糊的情况往往表现不佳.而深度学习技术通过使用神经网络模型,如卷积神经网络(Convolutional Neural Networks,CNN)[4-6]和U-Net[7],可以从大量的标注数据集中学习到肿瘤的表征和上下文信息,从而实现更准确、鲁棒的分割结果.深度学习模型能够自动学习图像中的特征,并通过逐层的特征提取,实现对胰腺囊性肿瘤的精准定位和分割.针对图像质量不佳以及胰腺边界模糊的问题,Cai等人[8]将循环神经网络(Recurrent Neural Network,RNN)和CNN结合,使模型在CT数据和MRI数据上具有更加出色的性能.Oktay等人[9]提出了一种新的适用于CT影像数据的注意力门(Attention Gate,AG)模块,此模块可以同时提高模型的敏感性和预测准确性,能自动学习并聚焦于不同形状和大小的目标结构.Wang等人[10]提出了双通道输入的V-mesh FCN架构和注意力网络,以及空间变换与融合(Spatial Transformation and Fusion,SF)模块,便于更加准确地获取CT中胰腺的位置信息.Xue等人[11]提出由两部分组成的级联网络,第一部分专注于快速定位CT中胰腺区域,第二部分使用具有密集连接的多任务FCN来细化分割图片以进行精细的像素级分割.Asaturyan等人[12]开发了一个名为Hausdorff Sine SegNet(HSSN)的新模型,该模型结合了改进的豪斯多夫距离(Hausdorff Distance, HD)度量和正弦分量来捕获局部边界信息,从而提高MRI中胰腺预测的精度.Proietto等人[13]提出一种新的3D全卷积神经网络,通过学习不同尺度的特征,将多个解码器得到的分割图融合成唯一的分割掩膜.上述模型都是基于不断提取数据的深层特征来达到提高模型对感兴趣区域(Region of Interest,ROI)分割的精度,没有充分利用数据的浅层特征信息,在加深网络层数的同时可能丢失了一部分浅层特征信息,也可能使模型过拟合,从而减低了模型的鲁棒性和有效性. ...
U-net: convolutional networks for biomedical image segmentation
1
2015
... 传统的图像分割方法往往依赖于手工设计的特征和规则,对于胰腺囊性肿瘤这种形态复杂、边界模糊的情况往往表现不佳.而深度学习技术通过使用神经网络模型,如卷积神经网络(Convolutional Neural Networks,CNN)[4-6]和U-Net[7],可以从大量的标注数据集中学习到肿瘤的表征和上下文信息,从而实现更准确、鲁棒的分割结果.深度学习模型能够自动学习图像中的特征,并通过逐层的特征提取,实现对胰腺囊性肿瘤的精准定位和分割.针对图像质量不佳以及胰腺边界模糊的问题,Cai等人[8]将循环神经网络(Recurrent Neural Network,RNN)和CNN结合,使模型在CT数据和MRI数据上具有更加出色的性能.Oktay等人[9]提出了一种新的适用于CT影像数据的注意力门(Attention Gate,AG)模块,此模块可以同时提高模型的敏感性和预测准确性,能自动学习并聚焦于不同形状和大小的目标结构.Wang等人[10]提出了双通道输入的V-mesh FCN架构和注意力网络,以及空间变换与融合(Spatial Transformation and Fusion,SF)模块,便于更加准确地获取CT中胰腺的位置信息.Xue等人[11]提出由两部分组成的级联网络,第一部分专注于快速定位CT中胰腺区域,第二部分使用具有密集连接的多任务FCN来细化分割图片以进行精细的像素级分割.Asaturyan等人[12]开发了一个名为Hausdorff Sine SegNet(HSSN)的新模型,该模型结合了改进的豪斯多夫距离(Hausdorff Distance, HD)度量和正弦分量来捕获局部边界信息,从而提高MRI中胰腺预测的精度.Proietto等人[13]提出一种新的3D全卷积神经网络,通过学习不同尺度的特征,将多个解码器得到的分割图融合成唯一的分割掩膜.上述模型都是基于不断提取数据的深层特征来达到提高模型对感兴趣区域(Region of Interest,ROI)分割的精度,没有充分利用数据的浅层特征信息,在加深网络层数的同时可能丢失了一部分浅层特征信息,也可能使模型过拟合,从而减低了模型的鲁棒性和有效性. ...
Pancreas segmentation in CT and MRI images via domain specific network designing and recurrent neural contextual learning
3
... 传统的图像分割方法往往依赖于手工设计的特征和规则,对于胰腺囊性肿瘤这种形态复杂、边界模糊的情况往往表现不佳.而深度学习技术通过使用神经网络模型,如卷积神经网络(Convolutional Neural Networks,CNN)[4-6]和U-Net[7],可以从大量的标注数据集中学习到肿瘤的表征和上下文信息,从而实现更准确、鲁棒的分割结果.深度学习模型能够自动学习图像中的特征,并通过逐层的特征提取,实现对胰腺囊性肿瘤的精准定位和分割.针对图像质量不佳以及胰腺边界模糊的问题,Cai等人[8]将循环神经网络(Recurrent Neural Network,RNN)和CNN结合,使模型在CT数据和MRI数据上具有更加出色的性能.Oktay等人[9]提出了一种新的适用于CT影像数据的注意力门(Attention Gate,AG)模块,此模块可以同时提高模型的敏感性和预测准确性,能自动学习并聚焦于不同形状和大小的目标结构.Wang等人[10]提出了双通道输入的V-mesh FCN架构和注意力网络,以及空间变换与融合(Spatial Transformation and Fusion,SF)模块,便于更加准确地获取CT中胰腺的位置信息.Xue等人[11]提出由两部分组成的级联网络,第一部分专注于快速定位CT中胰腺区域,第二部分使用具有密集连接的多任务FCN来细化分割图片以进行精细的像素级分割.Asaturyan等人[12]开发了一个名为Hausdorff Sine SegNet(HSSN)的新模型,该模型结合了改进的豪斯多夫距离(Hausdorff Distance, HD)度量和正弦分量来捕获局部边界信息,从而提高MRI中胰腺预测的精度.Proietto等人[13]提出一种新的3D全卷积神经网络,通过学习不同尺度的特征,将多个解码器得到的分割图融合成唯一的分割掩膜.上述模型都是基于不断提取数据的深层特征来达到提高模型对感兴趣区域(Region of Interest,ROI)分割的精度,没有充分利用数据的浅层特征信息,在加深网络层数的同时可能丢失了一部分浅层特征信息,也可能使模型过拟合,从而减低了模型的鲁棒性和有效性. ...
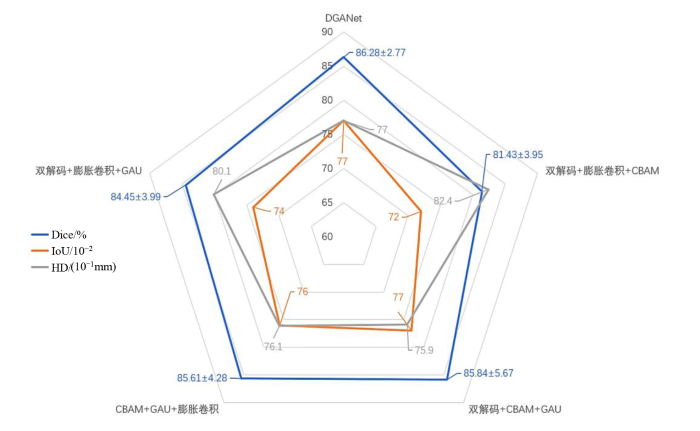
... Comparison of Dice similarity coefficients between the model in different literature and the model in this paper
Table 2 | 模型 | 数据来源 | 数据类型 | Dice/% |
| VA-3DUNet[21] | NIH | 3D(CT) | 86.19±9.03 |
| Attention U-Net[9] | CT-150 | 3D(CT) | 84.00±8.70 |
| Cascaded multitask 3-D fully convolutional networks[11] | 82(CT) | 3D(CT) | 85.9±5.10 |
| HSSN[12] | MRI | 3D(MRI) | 85.7±2.3 |
| PanKNet[13] | Pancreas-MRI | 3D(MRI) | 77.46±8.62 |
| ConResNet[22] | NIH | 3D(CT) | 86.06 |
| PNet-MSA[8] | MRI-79 | 2D(MRI) | 80.7±7.40 |
| Spatial aggregation of holistically-nested convolutional neural networks[23] | NIH | 2D(CT) | 81.27±6.27 |
| deep convolutional LSTM neural network[24] | MRI-79 | 2D(MRI) | 80.5±6.70 |
| RSTN[25] | NIH | 2D(CT) | 84.50±4.97 |
| TransUnet[26] | MICCAI 2015 | 2D(CT) | 73.96 |
| DGANet(本文模型) | 长海医院 | 2D(MRI) | 86.28±2.77 |
表2中前6种方法采用了3D模型,这6种模型都是基于编码解码的结构,通过不断对深层特征的提取,定位ROI区域,与此同时也丢失了一部分浅层信息,导致平均Dice相似系数并没有本文提出的模型高,这表明,增加数据复杂度并不能有效提升模型分割性能,还需要加强模型对浅层信息的利用.表2中文献[8]和文献[23]都分别提出了一种二阶段的分割方法,第一阶段先大概定位胰腺肿瘤的区域,将第一阶段输出的结果当作第二阶段的输入,数据经过第二阶段的处理后得到最后的分割图,但是Dice相似系数并没有我们提出的模型高,可能是在模型的第一阶段没有定位到胰腺肿瘤的大概位置,将错误的信息传入第二阶段,导致了Dice相似系数降低.文献[25]中引入了RNN,使平均Dice相似系数在文献[24]的基础上提高了4%,文献[26]融合了Transformer和U-Net两个经典网络,但是分割效果都没有本文提出的模型效果好,可能是因为胰腺这类软组织在CT成像时对比度较差,从而对后续的实验产生了一定影响,而本研究采用的是T2加权序列图像,囊肿在该序列的信号更强,更加有利于网络对特征的提取,并且我们提出的方法融合了高级特征和浅层特征,网络提取到的特征也就更加明显,所以模型分割效果更好. ...
... 表2中前6种方法采用了3D模型,这6种模型都是基于编码解码的结构,通过不断对深层特征的提取,定位ROI区域,与此同时也丢失了一部分浅层信息,导致平均Dice相似系数并没有本文提出的模型高,这表明,增加数据复杂度并不能有效提升模型分割性能,还需要加强模型对浅层信息的利用.表2中文献[8]和文献[23]都分别提出了一种二阶段的分割方法,第一阶段先大概定位胰腺肿瘤的区域,将第一阶段输出的结果当作第二阶段的输入,数据经过第二阶段的处理后得到最后的分割图,但是Dice相似系数并没有我们提出的模型高,可能是在模型的第一阶段没有定位到胰腺肿瘤的大概位置,将错误的信息传入第二阶段,导致了Dice相似系数降低.文献[25]中引入了RNN,使平均Dice相似系数在文献[24]的基础上提高了4%,文献[26]融合了Transformer和U-Net两个经典网络,但是分割效果都没有本文提出的模型效果好,可能是因为胰腺这类软组织在CT成像时对比度较差,从而对后续的实验产生了一定影响,而本研究采用的是T2加权序列图像,囊肿在该序列的信号更强,更加有利于网络对特征的提取,并且我们提出的方法融合了高级特征和浅层特征,网络提取到的特征也就更加明显,所以模型分割效果更好. ...
Attention U-net: Learning where to look for the pancreas
2
... 传统的图像分割方法往往依赖于手工设计的特征和规则,对于胰腺囊性肿瘤这种形态复杂、边界模糊的情况往往表现不佳.而深度学习技术通过使用神经网络模型,如卷积神经网络(Convolutional Neural Networks,CNN)[4-6]和U-Net[7],可以从大量的标注数据集中学习到肿瘤的表征和上下文信息,从而实现更准确、鲁棒的分割结果.深度学习模型能够自动学习图像中的特征,并通过逐层的特征提取,实现对胰腺囊性肿瘤的精准定位和分割.针对图像质量不佳以及胰腺边界模糊的问题,Cai等人[8]将循环神经网络(Recurrent Neural Network,RNN)和CNN结合,使模型在CT数据和MRI数据上具有更加出色的性能.Oktay等人[9]提出了一种新的适用于CT影像数据的注意力门(Attention Gate,AG)模块,此模块可以同时提高模型的敏感性和预测准确性,能自动学习并聚焦于不同形状和大小的目标结构.Wang等人[10]提出了双通道输入的V-mesh FCN架构和注意力网络,以及空间变换与融合(Spatial Transformation and Fusion,SF)模块,便于更加准确地获取CT中胰腺的位置信息.Xue等人[11]提出由两部分组成的级联网络,第一部分专注于快速定位CT中胰腺区域,第二部分使用具有密集连接的多任务FCN来细化分割图片以进行精细的像素级分割.Asaturyan等人[12]开发了一个名为Hausdorff Sine SegNet(HSSN)的新模型,该模型结合了改进的豪斯多夫距离(Hausdorff Distance, HD)度量和正弦分量来捕获局部边界信息,从而提高MRI中胰腺预测的精度.Proietto等人[13]提出一种新的3D全卷积神经网络,通过学习不同尺度的特征,将多个解码器得到的分割图融合成唯一的分割掩膜.上述模型都是基于不断提取数据的深层特征来达到提高模型对感兴趣区域(Region of Interest,ROI)分割的精度,没有充分利用数据的浅层特征信息,在加深网络层数的同时可能丢失了一部分浅层特征信息,也可能使模型过拟合,从而减低了模型的鲁棒性和有效性. ...
... Comparison of Dice similarity coefficients between the model in different literature and the model in this paper
Table 2 | 模型 | 数据来源 | 数据类型 | Dice/% |
| VA-3DUNet[21] | NIH | 3D(CT) | 86.19±9.03 |
| Attention U-Net[9] | CT-150 | 3D(CT) | 84.00±8.70 |
| Cascaded multitask 3-D fully convolutional networks[11] | 82(CT) | 3D(CT) | 85.9±5.10 |
| HSSN[12] | MRI | 3D(MRI) | 85.7±2.3 |
| PanKNet[13] | Pancreas-MRI | 3D(MRI) | 77.46±8.62 |
| ConResNet[22] | NIH | 3D(CT) | 86.06 |
| PNet-MSA[8] | MRI-79 | 2D(MRI) | 80.7±7.40 |
| Spatial aggregation of holistically-nested convolutional neural networks[23] | NIH | 2D(CT) | 81.27±6.27 |
| deep convolutional LSTM neural network[24] | MRI-79 | 2D(MRI) | 80.5±6.70 |
| RSTN[25] | NIH | 2D(CT) | 84.50±4.97 |
| TransUnet[26] | MICCAI 2015 | 2D(CT) | 73.96 |
| DGANet(本文模型) | 长海医院 | 2D(MRI) | 86.28±2.77 |
表2中前6种方法采用了3D模型,这6种模型都是基于编码解码的结构,通过不断对深层特征的提取,定位ROI区域,与此同时也丢失了一部分浅层信息,导致平均Dice相似系数并没有本文提出的模型高,这表明,增加数据复杂度并不能有效提升模型分割性能,还需要加强模型对浅层信息的利用.表2中文献[8]和文献[23]都分别提出了一种二阶段的分割方法,第一阶段先大概定位胰腺肿瘤的区域,将第一阶段输出的结果当作第二阶段的输入,数据经过第二阶段的处理后得到最后的分割图,但是Dice相似系数并没有我们提出的模型高,可能是在模型的第一阶段没有定位到胰腺肿瘤的大概位置,将错误的信息传入第二阶段,导致了Dice相似系数降低.文献[25]中引入了RNN,使平均Dice相似系数在文献[24]的基础上提高了4%,文献[26]融合了Transformer和U-Net两个经典网络,但是分割效果都没有本文提出的模型效果好,可能是因为胰腺这类软组织在CT成像时对比度较差,从而对后续的实验产生了一定影响,而本研究采用的是T2加权序列图像,囊肿在该序列的信号更强,更加有利于网络对特征的提取,并且我们提出的方法融合了高级特征和浅层特征,网络提取到的特征也就更加明显,所以模型分割效果更好. ...
Pancreas segmentation using a dual-input V-mesh network
1
2021
... 传统的图像分割方法往往依赖于手工设计的特征和规则,对于胰腺囊性肿瘤这种形态复杂、边界模糊的情况往往表现不佳.而深度学习技术通过使用神经网络模型,如卷积神经网络(Convolutional Neural Networks,CNN)[4-6]和U-Net[7],可以从大量的标注数据集中学习到肿瘤的表征和上下文信息,从而实现更准确、鲁棒的分割结果.深度学习模型能够自动学习图像中的特征,并通过逐层的特征提取,实现对胰腺囊性肿瘤的精准定位和分割.针对图像质量不佳以及胰腺边界模糊的问题,Cai等人[8]将循环神经网络(Recurrent Neural Network,RNN)和CNN结合,使模型在CT数据和MRI数据上具有更加出色的性能.Oktay等人[9]提出了一种新的适用于CT影像数据的注意力门(Attention Gate,AG)模块,此模块可以同时提高模型的敏感性和预测准确性,能自动学习并聚焦于不同形状和大小的目标结构.Wang等人[10]提出了双通道输入的V-mesh FCN架构和注意力网络,以及空间变换与融合(Spatial Transformation and Fusion,SF)模块,便于更加准确地获取CT中胰腺的位置信息.Xue等人[11]提出由两部分组成的级联网络,第一部分专注于快速定位CT中胰腺区域,第二部分使用具有密集连接的多任务FCN来细化分割图片以进行精细的像素级分割.Asaturyan等人[12]开发了一个名为Hausdorff Sine SegNet(HSSN)的新模型,该模型结合了改进的豪斯多夫距离(Hausdorff Distance, HD)度量和正弦分量来捕获局部边界信息,从而提高MRI中胰腺预测的精度.Proietto等人[13]提出一种新的3D全卷积神经网络,通过学习不同尺度的特征,将多个解码器得到的分割图融合成唯一的分割掩膜.上述模型都是基于不断提取数据的深层特征来达到提高模型对感兴趣区域(Region of Interest,ROI)分割的精度,没有充分利用数据的浅层特征信息,在加深网络层数的同时可能丢失了一部分浅层特征信息,也可能使模型过拟合,从而减低了模型的鲁棒性和有效性. ...
Cascaded multitask 3-D fully convolutional networks for pancreas segmentation
2
2019
... 传统的图像分割方法往往依赖于手工设计的特征和规则,对于胰腺囊性肿瘤这种形态复杂、边界模糊的情况往往表现不佳.而深度学习技术通过使用神经网络模型,如卷积神经网络(Convolutional Neural Networks,CNN)[4-6]和U-Net[7],可以从大量的标注数据集中学习到肿瘤的表征和上下文信息,从而实现更准确、鲁棒的分割结果.深度学习模型能够自动学习图像中的特征,并通过逐层的特征提取,实现对胰腺囊性肿瘤的精准定位和分割.针对图像质量不佳以及胰腺边界模糊的问题,Cai等人[8]将循环神经网络(Recurrent Neural Network,RNN)和CNN结合,使模型在CT数据和MRI数据上具有更加出色的性能.Oktay等人[9]提出了一种新的适用于CT影像数据的注意力门(Attention Gate,AG)模块,此模块可以同时提高模型的敏感性和预测准确性,能自动学习并聚焦于不同形状和大小的目标结构.Wang等人[10]提出了双通道输入的V-mesh FCN架构和注意力网络,以及空间变换与融合(Spatial Transformation and Fusion,SF)模块,便于更加准确地获取CT中胰腺的位置信息.Xue等人[11]提出由两部分组成的级联网络,第一部分专注于快速定位CT中胰腺区域,第二部分使用具有密集连接的多任务FCN来细化分割图片以进行精细的像素级分割.Asaturyan等人[12]开发了一个名为Hausdorff Sine SegNet(HSSN)的新模型,该模型结合了改进的豪斯多夫距离(Hausdorff Distance, HD)度量和正弦分量来捕获局部边界信息,从而提高MRI中胰腺预测的精度.Proietto等人[13]提出一种新的3D全卷积神经网络,通过学习不同尺度的特征,将多个解码器得到的分割图融合成唯一的分割掩膜.上述模型都是基于不断提取数据的深层特征来达到提高模型对感兴趣区域(Region of Interest,ROI)分割的精度,没有充分利用数据的浅层特征信息,在加深网络层数的同时可能丢失了一部分浅层特征信息,也可能使模型过拟合,从而减低了模型的鲁棒性和有效性. ...
... Comparison of Dice similarity coefficients between the model in different literature and the model in this paper
Table 2 | 模型 | 数据来源 | 数据类型 | Dice/% |
| VA-3DUNet[21] | NIH | 3D(CT) | 86.19±9.03 |
| Attention U-Net[9] | CT-150 | 3D(CT) | 84.00±8.70 |
| Cascaded multitask 3-D fully convolutional networks[11] | 82(CT) | 3D(CT) | 85.9±5.10 |
| HSSN[12] | MRI | 3D(MRI) | 85.7±2.3 |
| PanKNet[13] | Pancreas-MRI | 3D(MRI) | 77.46±8.62 |
| ConResNet[22] | NIH | 3D(CT) | 86.06 |
| PNet-MSA[8] | MRI-79 | 2D(MRI) | 80.7±7.40 |
| Spatial aggregation of holistically-nested convolutional neural networks[23] | NIH | 2D(CT) | 81.27±6.27 |
| deep convolutional LSTM neural network[24] | MRI-79 | 2D(MRI) | 80.5±6.70 |
| RSTN[25] | NIH | 2D(CT) | 84.50±4.97 |
| TransUnet[26] | MICCAI 2015 | 2D(CT) | 73.96 |
| DGANet(本文模型) | 长海医院 | 2D(MRI) | 86.28±2.77 |
表2中前6种方法采用了3D模型,这6种模型都是基于编码解码的结构,通过不断对深层特征的提取,定位ROI区域,与此同时也丢失了一部分浅层信息,导致平均Dice相似系数并没有本文提出的模型高,这表明,增加数据复杂度并不能有效提升模型分割性能,还需要加强模型对浅层信息的利用.表2中文献[8]和文献[23]都分别提出了一种二阶段的分割方法,第一阶段先大概定位胰腺肿瘤的区域,将第一阶段输出的结果当作第二阶段的输入,数据经过第二阶段的处理后得到最后的分割图,但是Dice相似系数并没有我们提出的模型高,可能是在模型的第一阶段没有定位到胰腺肿瘤的大概位置,将错误的信息传入第二阶段,导致了Dice相似系数降低.文献[25]中引入了RNN,使平均Dice相似系数在文献[24]的基础上提高了4%,文献[26]融合了Transformer和U-Net两个经典网络,但是分割效果都没有本文提出的模型效果好,可能是因为胰腺这类软组织在CT成像时对比度较差,从而对后续的实验产生了一定影响,而本研究采用的是T2加权序列图像,囊肿在该序列的信号更强,更加有利于网络对特征的提取,并且我们提出的方法融合了高级特征和浅层特征,网络提取到的特征也就更加明显,所以模型分割效果更好. ...
Advancing pancreas segmentation in multi-protocol mri volumes using hausdorff-sine loss function
2
2019
... 传统的图像分割方法往往依赖于手工设计的特征和规则,对于胰腺囊性肿瘤这种形态复杂、边界模糊的情况往往表现不佳.而深度学习技术通过使用神经网络模型,如卷积神经网络(Convolutional Neural Networks,CNN)[4-6]和U-Net[7],可以从大量的标注数据集中学习到肿瘤的表征和上下文信息,从而实现更准确、鲁棒的分割结果.深度学习模型能够自动学习图像中的特征,并通过逐层的特征提取,实现对胰腺囊性肿瘤的精准定位和分割.针对图像质量不佳以及胰腺边界模糊的问题,Cai等人[8]将循环神经网络(Recurrent Neural Network,RNN)和CNN结合,使模型在CT数据和MRI数据上具有更加出色的性能.Oktay等人[9]提出了一种新的适用于CT影像数据的注意力门(Attention Gate,AG)模块,此模块可以同时提高模型的敏感性和预测准确性,能自动学习并聚焦于不同形状和大小的目标结构.Wang等人[10]提出了双通道输入的V-mesh FCN架构和注意力网络,以及空间变换与融合(Spatial Transformation and Fusion,SF)模块,便于更加准确地获取CT中胰腺的位置信息.Xue等人[11]提出由两部分组成的级联网络,第一部分专注于快速定位CT中胰腺区域,第二部分使用具有密集连接的多任务FCN来细化分割图片以进行精细的像素级分割.Asaturyan等人[12]开发了一个名为Hausdorff Sine SegNet(HSSN)的新模型,该模型结合了改进的豪斯多夫距离(Hausdorff Distance, HD)度量和正弦分量来捕获局部边界信息,从而提高MRI中胰腺预测的精度.Proietto等人[13]提出一种新的3D全卷积神经网络,通过学习不同尺度的特征,将多个解码器得到的分割图融合成唯一的分割掩膜.上述模型都是基于不断提取数据的深层特征来达到提高模型对感兴趣区域(Region of Interest,ROI)分割的精度,没有充分利用数据的浅层特征信息,在加深网络层数的同时可能丢失了一部分浅层特征信息,也可能使模型过拟合,从而减低了模型的鲁棒性和有效性. ...
... Comparison of Dice similarity coefficients between the model in different literature and the model in this paper
Table 2 | 模型 | 数据来源 | 数据类型 | Dice/% |
| VA-3DUNet[21] | NIH | 3D(CT) | 86.19±9.03 |
| Attention U-Net[9] | CT-150 | 3D(CT) | 84.00±8.70 |
| Cascaded multitask 3-D fully convolutional networks[11] | 82(CT) | 3D(CT) | 85.9±5.10 |
| HSSN[12] | MRI | 3D(MRI) | 85.7±2.3 |
| PanKNet[13] | Pancreas-MRI | 3D(MRI) | 77.46±8.62 |
| ConResNet[22] | NIH | 3D(CT) | 86.06 |
| PNet-MSA[8] | MRI-79 | 2D(MRI) | 80.7±7.40 |
| Spatial aggregation of holistically-nested convolutional neural networks[23] | NIH | 2D(CT) | 81.27±6.27 |
| deep convolutional LSTM neural network[24] | MRI-79 | 2D(MRI) | 80.5±6.70 |
| RSTN[25] | NIH | 2D(CT) | 84.50±4.97 |
| TransUnet[26] | MICCAI 2015 | 2D(CT) | 73.96 |
| DGANet(本文模型) | 长海医院 | 2D(MRI) | 86.28±2.77 |
表2中前6种方法采用了3D模型,这6种模型都是基于编码解码的结构,通过不断对深层特征的提取,定位ROI区域,与此同时也丢失了一部分浅层信息,导致平均Dice相似系数并没有本文提出的模型高,这表明,增加数据复杂度并不能有效提升模型分割性能,还需要加强模型对浅层信息的利用.表2中文献[8]和文献[23]都分别提出了一种二阶段的分割方法,第一阶段先大概定位胰腺肿瘤的区域,将第一阶段输出的结果当作第二阶段的输入,数据经过第二阶段的处理后得到最后的分割图,但是Dice相似系数并没有我们提出的模型高,可能是在模型的第一阶段没有定位到胰腺肿瘤的大概位置,将错误的信息传入第二阶段,导致了Dice相似系数降低.文献[25]中引入了RNN,使平均Dice相似系数在文献[24]的基础上提高了4%,文献[26]融合了Transformer和U-Net两个经典网络,但是分割效果都没有本文提出的模型效果好,可能是因为胰腺这类软组织在CT成像时对比度较差,从而对后续的实验产生了一定影响,而本研究采用的是T2加权序列图像,囊肿在该序列的信号更强,更加有利于网络对特征的提取,并且我们提出的方法融合了高级特征和浅层特征,网络提取到的特征也就更加明显,所以模型分割效果更好. ...
Hierarchical 3D feature learning forpancreas segmentation
2
2021
... 传统的图像分割方法往往依赖于手工设计的特征和规则,对于胰腺囊性肿瘤这种形态复杂、边界模糊的情况往往表现不佳.而深度学习技术通过使用神经网络模型,如卷积神经网络(Convolutional Neural Networks,CNN)[4-6]和U-Net[7],可以从大量的标注数据集中学习到肿瘤的表征和上下文信息,从而实现更准确、鲁棒的分割结果.深度学习模型能够自动学习图像中的特征,并通过逐层的特征提取,实现对胰腺囊性肿瘤的精准定位和分割.针对图像质量不佳以及胰腺边界模糊的问题,Cai等人[8]将循环神经网络(Recurrent Neural Network,RNN)和CNN结合,使模型在CT数据和MRI数据上具有更加出色的性能.Oktay等人[9]提出了一种新的适用于CT影像数据的注意力门(Attention Gate,AG)模块,此模块可以同时提高模型的敏感性和预测准确性,能自动学习并聚焦于不同形状和大小的目标结构.Wang等人[10]提出了双通道输入的V-mesh FCN架构和注意力网络,以及空间变换与融合(Spatial Transformation and Fusion,SF)模块,便于更加准确地获取CT中胰腺的位置信息.Xue等人[11]提出由两部分组成的级联网络,第一部分专注于快速定位CT中胰腺区域,第二部分使用具有密集连接的多任务FCN来细化分割图片以进行精细的像素级分割.Asaturyan等人[12]开发了一个名为Hausdorff Sine SegNet(HSSN)的新模型,该模型结合了改进的豪斯多夫距离(Hausdorff Distance, HD)度量和正弦分量来捕获局部边界信息,从而提高MRI中胰腺预测的精度.Proietto等人[13]提出一种新的3D全卷积神经网络,通过学习不同尺度的特征,将多个解码器得到的分割图融合成唯一的分割掩膜.上述模型都是基于不断提取数据的深层特征来达到提高模型对感兴趣区域(Region of Interest,ROI)分割的精度,没有充分利用数据的浅层特征信息,在加深网络层数的同时可能丢失了一部分浅层特征信息,也可能使模型过拟合,从而减低了模型的鲁棒性和有效性. ...
... Comparison of Dice similarity coefficients between the model in different literature and the model in this paper
Table 2 | 模型 | 数据来源 | 数据类型 | Dice/% |
| VA-3DUNet[21] | NIH | 3D(CT) | 86.19±9.03 |
| Attention U-Net[9] | CT-150 | 3D(CT) | 84.00±8.70 |
| Cascaded multitask 3-D fully convolutional networks[11] | 82(CT) | 3D(CT) | 85.9±5.10 |
| HSSN[12] | MRI | 3D(MRI) | 85.7±2.3 |
| PanKNet[13] | Pancreas-MRI | 3D(MRI) | 77.46±8.62 |
| ConResNet[22] | NIH | 3D(CT) | 86.06 |
| PNet-MSA[8] | MRI-79 | 2D(MRI) | 80.7±7.40 |
| Spatial aggregation of holistically-nested convolutional neural networks[23] | NIH | 2D(CT) | 81.27±6.27 |
| deep convolutional LSTM neural network[24] | MRI-79 | 2D(MRI) | 80.5±6.70 |
| RSTN[25] | NIH | 2D(CT) | 84.50±4.97 |
| TransUnet[26] | MICCAI 2015 | 2D(CT) | 73.96 |
| DGANet(本文模型) | 长海医院 | 2D(MRI) | 86.28±2.77 |
表2中前6种方法采用了3D模型,这6种模型都是基于编码解码的结构,通过不断对深层特征的提取,定位ROI区域,与此同时也丢失了一部分浅层信息,导致平均Dice相似系数并没有本文提出的模型高,这表明,增加数据复杂度并不能有效提升模型分割性能,还需要加强模型对浅层信息的利用.表2中文献[8]和文献[23]都分别提出了一种二阶段的分割方法,第一阶段先大概定位胰腺肿瘤的区域,将第一阶段输出的结果当作第二阶段的输入,数据经过第二阶段的处理后得到最后的分割图,但是Dice相似系数并没有我们提出的模型高,可能是在模型的第一阶段没有定位到胰腺肿瘤的大概位置,将错误的信息传入第二阶段,导致了Dice相似系数降低.文献[25]中引入了RNN,使平均Dice相似系数在文献[24]的基础上提高了4%,文献[26]融合了Transformer和U-Net两个经典网络,但是分割效果都没有本文提出的模型效果好,可能是因为胰腺这类软组织在CT成像时对比度较差,从而对后续的实验产生了一定影响,而本研究采用的是T2加权序列图像,囊肿在该序列的信号更强,更加有利于网络对特征的提取,并且我们提出的方法融合了高级特征和浅层特征,网络提取到的特征也就更加明显,所以模型分割效果更好. ...
Pancreas segmentation based on dual-decoding U-Net
1
2022
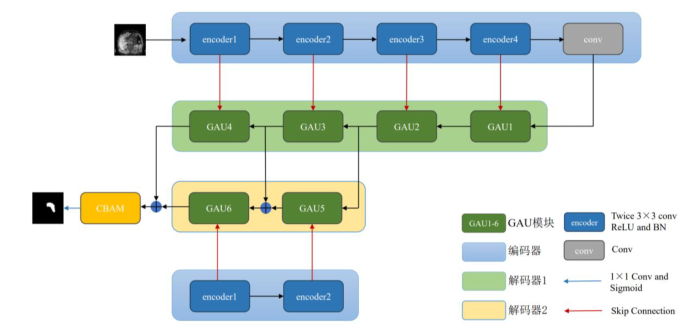
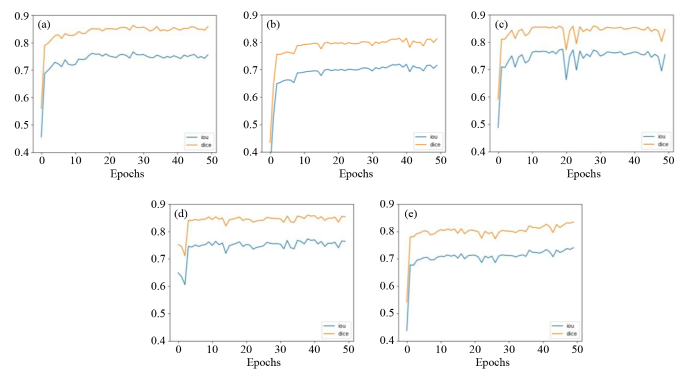
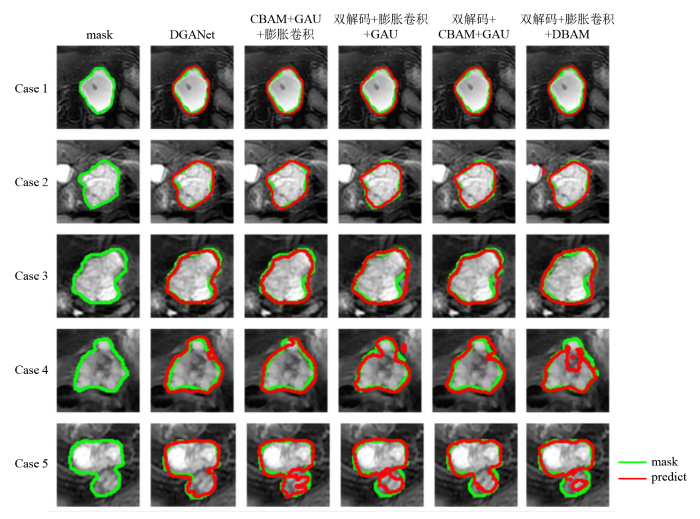
... 为了提升对胰腺囊性肿瘤边缘的分割精度以及加强对浅层特征信息的利用,本文提出一种融合双解码和全局注意力上采样模块的深度学习分割模型(Combining Dual Decoding and Global Attention Upsampling Modules Network,DGANet),该模型结合双解码[14]、膨胀卷积[15]、全局注意力上采样模块(Global Attention Upsample,GAU)[16]和卷积注意力模块(Convolutional Block Attention Module,CBAM)[17]的深度学习分割网络.首先在编码阶段使用膨胀卷积对图像进行编码,使用GAU和双解码进行解码,并融合浅层特征,在最后一层卷积之前添加CBAM模块,使网络更好地学习图像特征. ...
基于双解码U型卷积神经网络的胰腺分割
1
2022
... 为了提升对胰腺囊性肿瘤边缘的分割精度以及加强对浅层特征信息的利用,本文提出一种融合双解码和全局注意力上采样模块的深度学习分割模型(Combining Dual Decoding and Global Attention Upsampling Modules Network,DGANet),该模型结合双解码[14]、膨胀卷积[15]、全局注意力上采样模块(Global Attention Upsample,GAU)[16]和卷积注意力模块(Convolutional Block Attention Module,CBAM)[17]的深度学习分割网络.首先在编码阶段使用膨胀卷积对图像进行编码,使用GAU和双解码进行解码,并融合浅层特征,在最后一层卷积之前添加CBAM模块,使网络更好地学习图像特征. ...
CSRNet: dilated convolutional neural networks for understanding the highly congested scenes
1
2018
... 为了提升对胰腺囊性肿瘤边缘的分割精度以及加强对浅层特征信息的利用,本文提出一种融合双解码和全局注意力上采样模块的深度学习分割模型(Combining Dual Decoding and Global Attention Upsampling Modules Network,DGANet),该模型结合双解码[14]、膨胀卷积[15]、全局注意力上采样模块(Global Attention Upsample,GAU)[16]和卷积注意力模块(Convolutional Block Attention Module,CBAM)[17]的深度学习分割网络.首先在编码阶段使用膨胀卷积对图像进行编码,使用GAU和双解码进行解码,并融合浅层特征,在最后一层卷积之前添加CBAM模块,使网络更好地学习图像特征. ...
Pyramid attention network for semantic segmentation
1
... 为了提升对胰腺囊性肿瘤边缘的分割精度以及加强对浅层特征信息的利用,本文提出一种融合双解码和全局注意力上采样模块的深度学习分割模型(Combining Dual Decoding and Global Attention Upsampling Modules Network,DGANet),该模型结合双解码[14]、膨胀卷积[15]、全局注意力上采样模块(Global Attention Upsample,GAU)[16]和卷积注意力模块(Convolutional Block Attention Module,CBAM)[17]的深度学习分割网络.首先在编码阶段使用膨胀卷积对图像进行编码,使用GAU和双解码进行解码,并融合浅层特征,在最后一层卷积之前添加CBAM模块,使网络更好地学习图像特征. ...
CBAM: convolutional block attention module
1
2018
... 为了提升对胰腺囊性肿瘤边缘的分割精度以及加强对浅层特征信息的利用,本文提出一种融合双解码和全局注意力上采样模块的深度学习分割模型(Combining Dual Decoding and Global Attention Upsampling Modules Network,DGANet),该模型结合双解码[14]、膨胀卷积[15]、全局注意力上采样模块(Global Attention Upsample,GAU)[16]和卷积注意力模块(Convolutional Block Attention Module,CBAM)[17]的深度学习分割网络.首先在编码阶段使用膨胀卷积对图像进行编码,使用GAU和双解码进行解码,并融合浅层特征,在最后一层卷积之前添加CBAM模块,使网络更好地学习图像特征. ...
Unet++: Redesigning skip connections to exploit multiscale features in image segmentation
1
2019
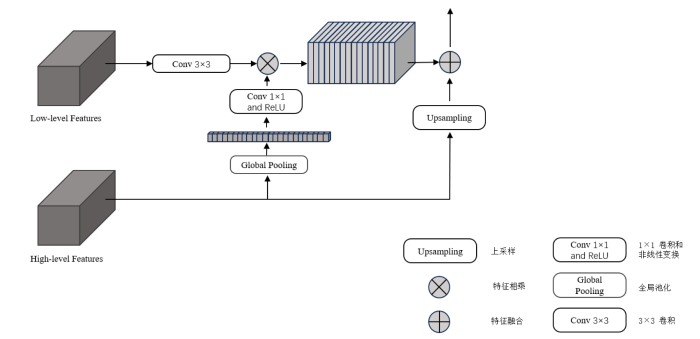
... 胰腺区域占整个腹部区域比例较小,所以在医学图像分割任务中,胰腺分割往往是最具挑战性的分割任务之一.在图像分割领域,传统的编码-解码结构网络通常只包含一个解码器,可以通过不断增加网络的深度,一定程度上可以达到精准提取目标区域的目的.但是,网络越深,丢失掉的边缘等特征信息也就越多,所以在小器官分割任务中,用传统的编码-解码结构分割的精度还有待提高.双解码可以在增加网络深度的前提下,融合图像的低级特征信息,保留更多边缘空间等特征信息,其结构如图1所示.将编码器(Encoder)最后一层卷积的输出作为解码器1的输入,解码器1由4个GAU模块组成,将编码器中每一层和解码器1中每一层进行跳跃连接(Skip Connection)[18].再用解码器1中最后两层的输出作为解码器2中每层的输入,同时将编码器的前两层和解码器2中的两个GAU模块进行跳跃连接.最后把解码器1和解码器2得到的特征信息进行特征融合,以此来加强模型对边缘分割的效果. ...
Multi-scale context aggregation by dilated convolutions
1
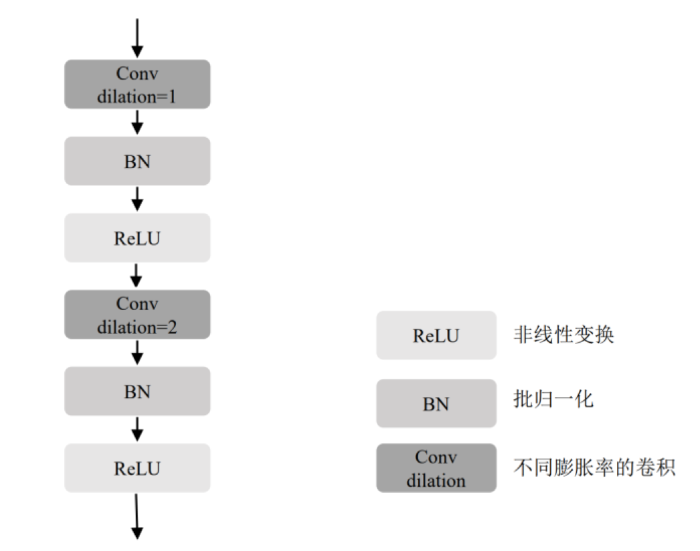
... 在深度学习网络中,为了增加感受野[19],一般会对图像进行下采样.但下采样降低了图像的空间分辨率,且胰腺区域占整个腹部区域较小,其本身的空间分辨率不高,为了保持图像的空间分辨率和扩大感受野,在编码过程中采用了膨胀卷积(Dilated Convolution),结构如图3所示.一个卷积层包含两次连续的Conv操作、BN和ReLU,在两个卷积操作中膨胀率(dilation)分别设置为1和2[20]. ...
Understanding convolution for semantic segmentation
1
2018
... 在深度学习网络中,为了增加感受野[19],一般会对图像进行下采样.但下采样降低了图像的空间分辨率,且胰腺区域占整个腹部区域较小,其本身的空间分辨率不高,为了保持图像的空间分辨率和扩大感受野,在编码过程中采用了膨胀卷积(Dilated Convolution),结构如图3所示.一个卷积层包含两次连续的Conv操作、BN和ReLU,在两个卷积操作中膨胀率(dilation)分别设置为1和2[20]. ...
View adaptive learning for pancreas segmentation
1
2021
... Comparison of Dice similarity coefficients between the model in different literature and the model in this paper
Table 2 | 模型 | 数据来源 | 数据类型 | Dice/% |
| VA-3DUNet[21] | NIH | 3D(CT) | 86.19±9.03 |
| Attention U-Net[9] | CT-150 | 3D(CT) | 84.00±8.70 |
| Cascaded multitask 3-D fully convolutional networks[11] | 82(CT) | 3D(CT) | 85.9±5.10 |
| HSSN[12] | MRI | 3D(MRI) | 85.7±2.3 |
| PanKNet[13] | Pancreas-MRI | 3D(MRI) | 77.46±8.62 |
| ConResNet[22] | NIH | 3D(CT) | 86.06 |
| PNet-MSA[8] | MRI-79 | 2D(MRI) | 80.7±7.40 |
| Spatial aggregation of holistically-nested convolutional neural networks[23] | NIH | 2D(CT) | 81.27±6.27 |
| deep convolutional LSTM neural network[24] | MRI-79 | 2D(MRI) | 80.5±6.70 |
| RSTN[25] | NIH | 2D(CT) | 84.50±4.97 |
| TransUnet[26] | MICCAI 2015 | 2D(CT) | 73.96 |
| DGANet(本文模型) | 长海医院 | 2D(MRI) | 86.28±2.77 |
表2中前6种方法采用了3D模型,这6种模型都是基于编码解码的结构,通过不断对深层特征的提取,定位ROI区域,与此同时也丢失了一部分浅层信息,导致平均Dice相似系数并没有本文提出的模型高,这表明,增加数据复杂度并不能有效提升模型分割性能,还需要加强模型对浅层信息的利用.表2中文献[8]和文献[23]都分别提出了一种二阶段的分割方法,第一阶段先大概定位胰腺肿瘤的区域,将第一阶段输出的结果当作第二阶段的输入,数据经过第二阶段的处理后得到最后的分割图,但是Dice相似系数并没有我们提出的模型高,可能是在模型的第一阶段没有定位到胰腺肿瘤的大概位置,将错误的信息传入第二阶段,导致了Dice相似系数降低.文献[25]中引入了RNN,使平均Dice相似系数在文献[24]的基础上提高了4%,文献[26]融合了Transformer和U-Net两个经典网络,但是分割效果都没有本文提出的模型效果好,可能是因为胰腺这类软组织在CT成像时对比度较差,从而对后续的实验产生了一定影响,而本研究采用的是T2加权序列图像,囊肿在该序列的信号更强,更加有利于网络对特征的提取,并且我们提出的方法融合了高级特征和浅层特征,网络提取到的特征也就更加明显,所以模型分割效果更好. ...
Inter-slice context residual learning for 3D medical image segmentation
1
2020
... Comparison of Dice similarity coefficients between the model in different literature and the model in this paper
Table 2 | 模型 | 数据来源 | 数据类型 | Dice/% |
| VA-3DUNet[21] | NIH | 3D(CT) | 86.19±9.03 |
| Attention U-Net[9] | CT-150 | 3D(CT) | 84.00±8.70 |
| Cascaded multitask 3-D fully convolutional networks[11] | 82(CT) | 3D(CT) | 85.9±5.10 |
| HSSN[12] | MRI | 3D(MRI) | 85.7±2.3 |
| PanKNet[13] | Pancreas-MRI | 3D(MRI) | 77.46±8.62 |
| ConResNet[22] | NIH | 3D(CT) | 86.06 |
| PNet-MSA[8] | MRI-79 | 2D(MRI) | 80.7±7.40 |
| Spatial aggregation of holistically-nested convolutional neural networks[23] | NIH | 2D(CT) | 81.27±6.27 |
| deep convolutional LSTM neural network[24] | MRI-79 | 2D(MRI) | 80.5±6.70 |
| RSTN[25] | NIH | 2D(CT) | 84.50±4.97 |
| TransUnet[26] | MICCAI 2015 | 2D(CT) | 73.96 |
| DGANet(本文模型) | 长海医院 | 2D(MRI) | 86.28±2.77 |
表2中前6种方法采用了3D模型,这6种模型都是基于编码解码的结构,通过不断对深层特征的提取,定位ROI区域,与此同时也丢失了一部分浅层信息,导致平均Dice相似系数并没有本文提出的模型高,这表明,增加数据复杂度并不能有效提升模型分割性能,还需要加强模型对浅层信息的利用.表2中文献[8]和文献[23]都分别提出了一种二阶段的分割方法,第一阶段先大概定位胰腺肿瘤的区域,将第一阶段输出的结果当作第二阶段的输入,数据经过第二阶段的处理后得到最后的分割图,但是Dice相似系数并没有我们提出的模型高,可能是在模型的第一阶段没有定位到胰腺肿瘤的大概位置,将错误的信息传入第二阶段,导致了Dice相似系数降低.文献[25]中引入了RNN,使平均Dice相似系数在文献[24]的基础上提高了4%,文献[26]融合了Transformer和U-Net两个经典网络,但是分割效果都没有本文提出的模型效果好,可能是因为胰腺这类软组织在CT成像时对比度较差,从而对后续的实验产生了一定影响,而本研究采用的是T2加权序列图像,囊肿在该序列的信号更强,更加有利于网络对特征的提取,并且我们提出的方法融合了高级特征和浅层特征,网络提取到的特征也就更加明显,所以模型分割效果更好. ...
Spatial aggregation of holistically-nested convolutional neural networks for automated pancreas localization and segmentation
2
2018
... Comparison of Dice similarity coefficients between the model in different literature and the model in this paper
Table 2 | 模型 | 数据来源 | 数据类型 | Dice/% |
| VA-3DUNet[21] | NIH | 3D(CT) | 86.19±9.03 |
| Attention U-Net[9] | CT-150 | 3D(CT) | 84.00±8.70 |
| Cascaded multitask 3-D fully convolutional networks[11] | 82(CT) | 3D(CT) | 85.9±5.10 |
| HSSN[12] | MRI | 3D(MRI) | 85.7±2.3 |
| PanKNet[13] | Pancreas-MRI | 3D(MRI) | 77.46±8.62 |
| ConResNet[22] | NIH | 3D(CT) | 86.06 |
| PNet-MSA[8] | MRI-79 | 2D(MRI) | 80.7±7.40 |
| Spatial aggregation of holistically-nested convolutional neural networks[23] | NIH | 2D(CT) | 81.27±6.27 |
| deep convolutional LSTM neural network[24] | MRI-79 | 2D(MRI) | 80.5±6.70 |
| RSTN[25] | NIH | 2D(CT) | 84.50±4.97 |
| TransUnet[26] | MICCAI 2015 | 2D(CT) | 73.96 |
| DGANet(本文模型) | 长海医院 | 2D(MRI) | 86.28±2.77 |
表2中前6种方法采用了3D模型,这6种模型都是基于编码解码的结构,通过不断对深层特征的提取,定位ROI区域,与此同时也丢失了一部分浅层信息,导致平均Dice相似系数并没有本文提出的模型高,这表明,增加数据复杂度并不能有效提升模型分割性能,还需要加强模型对浅层信息的利用.表2中文献[8]和文献[23]都分别提出了一种二阶段的分割方法,第一阶段先大概定位胰腺肿瘤的区域,将第一阶段输出的结果当作第二阶段的输入,数据经过第二阶段的处理后得到最后的分割图,但是Dice相似系数并没有我们提出的模型高,可能是在模型的第一阶段没有定位到胰腺肿瘤的大概位置,将错误的信息传入第二阶段,导致了Dice相似系数降低.文献[25]中引入了RNN,使平均Dice相似系数在文献[24]的基础上提高了4%,文献[26]融合了Transformer和U-Net两个经典网络,但是分割效果都没有本文提出的模型效果好,可能是因为胰腺这类软组织在CT成像时对比度较差,从而对后续的实验产生了一定影响,而本研究采用的是T2加权序列图像,囊肿在该序列的信号更强,更加有利于网络对特征的提取,并且我们提出的方法融合了高级特征和浅层特征,网络提取到的特征也就更加明显,所以模型分割效果更好. ...
... 表2中前6种方法采用了3D模型,这6种模型都是基于编码解码的结构,通过不断对深层特征的提取,定位ROI区域,与此同时也丢失了一部分浅层信息,导致平均Dice相似系数并没有本文提出的模型高,这表明,增加数据复杂度并不能有效提升模型分割性能,还需要加强模型对浅层信息的利用.表2中文献[8]和文献[23]都分别提出了一种二阶段的分割方法,第一阶段先大概定位胰腺肿瘤的区域,将第一阶段输出的结果当作第二阶段的输入,数据经过第二阶段的处理后得到最后的分割图,但是Dice相似系数并没有我们提出的模型高,可能是在模型的第一阶段没有定位到胰腺肿瘤的大概位置,将错误的信息传入第二阶段,导致了Dice相似系数降低.文献[25]中引入了RNN,使平均Dice相似系数在文献[24]的基础上提高了4%,文献[26]融合了Transformer和U-Net两个经典网络,但是分割效果都没有本文提出的模型效果好,可能是因为胰腺这类软组织在CT成像时对比度较差,从而对后续的实验产生了一定影响,而本研究采用的是T2加权序列图像,囊肿在该序列的信号更强,更加有利于网络对特征的提取,并且我们提出的方法融合了高级特征和浅层特征,网络提取到的特征也就更加明显,所以模型分割效果更好. ...
Improving deep pancreas segmentation in CT and MRI images via recurrent neural contextual learning and direct loss function
2
... Comparison of Dice similarity coefficients between the model in different literature and the model in this paper
Table 2 | 模型 | 数据来源 | 数据类型 | Dice/% |
| VA-3DUNet[21] | NIH | 3D(CT) | 86.19±9.03 |
| Attention U-Net[9] | CT-150 | 3D(CT) | 84.00±8.70 |
| Cascaded multitask 3-D fully convolutional networks[11] | 82(CT) | 3D(CT) | 85.9±5.10 |
| HSSN[12] | MRI | 3D(MRI) | 85.7±2.3 |
| PanKNet[13] | Pancreas-MRI | 3D(MRI) | 77.46±8.62 |
| ConResNet[22] | NIH | 3D(CT) | 86.06 |
| PNet-MSA[8] | MRI-79 | 2D(MRI) | 80.7±7.40 |
| Spatial aggregation of holistically-nested convolutional neural networks[23] | NIH | 2D(CT) | 81.27±6.27 |
| deep convolutional LSTM neural network[24] | MRI-79 | 2D(MRI) | 80.5±6.70 |
| RSTN[25] | NIH | 2D(CT) | 84.50±4.97 |
| TransUnet[26] | MICCAI 2015 | 2D(CT) | 73.96 |
| DGANet(本文模型) | 长海医院 | 2D(MRI) | 86.28±2.77 |
表2中前6种方法采用了3D模型,这6种模型都是基于编码解码的结构,通过不断对深层特征的提取,定位ROI区域,与此同时也丢失了一部分浅层信息,导致平均Dice相似系数并没有本文提出的模型高,这表明,增加数据复杂度并不能有效提升模型分割性能,还需要加强模型对浅层信息的利用.表2中文献[8]和文献[23]都分别提出了一种二阶段的分割方法,第一阶段先大概定位胰腺肿瘤的区域,将第一阶段输出的结果当作第二阶段的输入,数据经过第二阶段的处理后得到最后的分割图,但是Dice相似系数并没有我们提出的模型高,可能是在模型的第一阶段没有定位到胰腺肿瘤的大概位置,将错误的信息传入第二阶段,导致了Dice相似系数降低.文献[25]中引入了RNN,使平均Dice相似系数在文献[24]的基础上提高了4%,文献[26]融合了Transformer和U-Net两个经典网络,但是分割效果都没有本文提出的模型效果好,可能是因为胰腺这类软组织在CT成像时对比度较差,从而对后续的实验产生了一定影响,而本研究采用的是T2加权序列图像,囊肿在该序列的信号更强,更加有利于网络对特征的提取,并且我们提出的方法融合了高级特征和浅层特征,网络提取到的特征也就更加明显,所以模型分割效果更好. ...
... 表2中前6种方法采用了3D模型,这6种模型都是基于编码解码的结构,通过不断对深层特征的提取,定位ROI区域,与此同时也丢失了一部分浅层信息,导致平均Dice相似系数并没有本文提出的模型高,这表明,增加数据复杂度并不能有效提升模型分割性能,还需要加强模型对浅层信息的利用.表2中文献[8]和文献[23]都分别提出了一种二阶段的分割方法,第一阶段先大概定位胰腺肿瘤的区域,将第一阶段输出的结果当作第二阶段的输入,数据经过第二阶段的处理后得到最后的分割图,但是Dice相似系数并没有我们提出的模型高,可能是在模型的第一阶段没有定位到胰腺肿瘤的大概位置,将错误的信息传入第二阶段,导致了Dice相似系数降低.文献[25]中引入了RNN,使平均Dice相似系数在文献[24]的基础上提高了4%,文献[26]融合了Transformer和U-Net两个经典网络,但是分割效果都没有本文提出的模型效果好,可能是因为胰腺这类软组织在CT成像时对比度较差,从而对后续的实验产生了一定影响,而本研究采用的是T2加权序列图像,囊肿在该序列的信号更强,更加有利于网络对特征的提取,并且我们提出的方法融合了高级特征和浅层特征,网络提取到的特征也就更加明显,所以模型分割效果更好. ...
Recurrent saliency transformation network: Incorporating multi-stage visual cues for small organ segmentation
2
2018
... Comparison of Dice similarity coefficients between the model in different literature and the model in this paper
Table 2 | 模型 | 数据来源 | 数据类型 | Dice/% |
| VA-3DUNet[21] | NIH | 3D(CT) | 86.19±9.03 |
| Attention U-Net[9] | CT-150 | 3D(CT) | 84.00±8.70 |
| Cascaded multitask 3-D fully convolutional networks[11] | 82(CT) | 3D(CT) | 85.9±5.10 |
| HSSN[12] | MRI | 3D(MRI) | 85.7±2.3 |
| PanKNet[13] | Pancreas-MRI | 3D(MRI) | 77.46±8.62 |
| ConResNet[22] | NIH | 3D(CT) | 86.06 |
| PNet-MSA[8] | MRI-79 | 2D(MRI) | 80.7±7.40 |
| Spatial aggregation of holistically-nested convolutional neural networks[23] | NIH | 2D(CT) | 81.27±6.27 |
| deep convolutional LSTM neural network[24] | MRI-79 | 2D(MRI) | 80.5±6.70 |
| RSTN[25] | NIH | 2D(CT) | 84.50±4.97 |
| TransUnet[26] | MICCAI 2015 | 2D(CT) | 73.96 |
| DGANet(本文模型) | 长海医院 | 2D(MRI) | 86.28±2.77 |
表2中前6种方法采用了3D模型,这6种模型都是基于编码解码的结构,通过不断对深层特征的提取,定位ROI区域,与此同时也丢失了一部分浅层信息,导致平均Dice相似系数并没有本文提出的模型高,这表明,增加数据复杂度并不能有效提升模型分割性能,还需要加强模型对浅层信息的利用.表2中文献[8]和文献[23]都分别提出了一种二阶段的分割方法,第一阶段先大概定位胰腺肿瘤的区域,将第一阶段输出的结果当作第二阶段的输入,数据经过第二阶段的处理后得到最后的分割图,但是Dice相似系数并没有我们提出的模型高,可能是在模型的第一阶段没有定位到胰腺肿瘤的大概位置,将错误的信息传入第二阶段,导致了Dice相似系数降低.文献[25]中引入了RNN,使平均Dice相似系数在文献[24]的基础上提高了4%,文献[26]融合了Transformer和U-Net两个经典网络,但是分割效果都没有本文提出的模型效果好,可能是因为胰腺这类软组织在CT成像时对比度较差,从而对后续的实验产生了一定影响,而本研究采用的是T2加权序列图像,囊肿在该序列的信号更强,更加有利于网络对特征的提取,并且我们提出的方法融合了高级特征和浅层特征,网络提取到的特征也就更加明显,所以模型分割效果更好. ...
... 表2中前6种方法采用了3D模型,这6种模型都是基于编码解码的结构,通过不断对深层特征的提取,定位ROI区域,与此同时也丢失了一部分浅层信息,导致平均Dice相似系数并没有本文提出的模型高,这表明,增加数据复杂度并不能有效提升模型分割性能,还需要加强模型对浅层信息的利用.表2中文献[8]和文献[23]都分别提出了一种二阶段的分割方法,第一阶段先大概定位胰腺肿瘤的区域,将第一阶段输出的结果当作第二阶段的输入,数据经过第二阶段的处理后得到最后的分割图,但是Dice相似系数并没有我们提出的模型高,可能是在模型的第一阶段没有定位到胰腺肿瘤的大概位置,将错误的信息传入第二阶段,导致了Dice相似系数降低.文献[25]中引入了RNN,使平均Dice相似系数在文献[24]的基础上提高了4%,文献[26]融合了Transformer和U-Net两个经典网络,但是分割效果都没有本文提出的模型效果好,可能是因为胰腺这类软组织在CT成像时对比度较差,从而对后续的实验产生了一定影响,而本研究采用的是T2加权序列图像,囊肿在该序列的信号更强,更加有利于网络对特征的提取,并且我们提出的方法融合了高级特征和浅层特征,网络提取到的特征也就更加明显,所以模型分割效果更好. ...
Transunet: Transformers make strong encoders for medical image segmentation
2
2021
... Comparison of Dice similarity coefficients between the model in different literature and the model in this paper
Table 2 | 模型 | 数据来源 | 数据类型 | Dice/% |
| VA-3DUNet[21] | NIH | 3D(CT) | 86.19±9.03 |
| Attention U-Net[9] | CT-150 | 3D(CT) | 84.00±8.70 |
| Cascaded multitask 3-D fully convolutional networks[11] | 82(CT) | 3D(CT) | 85.9±5.10 |
| HSSN[12] | MRI | 3D(MRI) | 85.7±2.3 |
| PanKNet[13] | Pancreas-MRI | 3D(MRI) | 77.46±8.62 |
| ConResNet[22] | NIH | 3D(CT) | 86.06 |
| PNet-MSA[8] | MRI-79 | 2D(MRI) | 80.7±7.40 |
| Spatial aggregation of holistically-nested convolutional neural networks[23] | NIH | 2D(CT) | 81.27±6.27 |
| deep convolutional LSTM neural network[24] | MRI-79 | 2D(MRI) | 80.5±6.70 |
| RSTN[25] | NIH | 2D(CT) | 84.50±4.97 |
| TransUnet[26] | MICCAI 2015 | 2D(CT) | 73.96 |
| DGANet(本文模型) | 长海医院 | 2D(MRI) | 86.28±2.77 |
表2中前6种方法采用了3D模型,这6种模型都是基于编码解码的结构,通过不断对深层特征的提取,定位ROI区域,与此同时也丢失了一部分浅层信息,导致平均Dice相似系数并没有本文提出的模型高,这表明,增加数据复杂度并不能有效提升模型分割性能,还需要加强模型对浅层信息的利用.表2中文献[8]和文献[23]都分别提出了一种二阶段的分割方法,第一阶段先大概定位胰腺肿瘤的区域,将第一阶段输出的结果当作第二阶段的输入,数据经过第二阶段的处理后得到最后的分割图,但是Dice相似系数并没有我们提出的模型高,可能是在模型的第一阶段没有定位到胰腺肿瘤的大概位置,将错误的信息传入第二阶段,导致了Dice相似系数降低.文献[25]中引入了RNN,使平均Dice相似系数在文献[24]的基础上提高了4%,文献[26]融合了Transformer和U-Net两个经典网络,但是分割效果都没有本文提出的模型效果好,可能是因为胰腺这类软组织在CT成像时对比度较差,从而对后续的实验产生了一定影响,而本研究采用的是T2加权序列图像,囊肿在该序列的信号更强,更加有利于网络对特征的提取,并且我们提出的方法融合了高级特征和浅层特征,网络提取到的特征也就更加明显,所以模型分割效果更好. ...
... 表2中前6种方法采用了3D模型,这6种模型都是基于编码解码的结构,通过不断对深层特征的提取,定位ROI区域,与此同时也丢失了一部分浅层信息,导致平均Dice相似系数并没有本文提出的模型高,这表明,增加数据复杂度并不能有效提升模型分割性能,还需要加强模型对浅层信息的利用.表2中文献[8]和文献[23]都分别提出了一种二阶段的分割方法,第一阶段先大概定位胰腺肿瘤的区域,将第一阶段输出的结果当作第二阶段的输入,数据经过第二阶段的处理后得到最后的分割图,但是Dice相似系数并没有我们提出的模型高,可能是在模型的第一阶段没有定位到胰腺肿瘤的大概位置,将错误的信息传入第二阶段,导致了Dice相似系数降低.文献[25]中引入了RNN,使平均Dice相似系数在文献[24]的基础上提高了4%,文献[26]融合了Transformer和U-Net两个经典网络,但是分割效果都没有本文提出的模型效果好,可能是因为胰腺这类软组织在CT成像时对比度较差,从而对后续的实验产生了一定影响,而本研究采用的是T2加权序列图像,囊肿在该序列的信号更强,更加有利于网络对特征的提取,并且我们提出的方法融合了高级特征和浅层特征,网络提取到的特征也就更加明显,所以模型分割效果更好. ...
An approach for training data enrichment and batch labeling in AI+MRI aided diagnosis
1
2018
... 但是本研究还存在一定局限性.首先该研究只使用了单一模态数据进行实验,采用多模态融合可能会进一步提升模型性能,未来将考虑使用多模态数据进行训练来提升胰腺囊性肿瘤分割精度.其次,分割精度还有待提升,未来的研究可以对实验数据进行样本扩增[27]或者继续改善模型结构,来提高模型分割精度. ...
基于AI+MRI的影像诊断的样本增广与批量标注方法
1
2018
... 但是本研究还存在一定局限性.首先该研究只使用了单一模态数据进行实验,采用多模态融合可能会进一步提升模型性能,未来将考虑使用多模态数据进行训练来提升胰腺囊性肿瘤分割精度.其次,分割精度还有待提升,未来的研究可以对实验数据进行样本扩增[27]或者继续改善模型结构,来提高模型分割精度. ...