引言
医学影像检查为乳腺病变诊断提供了有效工具[3].然而,近年来随着乳腺影像检查数量不断攀升,临床医师的阅片压力激增,极易造成误诊、漏诊.乳腺癌计算机辅助诊断(computer aided diagnosis,CAD)技术应运而生.现有的乳腺癌CAD技术主要包括传统机器学习和深度学习两种方法.基于传统机器学习的方法依赖于精细地靶区人工勾画,以便于提取用于模型训练的特征[4, 5].而基于深度学习的方法能够实现高通量、自动化地感兴趣区域(region of interest,ROI)特征提取,并且对ROI精细勾画的依赖性较弱,在乳腺病变分类任务中得到了广泛应用[6-11].例如Samala团队[8]基于数字乳腺断层成像(digital breast tomosynthesis,DBT)和乳腺钼靶X射线成像,设计了一种多阶段迁移学习模型进行乳腺良恶性肿块的自动分类.他们利用了非医学图像(ImageNet)数据上训练的卷积神经网络(convolutional neural network,CNN)模型的知识,首先用具有相对大样本量的乳腺X射线数据进行微调,然后再迁移到基于小型DBT数据的目标任务中,以提高模型在小样本DBT影像数据上的学习效率和诊断准确率.该模型得到了平均0.93的受试者工作特征曲线下方面积(area under the curve,AUC)值,证明了当来自目标域的训练样本有限时,使用来自相似辅助域的数据进行额外的迁移学习是有利的.Dalmis等[9]通过训练DenseNet网络模型提取基于多参数的超快动态增强磁共振成像(dynamic contrast enhanced magnetic resonance imaging,DCE-MRI)的影像特征,实现了AUC为0.811的乳腺良恶性病灶分类准确率,表明超快DCE-MRI可对乳腺良恶性病变进行有效鉴别诊断.该团队进一步采用随机森林分类器对临床信息(患者信息、乳腺癌易感基因和表观弥散系数)进行特征提取,并将影像特征与临床特征相融合,显著提高了模型的分类性能(AUC提高至0.852).Saritas等[10]从钼靶X射线影像中提取影像特征,包括肿块形状、肿块边缘和肿块密度,并与乳腺影像数据报告和数据系统(breast imaging reporting and data system,BI-RADS)分级和年龄进行特征融合,而后输入人工神经网络(artificial neural network,ANN)进行乳腺病变良恶性的预测,从而辅助临床医生判断病人是否需要进行活检.
本研究提出了一种联合非对称卷积(asymmetric convolution,AC)和超轻子空间注意模块(ultra-lightweight subspace attention module,Ulsam)的AC_Ulsam_CNN网络模型进行乳腺病变良恶性的自动鉴别诊断.首先,该模型通过增加网络宽度和深度,并引入子空间注意力机制,从而能够增强网络的特征表达能力,提高模型的检测性能.其后,将DCE-MRI影像特征与其他临床指标进行特征融合,通过增加特征的多样性,以弥补因样本量少导致的分类精度低的缺陷,进而获得更准确可靠的分类结果.
1 实验部分
1.1 实验数据
本研究中的乳腺数据来自上海交通大学医学院附属新华医院.具体入组标准为:(1)根据BI-RADS[15],放射医师拟诊为患良性或恶性乳腺病变的患者(BI-RADS评估为0~Ⅴ级);(2)病灶最大截面直径≥5 mm;(3)于同一医院检查并进行手术或活检穿刺后取得病理结果;(4)术前影像检查包括DCE-MRI扫描,包括动态增强T1高分辨率各向同性容积采集序列(dyn-eTHRIVE,Dyn)扫描、动态增强T1高分辨率各向同性体积采集的减影序列(sdyn-eTHRIVE SENSE,Sdyn)扫描和扩散加权成像(diffusion-weighted imaging,DWI)扫描.自2014年1月至2019年12月间,符合数据入组条件的病例累计有277例女性患者,包括良性乳腺病变119例、恶性病变158例(表 1).恶性病变中浸润性癌占比最高,良性病变主要表现为纤维腺瘤和乳腺病.本研究设计和实施方案已获得由伦理委员会和合作医院机构审查签署的知情协议.所有参与者均签订书面知情同意书.
表1 乳腺病变良恶性数据分布
Table 1
| 良性病变 | 恶性病变 | |||
| 病变表现 | 病例数 | 病变表现 | 病例数 | |
| 纤维腺瘤 | 48 | 粘液癌 | 9 | |
| 乳腺病 | 43 | 导管原位癌 | 21 | |
| 导管内乳头状瘤 | 16 | 浸润性导管癌 | 28 | |
| 导管扩张 | 6 | 乳头状癌 | 9 | |
| 导管上皮增生 | 6 | 浸润性癌 | 91 | |
| 累计 | 119 | 累计 | 158 | |
MRI扫描均采用飞利浦Ingenia 3.0T超导型磁共振设备.在注射钆喷替酸葡甲胺(Gd-DTPA)对比剂前,进行一次平扫采集;注射对比剂后,再进行数次连续且不间断的重复性MRI扫描.对比剂经病患手背由高压注射器进行静脉团注,剂量为0.2 mmol/kg、流速为2.0 mL/s,之后使用相同方法注射15 mL浓度为0.9%的氯化钠水溶液,记录磁共振动态增强曲线(time intensity curve,TIC)类型.
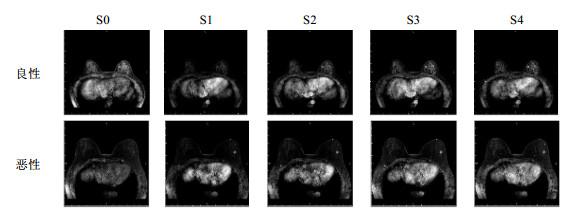
Dyn扫描设有5个时序,分别记为S0~S4(如图 1所示).Sdyn扫描设有4个时序,分别记为S0~S3.扫描层数均为150层,层厚为1.2 mm,扫描时间均为58 s,影像分辨率为512 mm×512 mm,通道数为3.DWI扫描设定扩散敏感系数b值为0.80 s/mm2,记录各被试的表现扩散系数(apparent diffusion coefficient,ADC).
图1
图1
设有5个时序(S0~S4)的Dyn扫描影像
Fig.1
The images acquired using Dyn with 5 time phase scans (S0~S4)
1.2 实验方法
本文提出的基于多模态MRI数据和深度学习的乳腺病变良恶性鉴别方法流程主要包括:(1)数据预处理;(2)基于DCE-MRI影像数据,采用迁移学习方法预训练模型,以从不同的时序中筛选出对乳腺病变良恶性鉴别最为有效的扫描时序.(3)基于最优扫描时序,构建针对乳腺病变良恶性鉴别任务的AC_Ulsam_CNN网络模型.(4)将影像特征与临床BI-RADS分级指标、ADC值和TIC类型进行特征融合,重新训练混合AC_Ulsam_CNN网络,从而进一步提高乳腺病变良恶性鉴别的准确率.
1.2.1 数据预处理
本文使用的数据模态包括DCE-MRI不同时序的扫描图像和其他衍生指标,如:BI-RADS分级指标、ADC值和TIC类型.
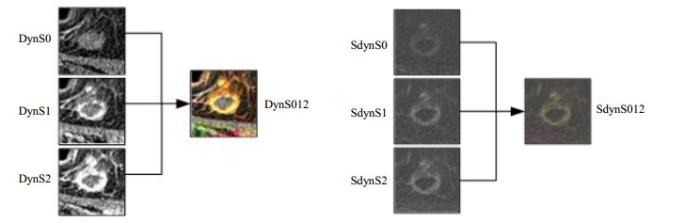
首先,对DCE-MRI影像数据进行预处理.根据临床医师勾画的金标准定位病灶位置,以病变区域为中心,设置大小为64*64的像素窗口,分割出包含病灶区域且占比适中的ROI图像,并保存为灰度图片,记为基于单一信源的ROI数据.为丰富ROI信息,分别选取时序相位扫描中的任意三个扫描进行通道融合.将融合后的图像保存为具有三通道的彩色图片,并记为基于多信源的ROI数据(如图 2所示).
图2
图2
DCE-MRI不同时序扫描的多信源组合ROI影像示意图
Fig.2
The multi-source combined ROI images scanned by different time phase scans of DCE-MRI
其次,对衍生指标中的临床BI-RADS分级和TIC评估数据分别进行量化处理.其中,根据临床BI-RADS分级结果,将对应分级(0级、Ⅰ级、Ⅱ级、Ⅲ级、Ⅳa级、Ⅳb级、Ⅳc级和Ⅴ级)量化为0到7,代表临床对病灶性质的初步筛查评估;将TIC评估中上升型、平台型和流出型分别量化为数值0、1和2;定量ADC值则进行线性归一化处理.
其中,X表示每个病例的ADC特征值,
最后,根据DCE-MRI中不同时序扫描及其随机排列组合,本实验共获得9组基于单一时序扫描的单信源影像数据和13组基于不同时序组合的多信源影像数据,如表 2所示.其中,每组均包含95 106张含乳腺病灶的ROI图像,包括良性病变31 722张、恶性病变63 384张.每张影像均有其对应的衍生指标,并且将临床病理检验得到的良恶性诊断结果作为其真实标签.
表2 DCE-MRI中不同时序扫描及其组合
Table 2
| 时序类别 | 时序相位名称 |
| 单一时序 | DynS0、DynS1、DynS2、DynS3、DynS4、SdynS0、SdynS1、SdynS2、SdynS3 |
| 组合时序 | DynS012、DynS013、DynS014、DynS023、DynS024、DynS034、DynS123、DynS124、 DynS134、DynS234、SdynS012、SdynS013、SdynS123 |
1.2.2 基于不同时序的DCE-MRI对乳腺病变良恶性的分类
DCE-MRI扫描中包含多个不同时序的扫描序列,而不同时序扫描的影像对良恶性病灶的组织对比度强度具有不同的敏感性.因此,为筛选出对病灶良恶性鉴别最为有效的扫描序列,本文首先进行了初步的预实验.然而,考虑到本实验中样本量不足,若直接从头训练模型,容易造成模型欠拟合或过拟合问题,难以进行最优时序的筛选.迁移学习为解决小样本量医学影像数据提供了有效方法[16].其中,InceptionV3模型因具有较强的特征表达能力被广泛应用于各项分类任务[17],并且已在乳房肿块检测研究[18-20]中取得了优异的成果.与其他CNN相比,InceptionV3计算成本和内存需求低,对有限数据集进行迁移学习的可行性高,因此本研究中采用经典的InceptionV3作为预训练模型.通过去除顶层分类器,并添加两个输出尺寸分别为128和16的Dense层进行模型微调,最后采用Sigmoid层作为模型的输出层,以评估基于不同时序的DCE-MRI对乳腺病变良恶性的分类性能.
1.2.3 基于AC_Ulsam_CNN网络的乳腺病变良恶性鉴别模型
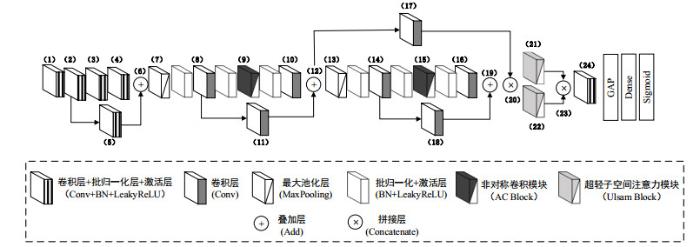
基于迁移学习的方法往往依赖于目标数据与原始数据之间的相似性,而针对高维复杂的MRI数据,该方法在模型精度提升方面仍具有一定局限性.因此,为进一步提高DCE-MRI影像数据对乳腺病变良恶性鉴别性能,基于筛选出的最优时序作为输入图像,本文构建了基于AC_Ulsam_CNN网络的模型.如图 3所示,AC_Ulsam_CNN网络主要由两个非对称卷积模块(AC Block)、两个超轻子空间注意模块(Ulsam Block)、两个最大池化层(MaxPooling)和四个残差式学习跳跃连接结构(分别为:(2)→(5)→(6),(8)→(11)→(12),(12)→(17)→(20),(14)→(18)→(19))组成,而后经过全局平均池化层(global average polling,GAP)、密集全连接层(Dense)和分类器层(Sigmoid)得到输出结果.网络结构参数设置如表 3所示.
图3
表3 AC_Ulsam_CNN网络结构参数设置
Table 3
| # | 输入层 | 输入尺寸 | 卷积核尺寸 | 通道数 | 步长 | 输出尺寸 |
| (1) | Conv1+BN+LeakyReLU | 150 × 150 × 3 | 3 × 3 | 16 | 1 | 150 × 150 × 16 |
| (2) | Conv2+BN+LeakyReLU | 150 × 150 × 16 | 1 × 1 | 8 | 1 | 150 × 150 × 8 |
| (3) | Conv3+BN+LeakyReLU | 150 × 150 × 8 | 3 × 3 | 8 | 1 | 150 × 150 × 8 |
| (4) | Conv4+BN+LeakyReLU | 150 × 150 × 8 | 1 × 1 | 16 | 1 | 150 × 150 × 16 |
| (5) | Conv5+BN+LeakyReLU | 150 × 150 × 16 | 1 × 1 | 16 | 1 | 150 × 150 × 16 |
| (6) | Add1 | 150 × 150 × 16 | - | - | - | 150 × 150 × 16 |
| (7) | Pool1 | 150 × 150 × 16 | 3 × 3 | - | 3 | 50 × 50 × 16 |
| (8) | Conv6 | 50 × 50 × 16 | 1 × 1 | 8 | 1 | 50 × 50 × 8 |
| (9) | AC Block1 | 50 × 50 × 8 | - | 8 | - | 50 × 50 × 16 |
| (10) | Conv7 | 50 × 50 × 16 | 1 × 1 | 32 | 1 | 50 × 50 × 32 |
| (11) | Conv8 | 50 × 50 × 16 | 1 × 1 | 32 | 1 | 50 × 50 × 32 |
| (12) | Add2 | 50 × 50 × 32 | - | - | - | 50 × 50 × 32 |
| (13) | Pool2 | 50 × 50 × 32 | 3 × 3 | - | 3 | 16 × 16 × 32 |
| (14) | Conv9 | 16 × 16 × 32 | 1 × 1 | 32 | 1 | 16 × 16 × 32 |
| (15) | AC Block2 | 16 × 16 × 32 | - | 32 | - | 16 × 16 × 64 |
| (16) | Conv10 | 16 × 16 × 64 | 1 × 1 | 128 | 1 | 16 × 16 × 128 |
| (17) | Conv11 | 50 × 50 × 32 | 3 × 3 | 16 | 3 | 16 × 16 × 16 |
| (18) | Conv12 | 16 × 16 × 32 | 1 × 1 | 128 | 1 | 16 × 16 × 128 |
| (19) | Add3 | 16 × 16 × 128 | - | - | - | 16 × 16 × 128 |
| (20) | Concatenate1 | 16 × 16 × 128 16 × 16 × 16 | - | - | - | 16 × 16 × 144 |
| (21) | Ulsam Block1 | 16 × 16 × 144 | - | - | - | 16 × 16 × 144 |
| (22) | Ulsam Block2 | 16 × 16 × 144 | - | - | - | 16 × 16 × 144 |
| (23) | Concatenate2 | 16 × 16 × 144 16 × 16 × 144 | - | - | - | 16 × 16 × 288 |
| (24) | Conv13+BN+LeakyReLU | 16 × 16 × 288 | 1 × 1 | 128 | 1 | 16 × 16 × 128 |
| - | GAP | 16 × 16 × 128 | - | - | - | 4096 |
| - | Dense | 4096 | - | - | - | 16 |
| - | Sigmoid | 16 | - | - | - | 1 |
Conv:卷积层,BN:批归一化层,AC:非对称卷积,Ulsam:超轻子空间注意力模块;GAP:全局平均池化层;Dense:密集全连接层,Add:叠加层,Pool:池化层,Concatenate:拼接层
此外,为解决随网络结构的加深、梯度优化速度减缓、模型收敛慢等问题,模型中引入了批归一化层(batch normalization,BN)和LeakyReLU激活函数,并采用Sigmoid函数进行激活输出,以加快网络优化速度,并提高模型的鲁棒性.
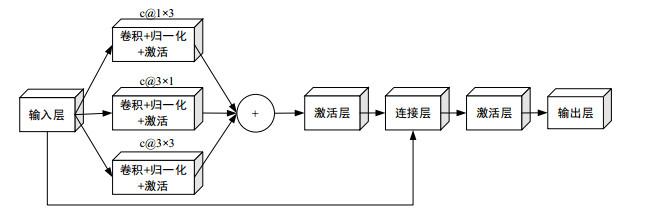
非对称卷积模块(AC Block)是一种采用多个非对称卷积核组合形式替换标准卷积核的特殊卷积结构,通过增加模型不同尺度方向的特征提取通路,从而提升模型的鲁棒性[21].非对称卷积参数训练后,与标准卷积参数的有效融合,也使得模型在训练阶段能够更专注于特征提取的强化,得到高表征能力、强相关的特征.虽然训练时间成本有提升,但卷积核参数的等效融合不会出现多余的计算量.因此,模型的性能提升无需多余的计算开支,该机制对解决因小样本量数据而导致的模型过拟合问题具有充分的应用价值.本文设计的非对称卷积模块结构如图 4所示.首先,将上一层得到的特征图分别输入到尺寸为1×3、3×1和3×3的卷积核(c@1×3、c@3×1和c@3×3,其中c为卷积通道数)中进行训练,并采用逐点求和方式进行融合,以提升局部显著性特征的影响因子.而后采用残差学习方式,将融合后的特征图与原特征图进行按通道拼接,从而避免图像信息丢失,增强网络的特征表达能力.
图4
图4
非对称卷积模块(AC Block)结构示意图
Fig.4
The structure diagram of asymmetric convolution module (AC Block)
图5
图5
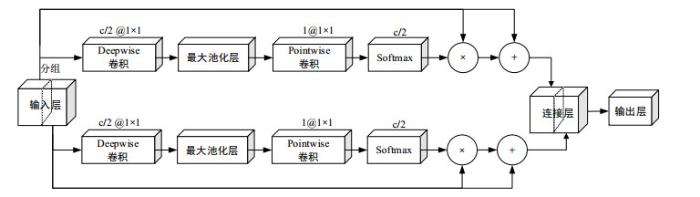
超轻子空间注意模块(Ulsam Block)结构示意图
Fig.5
The structure diagram of ultra-lightweight subspace attention module (Ulsam Block)
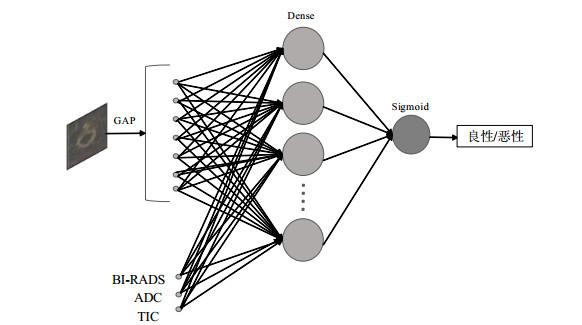
1.2.4 融合多模态数据的乳腺病变良恶性鉴别
DCE-MRI扫描能够生成TIC,为鉴别病变性质提供了血流动力学参数[23].ADC作为DWI成像中的一种衍生指标,能够反应软组织内水分子扩散情况,从而有效降低乳腺MRI的假阳性,并被证明与乳腺良恶性程度具有强相关性[24].因此,为进一步探索多模态数据融合在乳腺病变良恶性鉴别中的价值,本文设计了一种混合式AC_Ulsam_CNN卷积神经网络模型,通过将DCE-MRI影像特征分别与其他衍生指标(临床BI-RADS分级、ADC值和TIC类型)进行特征级别的融合,即在图 3网络全连接层部分,引入相应衍生指标特征,继而将融合后的新特征输入到Sigmoid分类器中进行良恶性分类(图 6).在此基础上,我们还将文献[6]和文献[8]中所设计的经典模型应用于本研究数据中,以进行横向对比实验,进一步验证所提模型的有效性.其中,文献[6]所提模型为一种简单线性堆叠卷积神经网络,由三种尺寸大小为11×11、5×5和3×3的卷积核和尺寸分别为2 048、1 024的全连接层组成,通道数从初始的64增加到512.文献[8]在网络层方面相较文献[6]多引入了批量归一化层,设计了一个10层CNN,包含尺寸大小为3×3的卷积核和大小分别为4 096、1 000、100个神经元的全连接层,通道数从初始的64增加到384又缩小至256.研究表明这两种网络结构在乳腺肿瘤良恶性分类任务中均表现出优异性能.
图6
图6
基于多模态数据融合的混合神经网络结构示意图
Fig.6
The structure diagram of hybrid neural network based on multimodal data fusion
1.3 模型评估
受数据集样本量大小限制,本实验采用五折交叉验证方法对各模型性能进行综合评估,并对每折中的训练集进行平移、旋转和镜像翻转的数据扩增处理,以减小模型过拟合.此外,考虑到本实验数据中良恶性病变具有类间不平衡问题,在网络损失函数计算部分,我们根据正负样本比例赋予类别权重因子,从而使模型结果不会偏向多数类别.采用准确率(accuracy,ACC)、灵敏度(sensitivity,Sens)、特异性(specificity,Spec),AUC指标进行模型性能评估.计算公式如下:
其中,TP表示恶性乳腺病变被正确分类的样本数;FP表示良性乳腺病变被错误分类的样本数;TN表示良性乳腺病变被正确分类的样本数;FN表示恶性乳腺病变被错误分类的样本数.
最后,采用IBM SPSS(v25.0)进行统计学分析,对不同模型的AUC和ACC进行比较.双尾t检验p < 0.05表示统计学意义.
1.4 实验设置
在硬件配置为计算机芯片Intel Core(TMi7-8700),其内存为32 GB,NVIDIA显卡GeForce GTX 1080ti,软件环境为Windows10系统,Nvidia显卡驱动CUDA9.0,以及用于深度神经网络的GPU加速库CUDNN7.0的实验配置下,本文基于Python和Keras框架进行网络模型开发.采用Adam优化器进行超参数优化,设置各卷积层初始化方式为Xavier,网络学习率为0.002,批处理大小为32,总训练次数至少200个epochs.
2 结果与讨论
2.1 基于不同时序的DCE-MRI对乳腺病变良恶性分类结果
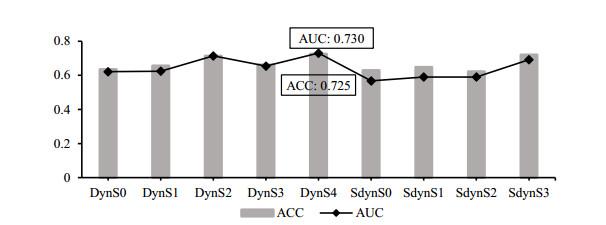
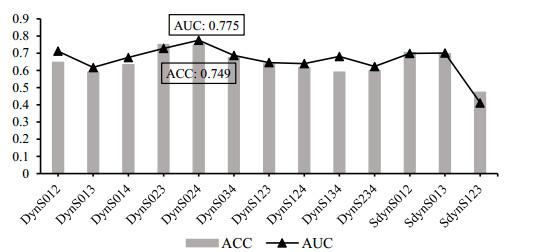
本研究分别对9个基于单一时序和13个基于不同时序扫描的多信源组合构建迁移学习模型,进行乳腺良恶性分类预实验.控制每次实验均是在相同的数据划分下基于相同InceptionV3网络结构完成训练.基于不同时序的五折交叉验证后的平均分类性能如图 7和图 8所示.其中,在基于各单一时序扫描中,基于DynS4时序扫描具有最好的良恶性鉴别能力,AUC为0.730,准确率ACC为0.725.而在组合序列中,基于DynS024的组合序列对乳腺良恶性鉴别的AUC和准确率ACC可达到0.775和0.749,高于基于单一时序的分类结果,这可能与单一时序中DynS2和DynS4具有较好的分类表现有关.因此,基于DynS4时序扫描和DynS024组合扫描序列对解决乳腺病变良恶性鉴别问题可能最具代表性,能够更好地反映恶性病灶与良性病灶的纹理、形态等特征的差异性.
图7
图7
基于单一时序扫描的乳腺病变良恶性分类结果
Fig.7
Differentiation results of benign and malignant breast lesions based on single-time phase scans
图8
图8
基于不同时序扫描的多信源组合的乳腺病变良恶性分类结果
Fig.8
Differentiation results of benign and malignant breast lesions based on multi-source combination of different time phase scans
2.2 基于AC_Ulsam_CNN网络模型的乳腺病变良恶性分类结果
如表 4所示,本实验所设计AC_Ulsam_CNN网络模型在基于最优DCE-MRI扫描序列下的乳腺良恶性鉴别性能有了进一步提升.其中,在DynS024组合序列下,模型的平均AUC可达0.826,比基于InceptionV3模型提高了0.051(p = 0.027),准确率提升了0.013(p = 0.047),特异性提升了0.094.虽然在灵敏度指标上,所提模型略低于InceptionV3模型,但其整体上对良性病变的鉴别能力与对恶性病变的鉴别能力相当,不存在偏倚,表明本文所提模型的有效性及可行性.
表4 基于InceptionV3网络与AC_Ulsam_CNN网络模型的分类结果对比
Table 4
| 网络模型结构 | 时序 | ACC | AUC | Sens | Spec |
| InceptionV3 | DynS4 | 0.725 | 0.730 | 0.763 | 0.620 |
| DynS024 | 0.749 | 0.775 | 0.789 | 0.664 | |
| AC_Ulsam_CNN | DynS4 | 0.771 | 0.784 | 0.733 | 0.790 |
| DynS024 | 0.762 | 0.826 | 0.783 | 0.758 |
2.3 基于多模态融合的乳腺病变良恶性分类
多模态数据融合能够为乳腺病变诊断提供多维度、综合性的病理信息,从而进一步提高其鉴别准确性.如表 5所示,在基于不同模态特征融合的消融实验中,我们发现各特征融合队列均可获得比单一DCE-MRI模型具有更高的分类性能.其中,基于四种特征的整体融合模型进行良恶性病灶鉴别的平均ACC达到了0.826,相比基于单一DynS024特征的模型分类准确率提升了0.064(p = 0.009),AUC值提升了0.051(p = 0.021),突出了多模态数据融合的优越性.
表5 基于多模态特征融合的消融实验结果
Table 5
| 特征类型 | ACC | AUC | Sens | Spec |
| DynS024 | 0.762 | 0.826 | 0.783 | 0.758 |
| DynS024+BI-RADS | 0.769 | 0.840 | 0.791 | 0.754 |
| DynS024+ADC | 0.807 | 0.863 | 0.822 | 0.792 |
| DynS024+TIC | 0.778 | 0.829 | 0.810 | 0.746 |
| DynS024+BI-RADS+ADC+TIC | 0.826 | 0.877 | 0.861 | 0.783 |
表6 本文方法与其他方法的分类结果对比
Table 6
3 结论
本研究构建了一种针对乳腺病变良恶性鉴别诊断的AC_Ulsam_CNN网络模型,通过多尺度特征提取并引入子空间注意力机制,增强了网络的特征表达能力及其鲁棒性.实验表明,在基于DynS024组合的DCE-MRI影像上,所提模型能够实现乳腺良恶性病变分类准确率为0.762.当模型进一步纳入临床BI-RADS分级、ADC值以及TIC类型等特征指标后,基于多模态特征融合后的模型分类准确率可达0.826,显著高于基于单独的DCE-MRI影像特征模型.因此,临床BI-RADS分级、ADC值和TIC类型对基于DCE-MRI进行乳腺诊断的性能提升具有显著贡献,而多模态特征的融合也为CAD提供了多维度、综合性的信息,从而进一步提高病灶的检测精度,有效辅助临床医生对乳腺病变的诊断.未来研究应进一步扩充乳腺病变数据量及模态的多样性,提高模型泛化能力.
无
参考文献
Pathological complete response and long-term clinical benefit in breast cancer: the CTNeoBC pooled analysis
[J].DOI:10.1016/S0140-6736(13)62422-8 [本文引用: 1]
乳腺良性病变的组织学分型及其乳腺癌风险
[J].
Histologic types of benign breast disease and the risk of breast cancer
[J].
Breast image analysis for risk assessment, detection, diagnosis, and treatment of cancer
[J].DOI:10.1146/annurev-bioeng-071812-152416 [本文引用: 1]
Machine learning with multiparametric magnetic resonance imaging of the breast for early prediction of response to neoadjuvant chemotherapy
[J].DOI:10.1016/j.breast.2019.11.009 [本文引用: 1]
A radiomics approach with CNN for shear-wave elastography breast tumor classification
[J].DOI:10.1109/TBME.2018.2844188 [本文引用: 6]
Computer-aided diagnosis system for breast ultrasound images using deep learning
[J].
Breast cancer diagnosis in digital breast tomosynthesis: Effects of training sample size on multi-stage transfer learning using deep neural nets
[J].
Artificial intelligence-based classification of breast lesions imaged with a multiparametric breast mri protocol with ultrafast DCE-MRI, T2, and DWI
[J].DOI:10.1097/RLI.0000000000000544 [本文引用: 1]
Prediction of breast cancer using artificial neural networks
[J].DOI:10.1007/s10916-011-9768-0 [本文引用: 1]
基于nnU-Net的乳腺DCE-MR图像中乳房和腺体自动分割
[J].
Automatic segmentation of breast and fibroglandular tissues in DCE-MR images based on nnU-Net
[J].
A deep feature fusion methodology for breast cancer diagnosis demonstrated on three imaging modality datasets
[J].DOI:10.1002/mp.12453 [本文引用: 1]
基于级联卷积神经网络的前列腺磁共振图像分类
[J].
Prostate cancer diagnosis based on cascaded convolutional neural networks
[J].
MR动态增强显像在乳腺肿瘤良恶性鉴别诊断中的价值分析
[J].
Value analysis of MR dynamic enhanced imaging in differential diagnosis of benign and malignant breast tumors
[J].
Modified bi-rads scoring of breast imaging findings improves clinical judgment
[J].DOI:10.1111/tbj.12492 [本文引用: 1]
A survey on transfer learning
[J].
Detection of masses in mammographic images using geometry, simpson's diversity index and SVM
[J].
Digital mammographic tumor classification using transfer learning from deep convolutional neural networks
[J].
应用计算机辅助诊断技术评价血流动力学特征在乳腺MRI中的价值
[J].
Values of kinetic features measured by computer-aided diagnosis for breast MRI
[J].
Differentiation of clinically benign and malignant breast lesions using diffusion-weighted imaging
[J].