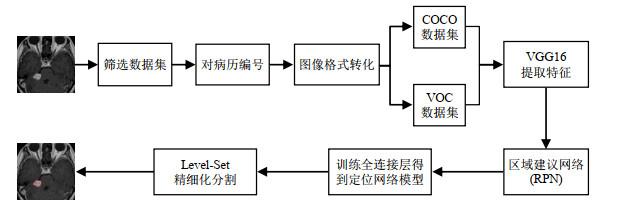
图1
本文的实验流程
Fig.1
The experimental process in this research
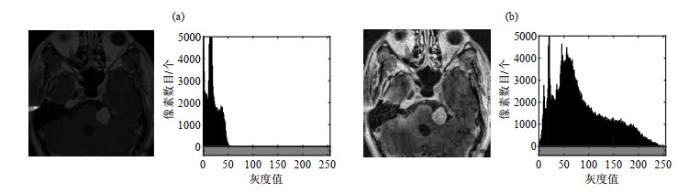
图2
(a) 预处理前的磁共振图像(左)及其灰度直方图(右);(b)经CLAHE预处理后的图像(左)及其灰度直方图(右)
Fig.2
(a) The magnetic resonance image before pre-processing (left) and its gray histogram (right); (b) The magnetic resonance image after CLAHE algorithm pre-processing and its gray histogram (right)
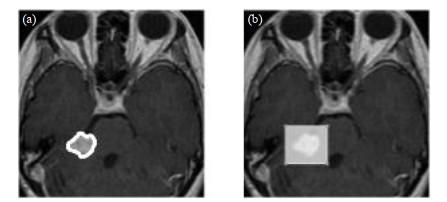
图3
(a) 符合COCO格式的轮廓勾画图;(b) 符合VOC格式的位置图
Fig.3
(a) COCO format image; (b) VOC format image
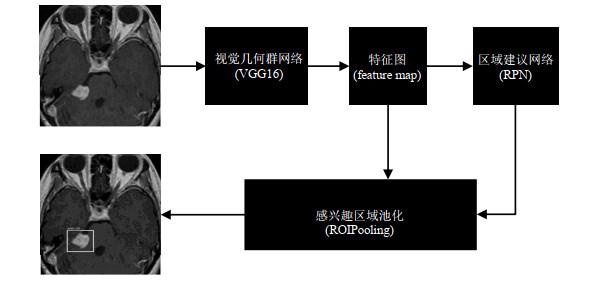
图4
Faster-RCNN网络结构图
Fig.4
Network structure of Faster-RCNN
图5
使用本文提出的方法不同迭代轮次CPA肿瘤的分割结果
Fig.5
Segmentation result of CPA tumor in different iterations using the proposed method
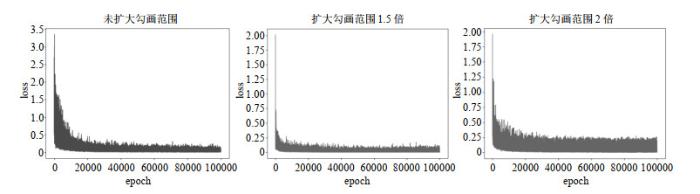
图6
模型于不同勾画范围下训练100 000轮的损失曲线
Fig.6
Loss curves of model under different delineated areas after 100 000 iterations
图7
不同勾画范围下,使用本文方法对桥小脑角区(CPA)肿瘤进行分割的结果
Fig.7
Segmentations of CPA tumor under different delineated areas with the proposed method
[1]
PING X X , MENG Q , TIAN X , et al . MRI findings of lesions in the cerebellopontine angle
[J]. J Med Imaging , 2014 , 24 (1 ): 12 - 15 .
DOI:10.3969/j.issn.1005-5185.2014.01.004
[本文引用: 1]
平小夏 , 孟倩 , 田霞 , 等 . 桥小脑角区病变的MRI表现
[J]. 医学影像学杂志, 2014 , 24 (1 ): 12 - 15 .
DOI:10.3969/j.issn.1005-5185.2014.01.004
[本文引用: 1]
[2]
FANG F , HU S P . Comparative analysis of MRI findings and pathology in acoustic neuroma
[J]. CT Theory and Applications , 2019 , 28 (6 ): 731 - 738 .
URL
[本文引用: 1]
方芳 , 胡少平 . 听神经瘤MRI表现与病理对照分析
[J]. CT理论与应用研究, 2019 , 28 (6 ): 731 - 738 .
URL
[本文引用: 1]
[4]
ZHANG J Q , ZHANG Y , YIN Y , et al . A review of machine learning in tumor radio therapy
[J]. Journal of Biomedical Engineering , 2019 , 36 (5 ): 879 - 844 .
URL
[本文引用: 1]
张珺倩 , 张远 , 尹勇 , 等 . 机器学习在肿瘤放射治疗领域应用进展
[J]. 生物医学工程学杂志, 2019 , 36 (5 ): 879 - 884 .
URL
[本文引用: 1]
[5]
LI Q , BAI K X , ZHAO L , et al . Progresss and challenges of MRI brain tumor image segmentation
[J]. Journal of Image and Graphics , 2020 , 25 (3 ): 419 - 431 .
URL
[本文引用: 1]
李锵 , 白柯鑫 , 赵柳 , 等 . MRI脑肿瘤图像分割研究进展及挑战
[J]. 中国图象图形学报, 2020 , 25 (3 ): 419 - 431 .
URL
[本文引用: 1]
[6]
WANG C F , WANG B J , WANG Z , et al . The target profile of multimodal magnetic resonance imaging in the radiotherapy of craniocerebral malignant tumor
[J]. Chinese Journal of Practical Nervous Diseases , 2018 , 21 (19 ): 2118 - 2124 .
DOI:10.12083/SYSJ.2018.19.462
[本文引用: 1]
王长福 , 王斌杰 , 王智 , 等 . 多模态磁共振成像及融合技术在颅脑恶性肿瘤放疗靶区勾画中的应用
[J]. 中国实用神经疾病杂志, 2018 , 21 (19 ): 2118 - 2124 .
DOI:10.12083/SYSJ.2018.19.462
[本文引用: 1]
[7]
ZIKIC D, IOANNOU Y, BROWN M, et al. Segmentation of brain tumor tissues with convolutional neural networks[C]. Proceedings MICCAI-BRATS, 2014: 36-39.
[本文引用: 1]
[8]
JIA Y, SHELHAMER E, DONAHUE J, et al. Caffe: Convolutional architecture for fast feature embedding[C]//Proceedings of the 22nd ACM international conference on Multimedia. 2014: 675-678.
[本文引用: 1]
[9]
IQBAL S , GHANI M U , SABA T , et al . Brain tumor segmentation in multi-spectral MRI using convolutional neural networks (CNN)
[J]. Microsc Res Techniq, 2018 , 81 (4 ): 419 - 427 .
DOI:10.1002/jemt.22994
[本文引用: 1]
[10]
EZHILARASI R, VARALAKSHMI P. Tumor detection in the brain using faster R-CNN[C]//20182nd International Conference on I-SMAC (IoT in Social, Mobile, Analytics and Cloud). IEEE, 2018: 388-392.
[本文引用: 1]
[12]
THILLAIKKARASI R , SARAVANAN S . An enhancement of deep learning algorithm for brain tumor segmentation using kernel based CNN with M-SVM
[J]. J Med Syst, 2019 , 43 (4 ): 1 - 7 .
DOI:10.1007/s10916-019-1223-7
[本文引用: 1]
[13]
IQBAL S , GHANI KHAN M U , SABA T , et al . Deep learning model integrating features and novel classifiers fusion for brain tumor segmentation
[J]. Microsc Res Techniq, 2019 , 82 (8 ): 1302 - 1315 .
DOI:10.1002/jemt.23281
[本文引用: 1]
[14]
KASS M , WITKIN A , TERZOPOULOS D . Snakes: Active contour models
[J]. Int J Comput Vision, 1988 , 1 (4 ): 321 - 331 .
DOI:10.1007/BF00133570
[本文引用: 1]
[15]
CHAN T F , VESE L A . Active contours without edges
[J]. IEEE T Image Process, 2001 , 10 (2 ): 266 - 277 .
DOI:10.1109/83.902291
[本文引用: 1]
[16]
LI C M , KAO C Y , GORE J C , et al . Minimization of region-scalable fitting energy for image segmentation
[J]. IEEE T Image Process, 2008 , 17 (10 ): 1940 - 1949 .
DOI:10.1109/TIP.2008.2002304
[本文引用: 4]
[17]
YIN S L , LI H , LIU D S , et al . Active contour modal based on density-oriented BIRCH clustering method for medical image segmentation
[J]. Multimed Tools Appl, 2020 , 79 (41 ): 31049 - 31068 .
[本文引用: 1]
[18]
YUAN C A , ZHENG Y , QIN X , et al . The medical image segmentation method of improved RSF active contour model
[J]. Journal of Zhengzhou University (Natural Science Edition) , 2017 , 49 (1 ): 34 - 38, 44 .
URL
[本文引用: 1]
元昌安 , 郑彦 , 覃晓 , 等 . 改进RSF主动轮廓模型的医学图像分割方法
[J]. 郑州大学学报(理学版), 2017 , 49 (1 ): 34 - 38 .
URL
[本文引用: 1]
[19]
SHI Q Y , YAN F , YANG Y , et al . Image segmentation of tooth and alveolar bone with the level set model
[J]. Chinese J Magn Reson , 2021 , 38 (2 ): 182 - 193 .
URL
[本文引用: 1]
石沁祎 , 闫方 , 杨阳 , 等 . 基于水平集的牙齿牙槽骨图像分割
[J]. 波谱学杂志, 2021 , 38 (2 ): 182 - 193 .
URL
[本文引用: 1]
[20]
ZHANG D, ZHU W, ZHAO H, et al. Automatic localization and segmentation of optical disk based on faster R-CNN and level set in fundus image[C]//Medical Imaging 2018: Image Processing. International Society for Optics and Photonics, 2018, 10574: 105741U.
[本文引用: 1]
[21]
ZUO J H , ZHAO C , ZHU X L , et al . High-resolution remote sensing image building extraction combined with Faster-RCNN and Level-Set
[J]. Chinese Journal of Liquid Crystals and Displays , 2019 , 34 (4 ): 439 - 447 .
URL
[本文引用: 1]
左俊皓 , 赵聪 , 朱晓龙 , 等 . Faster-RCNN和Level-Set结合的高分遥感影像建筑物提取
[J]. 液晶与显示, 2019 , 34 (4 ): 439 - 447 .
URL
[本文引用: 1]
[22]
LIU Y , CHEN J C , HU X Y , et al . Classification and localization of acoustic neuroma and meningioma in the cerebellopontine angle based on Mask RCNN
[J]. Chinese J Magn Reson , 2021 , 38 (1 ): 58 - 68 .
URL
[本文引用: 1]
刘颖 , 陈静聪 , 胡小洋 , 等 . 基于Mask RCNN的桥小脑角区听神经瘤与脑膜瘤分类定位研究
[J]. 波谱学杂志, 2021 , 38 (1 ): 58 - 68 .
URL
[本文引用: 1]
[23]
REN S Q, HE K M, GIRSHICK R, et al. Faster R-CNN: Towards real-time object detection with region proposal networks[C]//Advances in neural information processing systems. 2015: 91-99.
[本文引用: 1]
[24]
OSHER S , SETHIAN J A . Fronts propagating with curvature-dependent speed: algorithms based on Hamilton-Jacobi formulations
[J]. J Comput Phys, 1988 , 79 (1 ): 12 - 49 .
DOI:10.1016/0021-9991(88)90002-2
[本文引用: 1]
[26]
THAHA M M , KUMAR K P M , MURUGAN B S , et al . Brain tumor segmentation using convolutional neural networks in MRI images
[J]. J Med Syst, 2019 , 43 (9 ): 1 - 10 .
[本文引用: 2]
[27]
GE T , ZHAN T M , MU S X . Brain tumor segmentation algorithm based on multi-kernel collaborative representation classification
[J]. Journal of Nanjing University of Science and Technology , 2019 , 43 (5 ): 578 - 585 .
URL
[本文引用: 2]
葛婷 , 詹天明 , 牟善祥 . 基于多核协同表示分类的脑肿瘤分割算法
[J]. 南京理工大学学报, 2019 , 43 (5 ): 578 - 585 .
URL
[本文引用: 2]
[28]
RONNEBERGER O, FISCHER P, BROX T. U-net: Convolutional networks for biomedical image segmentation[C]//International Conference on Medical image computing and computer-assisted intervention. Springer, Cham, 2015: 234-241.
[本文引用: 2]
[29]
XU Z W, WU Z Y, FENG J J. CFUN: combining faster R-CNN and U-net network for efficient whole heart segmentation[J]. arXiv preprint arXiv: 1812.04914, 2018.
[本文引用: 3]
桥小脑角区病变的MRI表现
1
2014
... 对病灶的精准分割是医学图像研究领域的关键之一,本文主要研究桥小脑角区(cerebellopontine angle,CPA)的肿瘤分割.CPA是中枢神经系统肿瘤的好发部位之一,约占颅内占位性病变的6%~10%[1 ] .CPA肿瘤包括听神经瘤、脑膜瘤、胆脂瘤、神经鞘瘤、血管性病变瘤等等,其中听神经瘤和脑膜瘤是最为常见的两种CPA肿瘤.听神经瘤起源于听神经前庭,约占CPA肿瘤的85%,占颅内肿瘤的6%[2 ] .脑膜瘤为颅内常见的肿瘤之一,约占颅内原发性肿瘤的15%~20%[3 ] .目前治疗CPA肿瘤的方法主要包括手术疗法、放射治疗、化疗.而在手术切除和放疗过程中,CPA肿瘤位置和轮廓的确定对手术和放疗的成功与否都影响极大.由于不同器官对放疗处方剂量的耐受度不同,因此器官分割的精准性将直接影响放疗计划设计和放疗疗效[4 ] . ...
桥小脑角区病变的MRI表现
1
2014
... 对病灶的精准分割是医学图像研究领域的关键之一,本文主要研究桥小脑角区(cerebellopontine angle,CPA)的肿瘤分割.CPA是中枢神经系统肿瘤的好发部位之一,约占颅内占位性病变的6%~10%[1 ] .CPA肿瘤包括听神经瘤、脑膜瘤、胆脂瘤、神经鞘瘤、血管性病变瘤等等,其中听神经瘤和脑膜瘤是最为常见的两种CPA肿瘤.听神经瘤起源于听神经前庭,约占CPA肿瘤的85%,占颅内肿瘤的6%[2 ] .脑膜瘤为颅内常见的肿瘤之一,约占颅内原发性肿瘤的15%~20%[3 ] .目前治疗CPA肿瘤的方法主要包括手术疗法、放射治疗、化疗.而在手术切除和放疗过程中,CPA肿瘤位置和轮廓的确定对手术和放疗的成功与否都影响极大.由于不同器官对放疗处方剂量的耐受度不同,因此器官分割的精准性将直接影响放疗计划设计和放疗疗效[4 ] . ...
听神经瘤MRI表现与病理对照分析
1
2019
... 对病灶的精准分割是医学图像研究领域的关键之一,本文主要研究桥小脑角区(cerebellopontine angle,CPA)的肿瘤分割.CPA是中枢神经系统肿瘤的好发部位之一,约占颅内占位性病变的6%~10%[1 ] .CPA肿瘤包括听神经瘤、脑膜瘤、胆脂瘤、神经鞘瘤、血管性病变瘤等等,其中听神经瘤和脑膜瘤是最为常见的两种CPA肿瘤.听神经瘤起源于听神经前庭,约占CPA肿瘤的85%,占颅内肿瘤的6%[2 ] .脑膜瘤为颅内常见的肿瘤之一,约占颅内原发性肿瘤的15%~20%[3 ] .目前治疗CPA肿瘤的方法主要包括手术疗法、放射治疗、化疗.而在手术切除和放疗过程中,CPA肿瘤位置和轮廓的确定对手术和放疗的成功与否都影响极大.由于不同器官对放疗处方剂量的耐受度不同,因此器官分割的精准性将直接影响放疗计划设计和放疗疗效[4 ] . ...
听神经瘤MRI表现与病理对照分析
1
2019
... 对病灶的精准分割是医学图像研究领域的关键之一,本文主要研究桥小脑角区(cerebellopontine angle,CPA)的肿瘤分割.CPA是中枢神经系统肿瘤的好发部位之一,约占颅内占位性病变的6%~10%[1 ] .CPA肿瘤包括听神经瘤、脑膜瘤、胆脂瘤、神经鞘瘤、血管性病变瘤等等,其中听神经瘤和脑膜瘤是最为常见的两种CPA肿瘤.听神经瘤起源于听神经前庭,约占CPA肿瘤的85%,占颅内肿瘤的6%[2 ] .脑膜瘤为颅内常见的肿瘤之一,约占颅内原发性肿瘤的15%~20%[3 ] .目前治疗CPA肿瘤的方法主要包括手术疗法、放射治疗、化疗.而在手术切除和放疗过程中,CPA肿瘤位置和轮廓的确定对手术和放疗的成功与否都影响极大.由于不同器官对放疗处方剂量的耐受度不同,因此器官分割的精准性将直接影响放疗计划设计和放疗疗效[4 ] . ...
MRI对颅内脑膜瘤的诊断价值研究
1
2016
... 对病灶的精准分割是医学图像研究领域的关键之一,本文主要研究桥小脑角区(cerebellopontine angle,CPA)的肿瘤分割.CPA是中枢神经系统肿瘤的好发部位之一,约占颅内占位性病变的6%~10%[1 ] .CPA肿瘤包括听神经瘤、脑膜瘤、胆脂瘤、神经鞘瘤、血管性病变瘤等等,其中听神经瘤和脑膜瘤是最为常见的两种CPA肿瘤.听神经瘤起源于听神经前庭,约占CPA肿瘤的85%,占颅内肿瘤的6%[2 ] .脑膜瘤为颅内常见的肿瘤之一,约占颅内原发性肿瘤的15%~20%[3 ] .目前治疗CPA肿瘤的方法主要包括手术疗法、放射治疗、化疗.而在手术切除和放疗过程中,CPA肿瘤位置和轮廓的确定对手术和放疗的成功与否都影响极大.由于不同器官对放疗处方剂量的耐受度不同,因此器官分割的精准性将直接影响放疗计划设计和放疗疗效[4 ] . ...
MRI对颅内脑膜瘤的诊断价值研究
1
2016
... 对病灶的精准分割是医学图像研究领域的关键之一,本文主要研究桥小脑角区(cerebellopontine angle,CPA)的肿瘤分割.CPA是中枢神经系统肿瘤的好发部位之一,约占颅内占位性病变的6%~10%[1 ] .CPA肿瘤包括听神经瘤、脑膜瘤、胆脂瘤、神经鞘瘤、血管性病变瘤等等,其中听神经瘤和脑膜瘤是最为常见的两种CPA肿瘤.听神经瘤起源于听神经前庭,约占CPA肿瘤的85%,占颅内肿瘤的6%[2 ] .脑膜瘤为颅内常见的肿瘤之一,约占颅内原发性肿瘤的15%~20%[3 ] .目前治疗CPA肿瘤的方法主要包括手术疗法、放射治疗、化疗.而在手术切除和放疗过程中,CPA肿瘤位置和轮廓的确定对手术和放疗的成功与否都影响极大.由于不同器官对放疗处方剂量的耐受度不同,因此器官分割的精准性将直接影响放疗计划设计和放疗疗效[4 ] . ...
机器学习在肿瘤放射治疗领域应用进展
1
2019
... 对病灶的精准分割是医学图像研究领域的关键之一,本文主要研究桥小脑角区(cerebellopontine angle,CPA)的肿瘤分割.CPA是中枢神经系统肿瘤的好发部位之一,约占颅内占位性病变的6%~10%[1 ] .CPA肿瘤包括听神经瘤、脑膜瘤、胆脂瘤、神经鞘瘤、血管性病变瘤等等,其中听神经瘤和脑膜瘤是最为常见的两种CPA肿瘤.听神经瘤起源于听神经前庭,约占CPA肿瘤的85%,占颅内肿瘤的6%[2 ] .脑膜瘤为颅内常见的肿瘤之一,约占颅内原发性肿瘤的15%~20%[3 ] .目前治疗CPA肿瘤的方法主要包括手术疗法、放射治疗、化疗.而在手术切除和放疗过程中,CPA肿瘤位置和轮廓的确定对手术和放疗的成功与否都影响极大.由于不同器官对放疗处方剂量的耐受度不同,因此器官分割的精准性将直接影响放疗计划设计和放疗疗效[4 ] . ...
机器学习在肿瘤放射治疗领域应用进展
1
2019
... 对病灶的精准分割是医学图像研究领域的关键之一,本文主要研究桥小脑角区(cerebellopontine angle,CPA)的肿瘤分割.CPA是中枢神经系统肿瘤的好发部位之一,约占颅内占位性病变的6%~10%[1 ] .CPA肿瘤包括听神经瘤、脑膜瘤、胆脂瘤、神经鞘瘤、血管性病变瘤等等,其中听神经瘤和脑膜瘤是最为常见的两种CPA肿瘤.听神经瘤起源于听神经前庭,约占CPA肿瘤的85%,占颅内肿瘤的6%[2 ] .脑膜瘤为颅内常见的肿瘤之一,约占颅内原发性肿瘤的15%~20%[3 ] .目前治疗CPA肿瘤的方法主要包括手术疗法、放射治疗、化疗.而在手术切除和放疗过程中,CPA肿瘤位置和轮廓的确定对手术和放疗的成功与否都影响极大.由于不同器官对放疗处方剂量的耐受度不同,因此器官分割的精准性将直接影响放疗计划设计和放疗疗效[4 ] . ...
MRI脑肿瘤图像分割研究进展及挑战
1
2020
... 磁共振成像(magnetic resonance imaging,MRI)作为一种非倾入性的成像方法,具有良好的软组织对比度,且无放射线损害,适合用于临床脑部病变检查[5 ] .已有多种方法被应用于磁共振图像分割.王长福等[6 ] 提出了一种结合多模态MRI和电子计算机断层扫描(computed tomography,CT)-MRI图像融合的技术,用来对颅脑恶性肿瘤进行勾画,而这种方法主要还是依赖于医生的勾画,而且对图像分割来说过于单一.Zikic等[7 ] 于2014年提出将卷积神经网络(convolutional neural network,CNN)应用到基于磁共振图像的脑肿瘤分割,他们采用了Jia等[8 ] 提出的CNN网络,并将其改为五层网络结构,在BraTS 2013挑战的数据集上取得了一定效果,分割准确率达到83.7%±9.4%,说明将CNN应用于磁共振图像分割是可行的.Iqbal等[9 ] 于2018年提出了一种基于CNN的多尺度脑肿瘤磁共振图像分割方法,他们使用了包括跳跃路由网络(SkipNet)、插值卷积网络(IntNet)、压缩激励网络(SENet)三种不同的网络结构,也取得了一定的效果,证明神经网络可应用于磁共振图像分割.Ezhilarasi等[10 ] 于2018年提出了基于更快速区域卷积神经网络(faster region convolutional neural network,Faster-RCNN)的脑肿瘤检测方法,但是他们只实现了脑肿瘤的定位与良恶性判别,有一定的局限性.杨新焕等[11 ] 于2020年通过结合MRI多模态信息和二维CNN对脑肿瘤的分割准确率为83.67%,结合MRI多模态信息和三维CNN对脑肿瘤分割的准确率为88.26%.Thillaikkarasi等[12 ] 于2019年提出了一种利用支持向量机提取特征,再使用CNN分割的脑肿瘤分割算法,准确率达到了84%.Iqbal等[13 ] 于2019年使用长短期记忆网络(long short-term memory,LSTM)分割脑肿瘤的准确率为75%,使用CNN分割的准确率为80%,而将LSTM和CNN结合起来的分割准确率为82.29%. ...
MRI脑肿瘤图像分割研究进展及挑战
1
2020
... 磁共振成像(magnetic resonance imaging,MRI)作为一种非倾入性的成像方法,具有良好的软组织对比度,且无放射线损害,适合用于临床脑部病变检查[5 ] .已有多种方法被应用于磁共振图像分割.王长福等[6 ] 提出了一种结合多模态MRI和电子计算机断层扫描(computed tomography,CT)-MRI图像融合的技术,用来对颅脑恶性肿瘤进行勾画,而这种方法主要还是依赖于医生的勾画,而且对图像分割来说过于单一.Zikic等[7 ] 于2014年提出将卷积神经网络(convolutional neural network,CNN)应用到基于磁共振图像的脑肿瘤分割,他们采用了Jia等[8 ] 提出的CNN网络,并将其改为五层网络结构,在BraTS 2013挑战的数据集上取得了一定效果,分割准确率达到83.7%±9.4%,说明将CNN应用于磁共振图像分割是可行的.Iqbal等[9 ] 于2018年提出了一种基于CNN的多尺度脑肿瘤磁共振图像分割方法,他们使用了包括跳跃路由网络(SkipNet)、插值卷积网络(IntNet)、压缩激励网络(SENet)三种不同的网络结构,也取得了一定的效果,证明神经网络可应用于磁共振图像分割.Ezhilarasi等[10 ] 于2018年提出了基于更快速区域卷积神经网络(faster region convolutional neural network,Faster-RCNN)的脑肿瘤检测方法,但是他们只实现了脑肿瘤的定位与良恶性判别,有一定的局限性.杨新焕等[11 ] 于2020年通过结合MRI多模态信息和二维CNN对脑肿瘤的分割准确率为83.67%,结合MRI多模态信息和三维CNN对脑肿瘤分割的准确率为88.26%.Thillaikkarasi等[12 ] 于2019年提出了一种利用支持向量机提取特征,再使用CNN分割的脑肿瘤分割算法,准确率达到了84%.Iqbal等[13 ] 于2019年使用长短期记忆网络(long short-term memory,LSTM)分割脑肿瘤的准确率为75%,使用CNN分割的准确率为80%,而将LSTM和CNN结合起来的分割准确率为82.29%. ...
多模态磁共振成像及融合技术在颅脑恶性肿瘤放疗靶区勾画中的应用
1
2018
... 磁共振成像(magnetic resonance imaging,MRI)作为一种非倾入性的成像方法,具有良好的软组织对比度,且无放射线损害,适合用于临床脑部病变检查[5 ] .已有多种方法被应用于磁共振图像分割.王长福等[6 ] 提出了一种结合多模态MRI和电子计算机断层扫描(computed tomography,CT)-MRI图像融合的技术,用来对颅脑恶性肿瘤进行勾画,而这种方法主要还是依赖于医生的勾画,而且对图像分割来说过于单一.Zikic等[7 ] 于2014年提出将卷积神经网络(convolutional neural network,CNN)应用到基于磁共振图像的脑肿瘤分割,他们采用了Jia等[8 ] 提出的CNN网络,并将其改为五层网络结构,在BraTS 2013挑战的数据集上取得了一定效果,分割准确率达到83.7%±9.4%,说明将CNN应用于磁共振图像分割是可行的.Iqbal等[9 ] 于2018年提出了一种基于CNN的多尺度脑肿瘤磁共振图像分割方法,他们使用了包括跳跃路由网络(SkipNet)、插值卷积网络(IntNet)、压缩激励网络(SENet)三种不同的网络结构,也取得了一定的效果,证明神经网络可应用于磁共振图像分割.Ezhilarasi等[10 ] 于2018年提出了基于更快速区域卷积神经网络(faster region convolutional neural network,Faster-RCNN)的脑肿瘤检测方法,但是他们只实现了脑肿瘤的定位与良恶性判别,有一定的局限性.杨新焕等[11 ] 于2020年通过结合MRI多模态信息和二维CNN对脑肿瘤的分割准确率为83.67%,结合MRI多模态信息和三维CNN对脑肿瘤分割的准确率为88.26%.Thillaikkarasi等[12 ] 于2019年提出了一种利用支持向量机提取特征,再使用CNN分割的脑肿瘤分割算法,准确率达到了84%.Iqbal等[13 ] 于2019年使用长短期记忆网络(long short-term memory,LSTM)分割脑肿瘤的准确率为75%,使用CNN分割的准确率为80%,而将LSTM和CNN结合起来的分割准确率为82.29%. ...
多模态磁共振成像及融合技术在颅脑恶性肿瘤放疗靶区勾画中的应用
1
2018
... 磁共振成像(magnetic resonance imaging,MRI)作为一种非倾入性的成像方法,具有良好的软组织对比度,且无放射线损害,适合用于临床脑部病变检查[5 ] .已有多种方法被应用于磁共振图像分割.王长福等[6 ] 提出了一种结合多模态MRI和电子计算机断层扫描(computed tomography,CT)-MRI图像融合的技术,用来对颅脑恶性肿瘤进行勾画,而这种方法主要还是依赖于医生的勾画,而且对图像分割来说过于单一.Zikic等[7 ] 于2014年提出将卷积神经网络(convolutional neural network,CNN)应用到基于磁共振图像的脑肿瘤分割,他们采用了Jia等[8 ] 提出的CNN网络,并将其改为五层网络结构,在BraTS 2013挑战的数据集上取得了一定效果,分割准确率达到83.7%±9.4%,说明将CNN应用于磁共振图像分割是可行的.Iqbal等[9 ] 于2018年提出了一种基于CNN的多尺度脑肿瘤磁共振图像分割方法,他们使用了包括跳跃路由网络(SkipNet)、插值卷积网络(IntNet)、压缩激励网络(SENet)三种不同的网络结构,也取得了一定的效果,证明神经网络可应用于磁共振图像分割.Ezhilarasi等[10 ] 于2018年提出了基于更快速区域卷积神经网络(faster region convolutional neural network,Faster-RCNN)的脑肿瘤检测方法,但是他们只实现了脑肿瘤的定位与良恶性判别,有一定的局限性.杨新焕等[11 ] 于2020年通过结合MRI多模态信息和二维CNN对脑肿瘤的分割准确率为83.67%,结合MRI多模态信息和三维CNN对脑肿瘤分割的准确率为88.26%.Thillaikkarasi等[12 ] 于2019年提出了一种利用支持向量机提取特征,再使用CNN分割的脑肿瘤分割算法,准确率达到了84%.Iqbal等[13 ] 于2019年使用长短期记忆网络(long short-term memory,LSTM)分割脑肿瘤的准确率为75%,使用CNN分割的准确率为80%,而将LSTM和CNN结合起来的分割准确率为82.29%. ...
1
... 磁共振成像(magnetic resonance imaging,MRI)作为一种非倾入性的成像方法,具有良好的软组织对比度,且无放射线损害,适合用于临床脑部病变检查[5 ] .已有多种方法被应用于磁共振图像分割.王长福等[6 ] 提出了一种结合多模态MRI和电子计算机断层扫描(computed tomography,CT)-MRI图像融合的技术,用来对颅脑恶性肿瘤进行勾画,而这种方法主要还是依赖于医生的勾画,而且对图像分割来说过于单一.Zikic等[7 ] 于2014年提出将卷积神经网络(convolutional neural network,CNN)应用到基于磁共振图像的脑肿瘤分割,他们采用了Jia等[8 ] 提出的CNN网络,并将其改为五层网络结构,在BraTS 2013挑战的数据集上取得了一定效果,分割准确率达到83.7%±9.4%,说明将CNN应用于磁共振图像分割是可行的.Iqbal等[9 ] 于2018年提出了一种基于CNN的多尺度脑肿瘤磁共振图像分割方法,他们使用了包括跳跃路由网络(SkipNet)、插值卷积网络(IntNet)、压缩激励网络(SENet)三种不同的网络结构,也取得了一定的效果,证明神经网络可应用于磁共振图像分割.Ezhilarasi等[10 ] 于2018年提出了基于更快速区域卷积神经网络(faster region convolutional neural network,Faster-RCNN)的脑肿瘤检测方法,但是他们只实现了脑肿瘤的定位与良恶性判别,有一定的局限性.杨新焕等[11 ] 于2020年通过结合MRI多模态信息和二维CNN对脑肿瘤的分割准确率为83.67%,结合MRI多模态信息和三维CNN对脑肿瘤分割的准确率为88.26%.Thillaikkarasi等[12 ] 于2019年提出了一种利用支持向量机提取特征,再使用CNN分割的脑肿瘤分割算法,准确率达到了84%.Iqbal等[13 ] 于2019年使用长短期记忆网络(long short-term memory,LSTM)分割脑肿瘤的准确率为75%,使用CNN分割的准确率为80%,而将LSTM和CNN结合起来的分割准确率为82.29%. ...
1
... 磁共振成像(magnetic resonance imaging,MRI)作为一种非倾入性的成像方法,具有良好的软组织对比度,且无放射线损害,适合用于临床脑部病变检查[5 ] .已有多种方法被应用于磁共振图像分割.王长福等[6 ] 提出了一种结合多模态MRI和电子计算机断层扫描(computed tomography,CT)-MRI图像融合的技术,用来对颅脑恶性肿瘤进行勾画,而这种方法主要还是依赖于医生的勾画,而且对图像分割来说过于单一.Zikic等[7 ] 于2014年提出将卷积神经网络(convolutional neural network,CNN)应用到基于磁共振图像的脑肿瘤分割,他们采用了Jia等[8 ] 提出的CNN网络,并将其改为五层网络结构,在BraTS 2013挑战的数据集上取得了一定效果,分割准确率达到83.7%±9.4%,说明将CNN应用于磁共振图像分割是可行的.Iqbal等[9 ] 于2018年提出了一种基于CNN的多尺度脑肿瘤磁共振图像分割方法,他们使用了包括跳跃路由网络(SkipNet)、插值卷积网络(IntNet)、压缩激励网络(SENet)三种不同的网络结构,也取得了一定的效果,证明神经网络可应用于磁共振图像分割.Ezhilarasi等[10 ] 于2018年提出了基于更快速区域卷积神经网络(faster region convolutional neural network,Faster-RCNN)的脑肿瘤检测方法,但是他们只实现了脑肿瘤的定位与良恶性判别,有一定的局限性.杨新焕等[11 ] 于2020年通过结合MRI多模态信息和二维CNN对脑肿瘤的分割准确率为83.67%,结合MRI多模态信息和三维CNN对脑肿瘤分割的准确率为88.26%.Thillaikkarasi等[12 ] 于2019年提出了一种利用支持向量机提取特征,再使用CNN分割的脑肿瘤分割算法,准确率达到了84%.Iqbal等[13 ] 于2019年使用长短期记忆网络(long short-term memory,LSTM)分割脑肿瘤的准确率为75%,使用CNN分割的准确率为80%,而将LSTM和CNN结合起来的分割准确率为82.29%. ...
Brain tumor segmentation in multi-spectral MRI using convolutional neural networks (CNN)
1
2018
... 磁共振成像(magnetic resonance imaging,MRI)作为一种非倾入性的成像方法,具有良好的软组织对比度,且无放射线损害,适合用于临床脑部病变检查[5 ] .已有多种方法被应用于磁共振图像分割.王长福等[6 ] 提出了一种结合多模态MRI和电子计算机断层扫描(computed tomography,CT)-MRI图像融合的技术,用来对颅脑恶性肿瘤进行勾画,而这种方法主要还是依赖于医生的勾画,而且对图像分割来说过于单一.Zikic等[7 ] 于2014年提出将卷积神经网络(convolutional neural network,CNN)应用到基于磁共振图像的脑肿瘤分割,他们采用了Jia等[8 ] 提出的CNN网络,并将其改为五层网络结构,在BraTS 2013挑战的数据集上取得了一定效果,分割准确率达到83.7%±9.4%,说明将CNN应用于磁共振图像分割是可行的.Iqbal等[9 ] 于2018年提出了一种基于CNN的多尺度脑肿瘤磁共振图像分割方法,他们使用了包括跳跃路由网络(SkipNet)、插值卷积网络(IntNet)、压缩激励网络(SENet)三种不同的网络结构,也取得了一定的效果,证明神经网络可应用于磁共振图像分割.Ezhilarasi等[10 ] 于2018年提出了基于更快速区域卷积神经网络(faster region convolutional neural network,Faster-RCNN)的脑肿瘤检测方法,但是他们只实现了脑肿瘤的定位与良恶性判别,有一定的局限性.杨新焕等[11 ] 于2020年通过结合MRI多模态信息和二维CNN对脑肿瘤的分割准确率为83.67%,结合MRI多模态信息和三维CNN对脑肿瘤分割的准确率为88.26%.Thillaikkarasi等[12 ] 于2019年提出了一种利用支持向量机提取特征,再使用CNN分割的脑肿瘤分割算法,准确率达到了84%.Iqbal等[13 ] 于2019年使用长短期记忆网络(long short-term memory,LSTM)分割脑肿瘤的准确率为75%,使用CNN分割的准确率为80%,而将LSTM和CNN结合起来的分割准确率为82.29%. ...
1
... 磁共振成像(magnetic resonance imaging,MRI)作为一种非倾入性的成像方法,具有良好的软组织对比度,且无放射线损害,适合用于临床脑部病变检查[5 ] .已有多种方法被应用于磁共振图像分割.王长福等[6 ] 提出了一种结合多模态MRI和电子计算机断层扫描(computed tomography,CT)-MRI图像融合的技术,用来对颅脑恶性肿瘤进行勾画,而这种方法主要还是依赖于医生的勾画,而且对图像分割来说过于单一.Zikic等[7 ] 于2014年提出将卷积神经网络(convolutional neural network,CNN)应用到基于磁共振图像的脑肿瘤分割,他们采用了Jia等[8 ] 提出的CNN网络,并将其改为五层网络结构,在BraTS 2013挑战的数据集上取得了一定效果,分割准确率达到83.7%±9.4%,说明将CNN应用于磁共振图像分割是可行的.Iqbal等[9 ] 于2018年提出了一种基于CNN的多尺度脑肿瘤磁共振图像分割方法,他们使用了包括跳跃路由网络(SkipNet)、插值卷积网络(IntNet)、压缩激励网络(SENet)三种不同的网络结构,也取得了一定的效果,证明神经网络可应用于磁共振图像分割.Ezhilarasi等[10 ] 于2018年提出了基于更快速区域卷积神经网络(faster region convolutional neural network,Faster-RCNN)的脑肿瘤检测方法,但是他们只实现了脑肿瘤的定位与良恶性判别,有一定的局限性.杨新焕等[11 ] 于2020年通过结合MRI多模态信息和二维CNN对脑肿瘤的分割准确率为83.67%,结合MRI多模态信息和三维CNN对脑肿瘤分割的准确率为88.26%.Thillaikkarasi等[12 ] 于2019年提出了一种利用支持向量机提取特征,再使用CNN分割的脑肿瘤分割算法,准确率达到了84%.Iqbal等[13 ] 于2019年使用长短期记忆网络(long short-term memory,LSTM)分割脑肿瘤的准确率为75%,使用CNN分割的准确率为80%,而将LSTM和CNN结合起来的分割准确率为82.29%. ...
结合MRI多模态信息和3D-CNNs特征提取的脑肿瘤分割研究
1
2020
... 磁共振成像(magnetic resonance imaging,MRI)作为一种非倾入性的成像方法,具有良好的软组织对比度,且无放射线损害,适合用于临床脑部病变检查[5 ] .已有多种方法被应用于磁共振图像分割.王长福等[6 ] 提出了一种结合多模态MRI和电子计算机断层扫描(computed tomography,CT)-MRI图像融合的技术,用来对颅脑恶性肿瘤进行勾画,而这种方法主要还是依赖于医生的勾画,而且对图像分割来说过于单一.Zikic等[7 ] 于2014年提出将卷积神经网络(convolutional neural network,CNN)应用到基于磁共振图像的脑肿瘤分割,他们采用了Jia等[8 ] 提出的CNN网络,并将其改为五层网络结构,在BraTS 2013挑战的数据集上取得了一定效果,分割准确率达到83.7%±9.4%,说明将CNN应用于磁共振图像分割是可行的.Iqbal等[9 ] 于2018年提出了一种基于CNN的多尺度脑肿瘤磁共振图像分割方法,他们使用了包括跳跃路由网络(SkipNet)、插值卷积网络(IntNet)、压缩激励网络(SENet)三种不同的网络结构,也取得了一定的效果,证明神经网络可应用于磁共振图像分割.Ezhilarasi等[10 ] 于2018年提出了基于更快速区域卷积神经网络(faster region convolutional neural network,Faster-RCNN)的脑肿瘤检测方法,但是他们只实现了脑肿瘤的定位与良恶性判别,有一定的局限性.杨新焕等[11 ] 于2020年通过结合MRI多模态信息和二维CNN对脑肿瘤的分割准确率为83.67%,结合MRI多模态信息和三维CNN对脑肿瘤分割的准确率为88.26%.Thillaikkarasi等[12 ] 于2019年提出了一种利用支持向量机提取特征,再使用CNN分割的脑肿瘤分割算法,准确率达到了84%.Iqbal等[13 ] 于2019年使用长短期记忆网络(long short-term memory,LSTM)分割脑肿瘤的准确率为75%,使用CNN分割的准确率为80%,而将LSTM和CNN结合起来的分割准确率为82.29%. ...
结合MRI多模态信息和3D-CNNs特征提取的脑肿瘤分割研究
1
2020
... 磁共振成像(magnetic resonance imaging,MRI)作为一种非倾入性的成像方法,具有良好的软组织对比度,且无放射线损害,适合用于临床脑部病变检查[5 ] .已有多种方法被应用于磁共振图像分割.王长福等[6 ] 提出了一种结合多模态MRI和电子计算机断层扫描(computed tomography,CT)-MRI图像融合的技术,用来对颅脑恶性肿瘤进行勾画,而这种方法主要还是依赖于医生的勾画,而且对图像分割来说过于单一.Zikic等[7 ] 于2014年提出将卷积神经网络(convolutional neural network,CNN)应用到基于磁共振图像的脑肿瘤分割,他们采用了Jia等[8 ] 提出的CNN网络,并将其改为五层网络结构,在BraTS 2013挑战的数据集上取得了一定效果,分割准确率达到83.7%±9.4%,说明将CNN应用于磁共振图像分割是可行的.Iqbal等[9 ] 于2018年提出了一种基于CNN的多尺度脑肿瘤磁共振图像分割方法,他们使用了包括跳跃路由网络(SkipNet)、插值卷积网络(IntNet)、压缩激励网络(SENet)三种不同的网络结构,也取得了一定的效果,证明神经网络可应用于磁共振图像分割.Ezhilarasi等[10 ] 于2018年提出了基于更快速区域卷积神经网络(faster region convolutional neural network,Faster-RCNN)的脑肿瘤检测方法,但是他们只实现了脑肿瘤的定位与良恶性判别,有一定的局限性.杨新焕等[11 ] 于2020年通过结合MRI多模态信息和二维CNN对脑肿瘤的分割准确率为83.67%,结合MRI多模态信息和三维CNN对脑肿瘤分割的准确率为88.26%.Thillaikkarasi等[12 ] 于2019年提出了一种利用支持向量机提取特征,再使用CNN分割的脑肿瘤分割算法,准确率达到了84%.Iqbal等[13 ] 于2019年使用长短期记忆网络(long short-term memory,LSTM)分割脑肿瘤的准确率为75%,使用CNN分割的准确率为80%,而将LSTM和CNN结合起来的分割准确率为82.29%. ...
An enhancement of deep learning algorithm for brain tumor segmentation using kernel based CNN with M-SVM
1
2019
... 磁共振成像(magnetic resonance imaging,MRI)作为一种非倾入性的成像方法,具有良好的软组织对比度,且无放射线损害,适合用于临床脑部病变检查[5 ] .已有多种方法被应用于磁共振图像分割.王长福等[6 ] 提出了一种结合多模态MRI和电子计算机断层扫描(computed tomography,CT)-MRI图像融合的技术,用来对颅脑恶性肿瘤进行勾画,而这种方法主要还是依赖于医生的勾画,而且对图像分割来说过于单一.Zikic等[7 ] 于2014年提出将卷积神经网络(convolutional neural network,CNN)应用到基于磁共振图像的脑肿瘤分割,他们采用了Jia等[8 ] 提出的CNN网络,并将其改为五层网络结构,在BraTS 2013挑战的数据集上取得了一定效果,分割准确率达到83.7%±9.4%,说明将CNN应用于磁共振图像分割是可行的.Iqbal等[9 ] 于2018年提出了一种基于CNN的多尺度脑肿瘤磁共振图像分割方法,他们使用了包括跳跃路由网络(SkipNet)、插值卷积网络(IntNet)、压缩激励网络(SENet)三种不同的网络结构,也取得了一定的效果,证明神经网络可应用于磁共振图像分割.Ezhilarasi等[10 ] 于2018年提出了基于更快速区域卷积神经网络(faster region convolutional neural network,Faster-RCNN)的脑肿瘤检测方法,但是他们只实现了脑肿瘤的定位与良恶性判别,有一定的局限性.杨新焕等[11 ] 于2020年通过结合MRI多模态信息和二维CNN对脑肿瘤的分割准确率为83.67%,结合MRI多模态信息和三维CNN对脑肿瘤分割的准确率为88.26%.Thillaikkarasi等[12 ] 于2019年提出了一种利用支持向量机提取特征,再使用CNN分割的脑肿瘤分割算法,准确率达到了84%.Iqbal等[13 ] 于2019年使用长短期记忆网络(long short-term memory,LSTM)分割脑肿瘤的准确率为75%,使用CNN分割的准确率为80%,而将LSTM和CNN结合起来的分割准确率为82.29%. ...
Deep learning model integrating features and novel classifiers fusion for brain tumor segmentation
1
2019
... 磁共振成像(magnetic resonance imaging,MRI)作为一种非倾入性的成像方法,具有良好的软组织对比度,且无放射线损害,适合用于临床脑部病变检查[5 ] .已有多种方法被应用于磁共振图像分割.王长福等[6 ] 提出了一种结合多模态MRI和电子计算机断层扫描(computed tomography,CT)-MRI图像融合的技术,用来对颅脑恶性肿瘤进行勾画,而这种方法主要还是依赖于医生的勾画,而且对图像分割来说过于单一.Zikic等[7 ] 于2014年提出将卷积神经网络(convolutional neural network,CNN)应用到基于磁共振图像的脑肿瘤分割,他们采用了Jia等[8 ] 提出的CNN网络,并将其改为五层网络结构,在BraTS 2013挑战的数据集上取得了一定效果,分割准确率达到83.7%±9.4%,说明将CNN应用于磁共振图像分割是可行的.Iqbal等[9 ] 于2018年提出了一种基于CNN的多尺度脑肿瘤磁共振图像分割方法,他们使用了包括跳跃路由网络(SkipNet)、插值卷积网络(IntNet)、压缩激励网络(SENet)三种不同的网络结构,也取得了一定的效果,证明神经网络可应用于磁共振图像分割.Ezhilarasi等[10 ] 于2018年提出了基于更快速区域卷积神经网络(faster region convolutional neural network,Faster-RCNN)的脑肿瘤检测方法,但是他们只实现了脑肿瘤的定位与良恶性判别,有一定的局限性.杨新焕等[11 ] 于2020年通过结合MRI多模态信息和二维CNN对脑肿瘤的分割准确率为83.67%,结合MRI多模态信息和三维CNN对脑肿瘤分割的准确率为88.26%.Thillaikkarasi等[12 ] 于2019年提出了一种利用支持向量机提取特征,再使用CNN分割的脑肿瘤分割算法,准确率达到了84%.Iqbal等[13 ] 于2019年使用长短期记忆网络(long short-term memory,LSTM)分割脑肿瘤的准确率为75%,使用CNN分割的准确率为80%,而将LSTM和CNN结合起来的分割准确率为82.29%. ...
Snakes: Active contour models
1
1988
... 基于偏微分方程(partial different equation,PDE)的模型也可用于图像分割.Snake模型是Kass等[14 ] 于1987年提出的一种可变的基于主动轮廓线的最小化能量泛函的模型.Chan等[15 ] 于2001年提出了基于水平集(Level-Set)的区域活动轮廓模型.可变区域拟合(region scalable fitting,RSF)是Li等[16 ] 于2008年提出的一种新的轮廓模型,该模型利用局部灰度信息来构建能量泛函,以解决图像灰度不均匀的问题,而RSF模型对图像噪声和轮廓线的初始位置比较敏感[17 ] ,因此获取磁共振图像中病灶的位置信息就显得尤为关键.元昌安等[18 ] 于2017年将RSF模型应用于医学图像分割,石沁祎等[19 ] 于2020年将水平集运用于牙齿牙槽骨图像分割,都取得了不错的效果.由此可见,将水平集方法应用在医学图像分割上也是可行的. ...
Active contours without edges
1
2001
... 基于偏微分方程(partial different equation,PDE)的模型也可用于图像分割.Snake模型是Kass等[14 ] 于1987年提出的一种可变的基于主动轮廓线的最小化能量泛函的模型.Chan等[15 ] 于2001年提出了基于水平集(Level-Set)的区域活动轮廓模型.可变区域拟合(region scalable fitting,RSF)是Li等[16 ] 于2008年提出的一种新的轮廓模型,该模型利用局部灰度信息来构建能量泛函,以解决图像灰度不均匀的问题,而RSF模型对图像噪声和轮廓线的初始位置比较敏感[17 ] ,因此获取磁共振图像中病灶的位置信息就显得尤为关键.元昌安等[18 ] 于2017年将RSF模型应用于医学图像分割,石沁祎等[19 ] 于2020年将水平集运用于牙齿牙槽骨图像分割,都取得了不错的效果.由此可见,将水平集方法应用在医学图像分割上也是可行的. ...
Minimization of region-scalable fitting energy for image segmentation
4
2008
... 基于偏微分方程(partial different equation,PDE)的模型也可用于图像分割.Snake模型是Kass等[14 ] 于1987年提出的一种可变的基于主动轮廓线的最小化能量泛函的模型.Chan等[15 ] 于2001年提出了基于水平集(Level-Set)的区域活动轮廓模型.可变区域拟合(region scalable fitting,RSF)是Li等[16 ] 于2008年提出的一种新的轮廓模型,该模型利用局部灰度信息来构建能量泛函,以解决图像灰度不均匀的问题,而RSF模型对图像噪声和轮廓线的初始位置比较敏感[17 ] ,因此获取磁共振图像中病灶的位置信息就显得尤为关键.元昌安等[18 ] 于2017年将RSF模型应用于医学图像分割,石沁祎等[19 ] 于2020年将水平集运用于牙齿牙槽骨图像分割,都取得了不错的效果.由此可见,将水平集方法应用在医学图像分割上也是可行的. ...
... 其中,δ 为尺度参数,主要控制高斯核函数{K_\delta } [16 ] 提出的高斯核函数{K_\sigma }
... 其中,u 为自变量,当图像空间\varOmega C 正好落在轮廓线上时,能量\varepsilon _x^{Fit} \varepsilon _x^{Fit} [16 ] 指出,为使闭合轮廓线C 更为平滑,定义函数: ...
... 其中μ 为参数.使用梯度下降法求解如上方程,文献[16 ]中指出{f_{\rm{1}}} {f_{\rm{2}}}
Active contour modal based on density-oriented BIRCH clustering method for medical image segmentation
1
2020
... 基于偏微分方程(partial different equation,PDE)的模型也可用于图像分割.Snake模型是Kass等[14 ] 于1987年提出的一种可变的基于主动轮廓线的最小化能量泛函的模型.Chan等[15 ] 于2001年提出了基于水平集(Level-Set)的区域活动轮廓模型.可变区域拟合(region scalable fitting,RSF)是Li等[16 ] 于2008年提出的一种新的轮廓模型,该模型利用局部灰度信息来构建能量泛函,以解决图像灰度不均匀的问题,而RSF模型对图像噪声和轮廓线的初始位置比较敏感[17 ] ,因此获取磁共振图像中病灶的位置信息就显得尤为关键.元昌安等[18 ] 于2017年将RSF模型应用于医学图像分割,石沁祎等[19 ] 于2020年将水平集运用于牙齿牙槽骨图像分割,都取得了不错的效果.由此可见,将水平集方法应用在医学图像分割上也是可行的. ...
改进RSF主动轮廓模型的医学图像分割方法
1
2017
... 基于偏微分方程(partial different equation,PDE)的模型也可用于图像分割.Snake模型是Kass等[14 ] 于1987年提出的一种可变的基于主动轮廓线的最小化能量泛函的模型.Chan等[15 ] 于2001年提出了基于水平集(Level-Set)的区域活动轮廓模型.可变区域拟合(region scalable fitting,RSF)是Li等[16 ] 于2008年提出的一种新的轮廓模型,该模型利用局部灰度信息来构建能量泛函,以解决图像灰度不均匀的问题,而RSF模型对图像噪声和轮廓线的初始位置比较敏感[17 ] ,因此获取磁共振图像中病灶的位置信息就显得尤为关键.元昌安等[18 ] 于2017年将RSF模型应用于医学图像分割,石沁祎等[19 ] 于2020年将水平集运用于牙齿牙槽骨图像分割,都取得了不错的效果.由此可见,将水平集方法应用在医学图像分割上也是可行的. ...
改进RSF主动轮廓模型的医学图像分割方法
1
2017
... 基于偏微分方程(partial different equation,PDE)的模型也可用于图像分割.Snake模型是Kass等[14 ] 于1987年提出的一种可变的基于主动轮廓线的最小化能量泛函的模型.Chan等[15 ] 于2001年提出了基于水平集(Level-Set)的区域活动轮廓模型.可变区域拟合(region scalable fitting,RSF)是Li等[16 ] 于2008年提出的一种新的轮廓模型,该模型利用局部灰度信息来构建能量泛函,以解决图像灰度不均匀的问题,而RSF模型对图像噪声和轮廓线的初始位置比较敏感[17 ] ,因此获取磁共振图像中病灶的位置信息就显得尤为关键.元昌安等[18 ] 于2017年将RSF模型应用于医学图像分割,石沁祎等[19 ] 于2020年将水平集运用于牙齿牙槽骨图像分割,都取得了不错的效果.由此可见,将水平集方法应用在医学图像分割上也是可行的. ...
基于水平集的牙齿牙槽骨图像分割
1
2021
... 基于偏微分方程(partial different equation,PDE)的模型也可用于图像分割.Snake模型是Kass等[14 ] 于1987年提出的一种可变的基于主动轮廓线的最小化能量泛函的模型.Chan等[15 ] 于2001年提出了基于水平集(Level-Set)的区域活动轮廓模型.可变区域拟合(region scalable fitting,RSF)是Li等[16 ] 于2008年提出的一种新的轮廓模型,该模型利用局部灰度信息来构建能量泛函,以解决图像灰度不均匀的问题,而RSF模型对图像噪声和轮廓线的初始位置比较敏感[17 ] ,因此获取磁共振图像中病灶的位置信息就显得尤为关键.元昌安等[18 ] 于2017年将RSF模型应用于医学图像分割,石沁祎等[19 ] 于2020年将水平集运用于牙齿牙槽骨图像分割,都取得了不错的效果.由此可见,将水平集方法应用在医学图像分割上也是可行的. ...
基于水平集的牙齿牙槽骨图像分割
1
2021
... 基于偏微分方程(partial different equation,PDE)的模型也可用于图像分割.Snake模型是Kass等[14 ] 于1987年提出的一种可变的基于主动轮廓线的最小化能量泛函的模型.Chan等[15 ] 于2001年提出了基于水平集(Level-Set)的区域活动轮廓模型.可变区域拟合(region scalable fitting,RSF)是Li等[16 ] 于2008年提出的一种新的轮廓模型,该模型利用局部灰度信息来构建能量泛函,以解决图像灰度不均匀的问题,而RSF模型对图像噪声和轮廓线的初始位置比较敏感[17 ] ,因此获取磁共振图像中病灶的位置信息就显得尤为关键.元昌安等[18 ] 于2017年将RSF模型应用于医学图像分割,石沁祎等[19 ] 于2020年将水平集运用于牙齿牙槽骨图像分割,都取得了不错的效果.由此可见,将水平集方法应用在医学图像分割上也是可行的. ...
1
... Faster-RCNN是一种可以对目标进行检测的定位网络,但不能对图像进行实例分割.而Level-Set方法对初始轮廓较为敏感.Zhang等[20 ] 于2018年将Faster-RCNN和Level-Set二者结合起来用于眼底图像的分割.左俊皓等[21 ] 于2019年将Faster-RCNN和Level-Set结合起来运用在了遥感领域,实现了对建筑物模型的提取.本文探索了二者的结合用于CPA肿瘤图像分割的可行性,使用Faster-RCNN可以进行回归定位的特性来弥补Level-Set方法依赖初始轮廓的不足,使用Level-Set方法来弥补Faster-RCNN只能进行回归定位的不足,提出了一种结合Faster-RCNN和Level-Set对CPA肿瘤先定位再进行精确分割的方法,同时对比了CPA肿瘤勾画区域扩大前后的肿瘤分割效果,未来有望在临床上协助医生进行诊断和治疗. ...
Faster-RCNN和Level-Set结合的高分遥感影像建筑物提取
1
2019
... Faster-RCNN是一种可以对目标进行检测的定位网络,但不能对图像进行实例分割.而Level-Set方法对初始轮廓较为敏感.Zhang等[20 ] 于2018年将Faster-RCNN和Level-Set二者结合起来用于眼底图像的分割.左俊皓等[21 ] 于2019年将Faster-RCNN和Level-Set结合起来运用在了遥感领域,实现了对建筑物模型的提取.本文探索了二者的结合用于CPA肿瘤图像分割的可行性,使用Faster-RCNN可以进行回归定位的特性来弥补Level-Set方法依赖初始轮廓的不足,使用Level-Set方法来弥补Faster-RCNN只能进行回归定位的不足,提出了一种结合Faster-RCNN和Level-Set对CPA肿瘤先定位再进行精确分割的方法,同时对比了CPA肿瘤勾画区域扩大前后的肿瘤分割效果,未来有望在临床上协助医生进行诊断和治疗. ...
Faster-RCNN和Level-Set结合的高分遥感影像建筑物提取
1
2019
... Faster-RCNN是一种可以对目标进行检测的定位网络,但不能对图像进行实例分割.而Level-Set方法对初始轮廓较为敏感.Zhang等[20 ] 于2018年将Faster-RCNN和Level-Set二者结合起来用于眼底图像的分割.左俊皓等[21 ] 于2019年将Faster-RCNN和Level-Set结合起来运用在了遥感领域,实现了对建筑物模型的提取.本文探索了二者的结合用于CPA肿瘤图像分割的可行性,使用Faster-RCNN可以进行回归定位的特性来弥补Level-Set方法依赖初始轮廓的不足,使用Level-Set方法来弥补Faster-RCNN只能进行回归定位的不足,提出了一种结合Faster-RCNN和Level-Set对CPA肿瘤先定位再进行精确分割的方法,同时对比了CPA肿瘤勾画区域扩大前后的肿瘤分割效果,未来有望在临床上协助医生进行诊断和治疗. ...
基于Mask RCNN的桥小脑角区听神经瘤与脑膜瘤分类定位研究
1
2021
... 磁共振图像往往会出现灰度、对比度不均匀的情况,这会影响到基于该图像的诊断结果的准确性.针对这种图像,必须进行图像增强,调整该图像的对比度和灰度的分布,以便使病灶区域的纹理特征更为清晰.本文使用对比度受限的CLAHE算法对磁共振图像进行预处理.CLAHE算法对噪声敏感度较小,可以增强图像对比度,使局部细节更加清晰、图像更平滑,从而使CPA肿瘤病灶区域更加明显[22 ] .增强前后的对比图如图 2 所示. ...
基于Mask RCNN的桥小脑角区听神经瘤与脑膜瘤分类定位研究
1
2021
... 磁共振图像往往会出现灰度、对比度不均匀的情况,这会影响到基于该图像的诊断结果的准确性.针对这种图像,必须进行图像增强,调整该图像的对比度和灰度的分布,以便使病灶区域的纹理特征更为清晰.本文使用对比度受限的CLAHE算法对磁共振图像进行预处理.CLAHE算法对噪声敏感度较小,可以增强图像对比度,使局部细节更加清晰、图像更平滑,从而使CPA肿瘤病灶区域更加明显[22 ] .增强前后的对比图如图 2 所示. ...
1
... Faster-RCNN是Ren等[23 ] 于2015年基于Fast-RCNN提出的一个目标检测算法.本文采用VGG16作为Faster-RCNN的主干网络结构,并且前13层会对输入图像进行一系列的卷积(Cov)、ReLu激活和池化(pooling)操作,以提取特征.经过一系列的特征提取之后的提取结果我们称之为特征图(feature map).Feature map将会输入到RPN中,然后RPN会将feature map分别传送到两个通道:其中一个通道是先生成一堆锚框(anchor box),再利用Softmax层进行判断这些锚框属于前景还是后景,即经过筛选的这些框是否具有语义;另外一个通道则是用以计算包住这些锚的框的回归偏移量.然后结合这两个通道,用来获取更为精准的候选框.而这两个通道的结果会在建议(proposal)层叠加,proposal层会结合所判断出的前景框和边框回归偏移量来获取候选框,同时剔除过小和超出图像边界的候选框.这些候选框最后会和映射到感兴趣区域池化(region of interest pooling,ROIPooling)层的feature map一起做定位框的回归,最后输出网络对目标位置的预测.Faster-RCNN的网络结构如图 4 所示. ...
Fronts propagating with curvature-dependent speed: algorithms based on Hamilton-Jacobi formulations
1
1988
... Level-Set方法是由Osher和Sethian[24 ] 于1988年提出的一种依赖于时间的运动界面的水平集描述.本文定义磁共振图像空间上可以表示高维曲面函数的隐式曲线函数为水平集函数.高维水平集函数以零水平集演变至低维水平集函数,这样便可以由零水平集各点的集合来表示磁共振图像中病灶的拓扑变化. ...
基于粗糙集自适应粒度的MR脑肿瘤图像分割
3
2021
... 表 2 列举了文献[25 -29 ] 中使用的不同方法在脑肿瘤图像分割上获得的Dice 系数均值,考虑到使用的临床数据以及实验配置、参数不同,各个方法之间存在客观差异,因此本文仅以Dice 系数均值进行比较.姚传文等[25 ] 于2021年提出用粗糙集对脑肿瘤磁共振图像进行分割,效果最差,低于其他方法.文献[26 , 27 ]提出的方法Dice 系数均值分别为0.853 4和0.870 0.Xu等[29 ] 于2018年提出了结合Faster RCNN和Unet的方法取得的分割效果(0.877 5)高于Ronneberger等[28 ] 于2015年提出的Unet(0.823 4),这是由于针对实际问题的Unet变种会比Unet自身取得更好的效果.但以上方法获得的Dice 系数均值均小于本文提出的方法(扩大勾画范围1.5倍时为0.888 7),这表明本文提出的方法对CPA肿瘤图像的分割效果较为理想,有望辅助诊断放疗. ...
... [25 ]于2021年提出用粗糙集对脑肿瘤磁共振图像进行分割,效果最差,低于其他方法.文献[26 , 27 ]提出的方法Dice 系数均值分别为0.853 4和0.870 0.Xu等[29 ] 于2018年提出了结合Faster RCNN和Unet的方法取得的分割效果(0.877 5)高于Ronneberger等[28 ] 于2015年提出的Unet(0.823 4),这是由于针对实际问题的Unet变种会比Unet自身取得更好的效果.但以上方法获得的Dice 系数均值均小于本文提出的方法(扩大勾画范围1.5倍时为0.888 7),这表明本文提出的方法对CPA肿瘤图像的分割效果较为理想,有望辅助诊断放疗. ...
... Comparison of mean
Dice coefficients using different methods to segment CPA tumor
Table 2 分割方法 Dice 粗糙集自适应粒度[25 ] 0.7734 ECNN[26 ] 0.8534 多核协同[27 ] 0.8700 Unet[28 ] 0.8234 CFUN[29 ] 0.8775 本文方法 0.8887
3 结论与展望 本文将Faster-RCNN训练所得的定位模型和Level-Set方法结合起来,对CPA肿瘤磁共振图像进行了分割,并进行了肿瘤勾画区域扩大不同倍率的对比实验.结果显示,本文所提出的结合Faster-RCNN框架和Level-Set方法的分割模型对CPA肿瘤的分割效果较好,并且通过定位网络输出的精确位置信息克服了原生Level-Set方法在初始分割时对人为分割的依赖,在迭代次数较少的情况下便能得到分割结果,而且本文分割效果优于其他方法.本文提出了一种CPA肿瘤图像精准分割的新思路,可以在治疗、放疗中大大减轻医生的负担的同时,提升治疗、放疗效果,具有良好的应用价值.我们未来计划进一步扩充数据集以提升本文方法的泛化能力,同时由二维图像分割转为三维图像分割. ...
基于粗糙集自适应粒度的MR脑肿瘤图像分割
3
2021
... 表 2 列举了文献[25 -29 ] 中使用的不同方法在脑肿瘤图像分割上获得的Dice 系数均值,考虑到使用的临床数据以及实验配置、参数不同,各个方法之间存在客观差异,因此本文仅以Dice 系数均值进行比较.姚传文等[25 ] 于2021年提出用粗糙集对脑肿瘤磁共振图像进行分割,效果最差,低于其他方法.文献[26 , 27 ]提出的方法Dice 系数均值分别为0.853 4和0.870 0.Xu等[29 ] 于2018年提出了结合Faster RCNN和Unet的方法取得的分割效果(0.877 5)高于Ronneberger等[28 ] 于2015年提出的Unet(0.823 4),这是由于针对实际问题的Unet变种会比Unet自身取得更好的效果.但以上方法获得的Dice 系数均值均小于本文提出的方法(扩大勾画范围1.5倍时为0.888 7),这表明本文提出的方法对CPA肿瘤图像的分割效果较为理想,有望辅助诊断放疗. ...
... [25 ]于2021年提出用粗糙集对脑肿瘤磁共振图像进行分割,效果最差,低于其他方法.文献[26 , 27 ]提出的方法Dice 系数均值分别为0.853 4和0.870 0.Xu等[29 ] 于2018年提出了结合Faster RCNN和Unet的方法取得的分割效果(0.877 5)高于Ronneberger等[28 ] 于2015年提出的Unet(0.823 4),这是由于针对实际问题的Unet变种会比Unet自身取得更好的效果.但以上方法获得的Dice 系数均值均小于本文提出的方法(扩大勾画范围1.5倍时为0.888 7),这表明本文提出的方法对CPA肿瘤图像的分割效果较为理想,有望辅助诊断放疗. ...
... Comparison of mean
Dice coefficients using different methods to segment CPA tumor
Table 2 分割方法 Dice 粗糙集自适应粒度[25 ] 0.7734 ECNN[26 ] 0.8534 多核协同[27 ] 0.8700 Unet[28 ] 0.8234 CFUN[29 ] 0.8775 本文方法 0.8887
3 结论与展望 本文将Faster-RCNN训练所得的定位模型和Level-Set方法结合起来,对CPA肿瘤磁共振图像进行了分割,并进行了肿瘤勾画区域扩大不同倍率的对比实验.结果显示,本文所提出的结合Faster-RCNN框架和Level-Set方法的分割模型对CPA肿瘤的分割效果较好,并且通过定位网络输出的精确位置信息克服了原生Level-Set方法在初始分割时对人为分割的依赖,在迭代次数较少的情况下便能得到分割结果,而且本文分割效果优于其他方法.本文提出了一种CPA肿瘤图像精准分割的新思路,可以在治疗、放疗中大大减轻医生的负担的同时,提升治疗、放疗效果,具有良好的应用价值.我们未来计划进一步扩充数据集以提升本文方法的泛化能力,同时由二维图像分割转为三维图像分割. ...
Brain tumor segmentation using convolutional neural networks in MRI images
2
2019
... 表 2 列举了文献[25 -29 ] 中使用的不同方法在脑肿瘤图像分割上获得的Dice 系数均值,考虑到使用的临床数据以及实验配置、参数不同,各个方法之间存在客观差异,因此本文仅以Dice 系数均值进行比较.姚传文等[25 ] 于2021年提出用粗糙集对脑肿瘤磁共振图像进行分割,效果最差,低于其他方法.文献[26 , 27 ]提出的方法Dice 系数均值分别为0.853 4和0.870 0.Xu等[29 ] 于2018年提出了结合Faster RCNN和Unet的方法取得的分割效果(0.877 5)高于Ronneberger等[28 ] 于2015年提出的Unet(0.823 4),这是由于针对实际问题的Unet变种会比Unet自身取得更好的效果.但以上方法获得的Dice 系数均值均小于本文提出的方法(扩大勾画范围1.5倍时为0.888 7),这表明本文提出的方法对CPA肿瘤图像的分割效果较为理想,有望辅助诊断放疗. ...
... Comparison of mean
Dice coefficients using different methods to segment CPA tumor
Table 2 分割方法 Dice 粗糙集自适应粒度[25 ] 0.7734 ECNN[26 ] 0.8534 多核协同[27 ] 0.8700 Unet[28 ] 0.8234 CFUN[29 ] 0.8775 本文方法 0.8887
3 结论与展望 本文将Faster-RCNN训练所得的定位模型和Level-Set方法结合起来,对CPA肿瘤磁共振图像进行了分割,并进行了肿瘤勾画区域扩大不同倍率的对比实验.结果显示,本文所提出的结合Faster-RCNN框架和Level-Set方法的分割模型对CPA肿瘤的分割效果较好,并且通过定位网络输出的精确位置信息克服了原生Level-Set方法在初始分割时对人为分割的依赖,在迭代次数较少的情况下便能得到分割结果,而且本文分割效果优于其他方法.本文提出了一种CPA肿瘤图像精准分割的新思路,可以在治疗、放疗中大大减轻医生的负担的同时,提升治疗、放疗效果,具有良好的应用价值.我们未来计划进一步扩充数据集以提升本文方法的泛化能力,同时由二维图像分割转为三维图像分割. ...
基于多核协同表示分类的脑肿瘤分割算法
2
2019
... 表 2 列举了文献[25 -29 ] 中使用的不同方法在脑肿瘤图像分割上获得的Dice 系数均值,考虑到使用的临床数据以及实验配置、参数不同,各个方法之间存在客观差异,因此本文仅以Dice 系数均值进行比较.姚传文等[25 ] 于2021年提出用粗糙集对脑肿瘤磁共振图像进行分割,效果最差,低于其他方法.文献[26 , 27 ]提出的方法Dice 系数均值分别为0.853 4和0.870 0.Xu等[29 ] 于2018年提出了结合Faster RCNN和Unet的方法取得的分割效果(0.877 5)高于Ronneberger等[28 ] 于2015年提出的Unet(0.823 4),这是由于针对实际问题的Unet变种会比Unet自身取得更好的效果.但以上方法获得的Dice 系数均值均小于本文提出的方法(扩大勾画范围1.5倍时为0.888 7),这表明本文提出的方法对CPA肿瘤图像的分割效果较为理想,有望辅助诊断放疗. ...
... Comparison of mean
Dice coefficients using different methods to segment CPA tumor
Table 2 分割方法 Dice 粗糙集自适应粒度[25 ] 0.7734 ECNN[26 ] 0.8534 多核协同[27 ] 0.8700 Unet[28 ] 0.8234 CFUN[29 ] 0.8775 本文方法 0.8887
3 结论与展望 本文将Faster-RCNN训练所得的定位模型和Level-Set方法结合起来,对CPA肿瘤磁共振图像进行了分割,并进行了肿瘤勾画区域扩大不同倍率的对比实验.结果显示,本文所提出的结合Faster-RCNN框架和Level-Set方法的分割模型对CPA肿瘤的分割效果较好,并且通过定位网络输出的精确位置信息克服了原生Level-Set方法在初始分割时对人为分割的依赖,在迭代次数较少的情况下便能得到分割结果,而且本文分割效果优于其他方法.本文提出了一种CPA肿瘤图像精准分割的新思路,可以在治疗、放疗中大大减轻医生的负担的同时,提升治疗、放疗效果,具有良好的应用价值.我们未来计划进一步扩充数据集以提升本文方法的泛化能力,同时由二维图像分割转为三维图像分割. ...
基于多核协同表示分类的脑肿瘤分割算法
2
2019
... 表 2 列举了文献[25 -29 ] 中使用的不同方法在脑肿瘤图像分割上获得的Dice 系数均值,考虑到使用的临床数据以及实验配置、参数不同,各个方法之间存在客观差异,因此本文仅以Dice 系数均值进行比较.姚传文等[25 ] 于2021年提出用粗糙集对脑肿瘤磁共振图像进行分割,效果最差,低于其他方法.文献[26 , 27 ]提出的方法Dice 系数均值分别为0.853 4和0.870 0.Xu等[29 ] 于2018年提出了结合Faster RCNN和Unet的方法取得的分割效果(0.877 5)高于Ronneberger等[28 ] 于2015年提出的Unet(0.823 4),这是由于针对实际问题的Unet变种会比Unet自身取得更好的效果.但以上方法获得的Dice 系数均值均小于本文提出的方法(扩大勾画范围1.5倍时为0.888 7),这表明本文提出的方法对CPA肿瘤图像的分割效果较为理想,有望辅助诊断放疗. ...
... Comparison of mean
Dice coefficients using different methods to segment CPA tumor
Table 2 分割方法 Dice 粗糙集自适应粒度[25 ] 0.7734 ECNN[26 ] 0.8534 多核协同[27 ] 0.8700 Unet[28 ] 0.8234 CFUN[29 ] 0.8775 本文方法 0.8887
3 结论与展望 本文将Faster-RCNN训练所得的定位模型和Level-Set方法结合起来,对CPA肿瘤磁共振图像进行了分割,并进行了肿瘤勾画区域扩大不同倍率的对比实验.结果显示,本文所提出的结合Faster-RCNN框架和Level-Set方法的分割模型对CPA肿瘤的分割效果较好,并且通过定位网络输出的精确位置信息克服了原生Level-Set方法在初始分割时对人为分割的依赖,在迭代次数较少的情况下便能得到分割结果,而且本文分割效果优于其他方法.本文提出了一种CPA肿瘤图像精准分割的新思路,可以在治疗、放疗中大大减轻医生的负担的同时,提升治疗、放疗效果,具有良好的应用价值.我们未来计划进一步扩充数据集以提升本文方法的泛化能力,同时由二维图像分割转为三维图像分割. ...
2
... 表 2 列举了文献[25 -29 ] 中使用的不同方法在脑肿瘤图像分割上获得的Dice 系数均值,考虑到使用的临床数据以及实验配置、参数不同,各个方法之间存在客观差异,因此本文仅以Dice 系数均值进行比较.姚传文等[25 ] 于2021年提出用粗糙集对脑肿瘤磁共振图像进行分割,效果最差,低于其他方法.文献[26 , 27 ]提出的方法Dice 系数均值分别为0.853 4和0.870 0.Xu等[29 ] 于2018年提出了结合Faster RCNN和Unet的方法取得的分割效果(0.877 5)高于Ronneberger等[28 ] 于2015年提出的Unet(0.823 4),这是由于针对实际问题的Unet变种会比Unet自身取得更好的效果.但以上方法获得的Dice 系数均值均小于本文提出的方法(扩大勾画范围1.5倍时为0.888 7),这表明本文提出的方法对CPA肿瘤图像的分割效果较为理想,有望辅助诊断放疗. ...
... Comparison of mean
Dice coefficients using different methods to segment CPA tumor
Table 2 分割方法 Dice 粗糙集自适应粒度[25 ] 0.7734 ECNN[26 ] 0.8534 多核协同[27 ] 0.8700 Unet[28 ] 0.8234 CFUN[29 ] 0.8775 本文方法 0.8887
3 结论与展望 本文将Faster-RCNN训练所得的定位模型和Level-Set方法结合起来,对CPA肿瘤磁共振图像进行了分割,并进行了肿瘤勾画区域扩大不同倍率的对比实验.结果显示,本文所提出的结合Faster-RCNN框架和Level-Set方法的分割模型对CPA肿瘤的分割效果较好,并且通过定位网络输出的精确位置信息克服了原生Level-Set方法在初始分割时对人为分割的依赖,在迭代次数较少的情况下便能得到分割结果,而且本文分割效果优于其他方法.本文提出了一种CPA肿瘤图像精准分割的新思路,可以在治疗、放疗中大大减轻医生的负担的同时,提升治疗、放疗效果,具有良好的应用价值.我们未来计划进一步扩充数据集以提升本文方法的泛化能力,同时由二维图像分割转为三维图像分割. ...
3
... 表 2 列举了文献[25 -29 ] 中使用的不同方法在脑肿瘤图像分割上获得的Dice 系数均值,考虑到使用的临床数据以及实验配置、参数不同,各个方法之间存在客观差异,因此本文仅以Dice 系数均值进行比较.姚传文等[25 ] 于2021年提出用粗糙集对脑肿瘤磁共振图像进行分割,效果最差,低于其他方法.文献[26 , 27 ]提出的方法Dice 系数均值分别为0.853 4和0.870 0.Xu等[29 ] 于2018年提出了结合Faster RCNN和Unet的方法取得的分割效果(0.877 5)高于Ronneberger等[28 ] 于2015年提出的Unet(0.823 4),这是由于针对实际问题的Unet变种会比Unet自身取得更好的效果.但以上方法获得的Dice 系数均值均小于本文提出的方法(扩大勾画范围1.5倍时为0.888 7),这表明本文提出的方法对CPA肿瘤图像的分割效果较为理想,有望辅助诊断放疗. ...
... [29 ]于2018年提出了结合Faster RCNN和Unet的方法取得的分割效果(0.877 5)高于Ronneberger等[28 ] 于2015年提出的Unet(0.823 4),这是由于针对实际问题的Unet变种会比Unet自身取得更好的效果.但以上方法获得的Dice 系数均值均小于本文提出的方法(扩大勾画范围1.5倍时为0.888 7),这表明本文提出的方法对CPA肿瘤图像的分割效果较为理想,有望辅助诊断放疗. ...
... Comparison of mean
Dice coefficients using different methods to segment CPA tumor
Table 2 分割方法 Dice 粗糙集自适应粒度[25 ] 0.7734 ECNN[26 ] 0.8534 多核协同[27 ] 0.8700 Unet[28 ] 0.8234 CFUN[29 ] 0.8775 本文方法 0.8887
3 结论与展望 本文将Faster-RCNN训练所得的定位模型和Level-Set方法结合起来,对CPA肿瘤磁共振图像进行了分割,并进行了肿瘤勾画区域扩大不同倍率的对比实验.结果显示,本文所提出的结合Faster-RCNN框架和Level-Set方法的分割模型对CPA肿瘤的分割效果较好,并且通过定位网络输出的精确位置信息克服了原生Level-Set方法在初始分割时对人为分割的依赖,在迭代次数较少的情况下便能得到分割结果,而且本文分割效果优于其他方法.本文提出了一种CPA肿瘤图像精准分割的新思路,可以在治疗、放疗中大大减轻医生的负担的同时,提升治疗、放疗效果,具有良好的应用价值.我们未来计划进一步扩充数据集以提升本文方法的泛化能力,同时由二维图像分割转为三维图像分割. ...